Einleitung
Die Entdeckung während des Humangenomprojekts Anfang der 2000er Jahre, dass wir Menschen nur etwa 20,000 proteinkodierende Gene haben – etwa so viele wie der winzige im Boden lebende Nematodenwurm und weniger als halb so viele wie die Reispflanze – war ein Schock . Dieser Schlag gegen unseren Stolz wurde jedoch durch die Vorstellung gemildert, dass das menschliche Genom reich an regulatorischen Verbindungen ist. Unsere Gene interagieren in einem dichten Netzwerk, in dem DNA-Teile und die von ihnen kodierten Moleküle (RNA und Proteine) die „Expression“ anderer Gene steuern und beeinflussen, ob diese ihre jeweilige RNA und Proteine herstellen. Um das menschliche Genom zu verstehen, mussten wir diesen Prozess der Genregulation verstehen.
Diese Aufgabe erweist sich jedoch als viel schwieriger als die Entschlüsselung der Genomsequenz.
Zunächst wurde vermutet, dass es sich bei der Genregulation lediglich darum handelt, dass ein Genprodukt auf digitale Weise als Ein-/Ausschalter für ein anderes Gen fungiert. In den 1960er Jahren gelang es den französischen Biologen François Jacob und Jacques Monod erstmals, die Sache aufzuklären ein Genregulationsprozess im mechanistischen Detail: In Escherichia coli Wenn sich bei Bakterien ein Repressorprotein an einen bestimmten Abschnitt der DNA bindet, blockiert es die Transkription und Translation einer benachbarten Reihe von Genen, die Enzyme für die Verdauung des Zuckers Laktose kodieren. Dieser Regulierungskreislauf, den Monod und Jacob als „ See Operon verfügt über eine übersichtliche, transparente Logik.
Aber die Genregulation bei komplexen Metazoen – Tieren wie Menschen mit komplexen eukaryotischen Zellen – scheint im Allgemeinen nicht auf diese Weise zu funktionieren. Stattdessen handelt es sich um eine Gruppe von Molekülen, darunter Proteine, RNAs und DNA-Stücke aus dem gesamten Chromosom, die auf irgendeine Weise zusammenarbeiten, um die Expression eines Gens zu steuern.
Es ist nicht nur so, dass dieser Regulierungsprozess in Eukaryoten mehr Akteure hat, als es normalerweise bei Bakterien und anderen einfachen prokaryotischen Zellen der Fall ist; Es scheint ein völlig anderer und verschwommenerer Prozess zu sein.
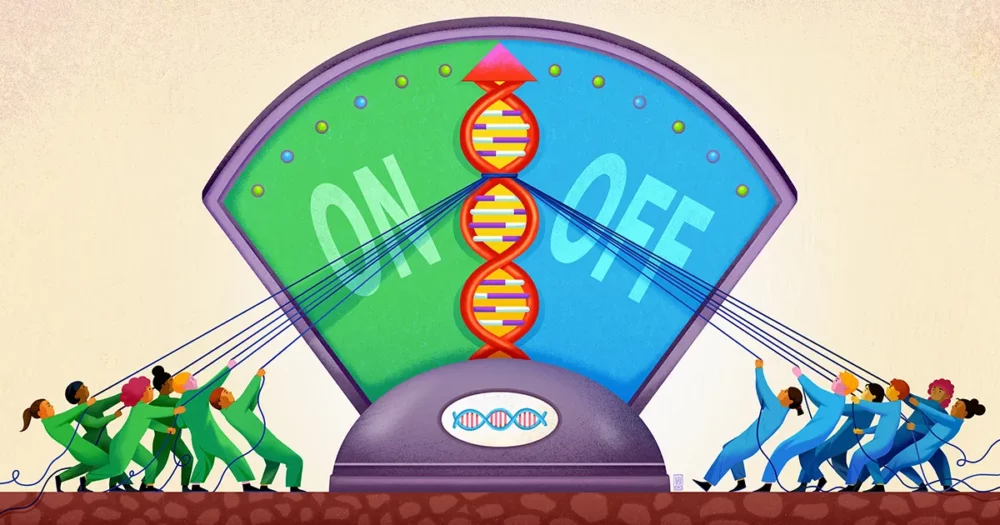
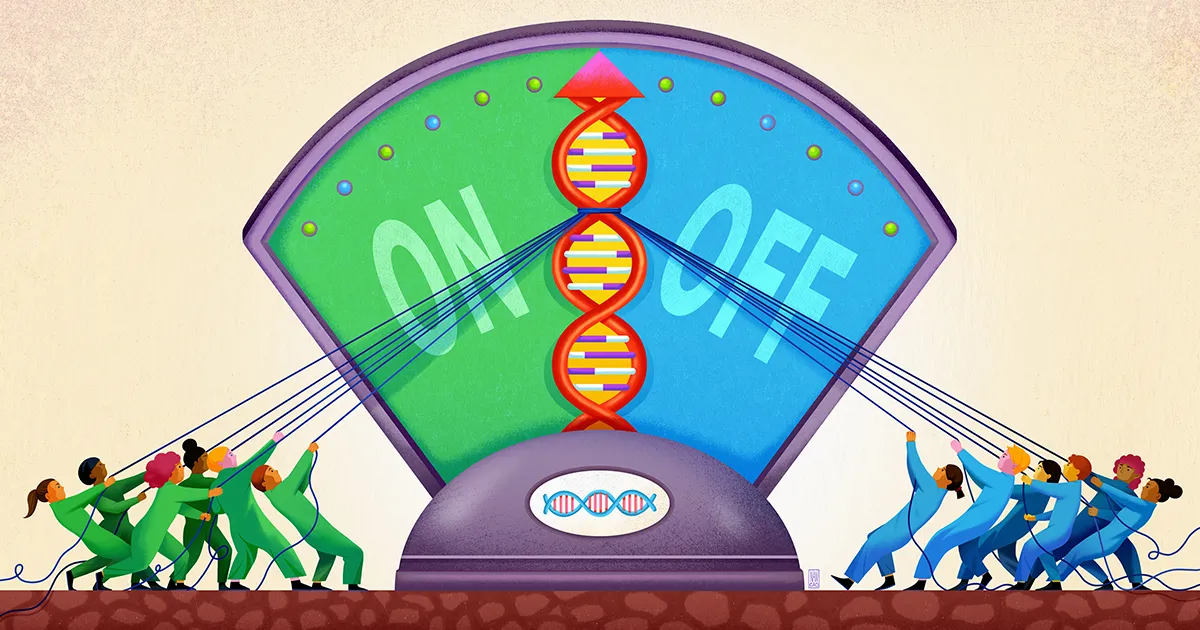
Ein Team der Stanford University unter der Leitung des Biophysikers und Bioingenieurs Polly Fordycescheint nun eine Komponente dieser unscharfen Art der Genregulation entdeckt zu haben. Ihre Arbeit, veröffentlicht letzten September in Wissenschaftlegt nahe, dass die DNA in der Nähe eines Gens als eine Art flacher Brunnen fungiert, in dem verschiedene regulatorische Moleküle gefangen werden und sie einsatzbereit bleiben, sodass sie bei Bedarf ihre Stimme bei der Entscheidung über die Aktivierung des Gens einbringen können.
Einleitung
Diese regulatorischen Vertiefungen bestehen aus ausgesprochen seltsamen DNA-Abschnitten. Sie bestehen aus Sequenzen, in denen sich ein kurzer DNA-Abschnitt mit einer Länge von einem bis sechs Basenpaaren viele Male wiederholt. Dutzende Kopien dieser „Short Tandem Repeats“ (STRs) können in diesen Sequenzen aneinandergereiht werden, wie das gleiche kleine „Wort“, das immer wieder geschrieben wird.
STRs sind im menschlichen Genom reichlich vorhanden: Sie machen etwa 5 % unserer gesamten DNA aus. Früher galten sie als klassische Beispiele für „Junk“-DNA, da ein sich wiederholender DNA-„Text“, der nur aus STRs besteht, nicht annähernd so viele aussagekräftige Informationen enthalten kann wie beispielsweise die unregelmäßige Buchstabenfolge, aus der ein Satz besteht Artikel.
Dennoch sind STRs offensichtlich nicht unbedeutend: Sie werden mit Krankheiten wie der Huntington-Krankheit, spinobulbärer Muskelatrophie, Morbus Crohn und einigen Krebsarten in Verbindung gebracht. In den letzten Jahrzehnten haben sich Hinweise darauf angehäuft, dass sie die Genregulation irgendwie verstärken oder hemmen können. Das Rätsel war, wie sie mit so wenig Informationsgehalt so mächtig sein konnten.
Komplexe Kontrollen für komplexe Zellen
Um zu verstehen, wie STRs in das Gesamtbild der Genregulation passen, gehen wir einen Schritt zurück. Gene werden routinemäßig von DNA-Stücken flankiert, die keine RNA oder Proteine kodieren, aber regulatorische Funktionen haben. Bakterielle Gene verfügen über „Promotor“-Regionen, an die Polymeraseenzyme binden können, um die Transkription benachbarter DNA in RNA zu starten. Sie verfügen außerdem routinemäßig über „Operator“-Regionen, an die Repressorproteine binden können, um die Transkription zu blockieren und so ein Gen auszuschalten, wie im Fall See Operon.
Bei Menschen und anderen Eukaryoten können die regulatorischen Sequenzen zahlreicher, vielfältiger – und verwirrender sein. Beispielsweise beeinflussen Regionen, die als Enhancer bezeichnet werden, die Wahrscheinlichkeit, dass ein Gen transkribiert wird. Enhancer sind häufig das Ziel von Proteinen, die als Transkriptionsfaktoren bezeichnet werden und durch ihre Bindung die Genexpression verstärken oder hemmen können. Seltsamerweise sind einige Enhancer Zehntausende Basenpaare von den Genen entfernt, die sie regulieren, und werden nur durch die physische Neuordnung der DNA-Schleifen in einem gepackten Chromosom in ihre Nähe gebracht.
Bei der eukaryotischen Genregulation sind typischerweise diese vielen verschiedenen regulatorischen DNA-Blöcke zusammen mit einem oder mehreren Transkriptionsfaktoren und anderen Molekülen beteiligt, die sich alle um ein Gen versammeln, wie ein Ausschuss, der einberufen wird, um zu entscheiden, was es tun soll. Sie versammeln sich in einer lockeren, dichten Gruppe.
Oft scheinen die molekularen Teilnehmer auch nicht über die in der Molekularbiologie üblichen hochselektiven Schlüssel-Schloss-Paare zu interagieren. Stattdessen sind sie viel weniger wählerisch und interagieren eher schwach und unselektiv, als würden sie umherwandern und kurze Gespräche miteinander beginnen.
Tatsächlich ist die Art und Weise, wie Transkriptionsfaktoren in Eukaryoten an die DNA binden, ein Rätsel. Lange Zeit wurde angenommen, dass ein Teil eines Transkriptionsfaktors genau wie die Teile eines Puzzles mit einer bindenden „Motiv“-Sequenz in der DNA übereinstimmen muss. Doch obwohl einige dieser Motive identifiziert wurden, korreliert ihr Vorhandensein nicht immer sehr gut damit, wo Wissenschaftler Transkriptionsfaktoren finden, die an der DNA in Zellen haften. Manchmal verbleiben Transkriptionsfaktoren in Regionen ohne Motive, während einige Motive, die so aussehen, als ob sie Transkriptionsfaktoren stark binden sollten, leer bleiben.
„Traditionell bestand das Ziel in der Genomik darin, genomische Stellen auf [binäre] Weise als entweder ‚gebunden‘ oder ‚ungebunden‘ zu klassifizieren“, anhand von Transkriptionsfaktoren, sagte Fordyce. „Aber das Bild ist viel nuancierter.“ Die einzelnen Mitglieder dieser Genregulations-„Komitees“ scheinen bei ihren Sitzungen nicht immer anwesend oder abwesend zu sein, sondern haben vielmehr unterschiedliche Wahrscheinlichkeiten, dort zu sein oder nicht.
Die Tendenz der Genregulation in Eukaryoten, auf so vielen unterschiedlichen schwachen Wechselwirkungen zwischen großen Molekülkomplexen zu beruhen, „ist einer der Dinge, die es notorisch schwierig machen, theoretisch in den Griff zu bekommen“, sagte der Biophysiker Thomas Kuhlmann von der University of California, Riverside, der schrieb ein Kommentar auf der Arbeit des Fordyce-Labors für Wissenschaft. Es ist ein tiefes Rätsel, wie aus diesem scheinbar chaotischen Prozess präzise Entscheidungen über das Ein- und Ausschalten von Genen hervorgehen.
Über die mysteriöse Fuzzy-Logik dieses Entscheidungsprozesses hinaus stellt sich auch die Frage, wie alle Ausschussmitglieder überhaupt den Weg in den richtigen Raum finden – und dann dort bleiben. Moleküle bewegen sich im Allgemeinen durch Diffusion um die Zelle herum, werden von allen anderen umgebenden Molekülen, wie zum Beispiel Wasser, hin und her getrieben und wandern in zufällige Richtungen. Wir könnten davon ausgehen, dass diese losen Gremien zu schnell auseinanderdriften, um ihre regulatorische Aufgabe zu erfüllen.
Fordyce und ihre Kollegen glauben, dass hier die STRs ins Spiel kommen. STRs kommen auffallend häufig innerhalb von Enhancer-Stellen auf der DNA vor. In ihrer Arbeit argumentieren die Forscher, dass die STRs als klebrige Flecken fungieren, die Transkriptionsfaktoren zusammenbringen und sie daran hindern, sich zu verirren.
Feinabstimmung der Klebrigkeit
Fordyces Gruppe untersuchte systematisch, wie Unterschiede in der STR-Sequenz das Anheften von Transkriptionsfaktoren an ein Bindungsmotiv beeinflussen. Sie untersuchten zwei Faktoren – einen von Hefe, einen vom Menschen –, die an einem bestimmten Sechs-Basen-Motiv festhalten. Die Forscher haben sowohl die Stärke (oder Affinität) dieser Bindung als auch die Geschwindigkeit gemessen, mit der die Transkriptionsfaktoren hängen bleiben und sich lösen (Kinetik), wenn das Motiv von einem STR statt von einer Zufallssequenz flankiert wird. Zum Vergleich untersuchten sie, wie leicht die Faktoren an den STR allein und an eine völlig zufällige DNA-Sequenz binden.
„Eine der größten Herausforderungen auf diesem Gebiet besteht darin, die unzähligen Variablen zu entwirren, die die Bindung des [Transkriptionsfaktors] an einer bestimmten Position des Genoms beeinflussen“, sagte er David Suter, Molekularbiologe an der Eidgenössischen Technischen Hochschule Lausanne in der Schweiz. Die DNA-Form, die Nähe zu anderen DNA-Segmenten und die physikalische Spannung in den DNA-Molekülen können alle eine Rolle bei der Bindung des Transkriptionsfaktors spielen. Die Werte dieser Parameter unterscheiden sich wahrscheinlich an jeder Position im Genom und möglicherweise auch zwischen Zelltypen und innerhalb einer einzelnen Zelle im Laufe der Zeit an einer bestimmten Position. „Dies ist ein riesiger Raum unbekannter Variablen, die sehr schwer zu quantifizieren sind“, sagte Suter.
Einleitung
Deshalb seien gut kontrollierte Experimente wie die des Stanford-Teams so nützlich, fügte Kuhlman hinzu. Wenn Forscher schwache Wechselwirkungen wie diese messen müssen, haben sie normalerweise zwei Möglichkeiten: Sie können ein paar sehr detaillierte, äußerst präzise Messungen durchführen und daraus verallgemeinern, oder sie können sehr viele schnelle und schmutzige Messungen durchführen und mathematisch komplexe Methoden anwenden statistische Methoden zur Ableitung von Ergebnissen. Aber Fordyce und ihre Kollegen, so Kuhlman, nutzten ein automatisiertes, mikrofluidisches Chip-basiertes Verfahren, um bei Hochdurchsatzexperimenten präzise Messungen durchzuführen, „um das Beste aus beiden Welten herauszuholen“.
Das Stanford-Team fand heraus, dass verschiedene STR-Sequenzen die Bindungsaffinitäten von Transkriptionsfaktoren an DNA um den Faktor 70 verändern können; Sie haben manchmal einen größeren Einfluss auf die Bindung des Transkriptionsfaktors als auf die Änderung der Sequenz des Bindungsmotivs selbst. Und die Auswirkungen waren für die beiden verschiedenen Transkriptionsfaktoren, die sie untersuchten, unterschiedlich.
STRs scheinen also in der Lage zu sein, die Fähigkeit von Transkriptionsfaktoren, an einer DNA-Stelle anzudocken und so ein Gen zu regulieren, fein abzustimmen. Aber wie genau?
Ein Wartezimmer in der Nähe eines Gens
Die Forscher gingen davon aus, dass der Teil eines Transkriptionsfaktors, der DNA bindet, möglicherweise schwach mit einem STR interagiert, wobei die genaue Stärke dieser Affinität von der STR-Sequenz abhängt. Da eine solche Bindung schwach ist, weist sie keine große Spezifität auf. Wenn ein Transkriptionsfaktor jedoch locker erfasst und von einem STR immer wieder freigesetzt wird, besteht die kumulative Wirkung darin, dass der Transkriptionsfaktor in der Nähe des Gens bleibt, sodass er bei Bedarf mit größerer Wahrscheinlichkeit sicher an die Motivregion bindet.
Fordyce und ihre Kollegen sagten voraus, dass STRs somit als „Lobby“ oder Brunnen fungieren, wo sich Transkriptionsfaktoren, wenn auch nur vorübergehend, in der Nähe einer regulatorischen Bindungsstelle ansammeln können. „Die repetitive Natur eines STR verstärkt die schwache Wirkung jeder einzelnen Bindungsstelle, aus der er besteht“, sagte er Connor Horton, der Erstautor der Studie, der jetzt Doktorand an der University of California, Berkeley ist.
Umgekehrt, fügte er hinzu, können einige STRs auch dazu dienen, Transkriptionsfaktoren von regulatorischen Sequenzen wegzuziehen und Transkriptionsfaktoren an anderer Stelle wie ein Schwamm aufzusaugen. Auf diese Weise können sie die Genexpression hemmen.
Die Arbeit, sagte Suter, „zeigt überzeugend, dass STRs die Bindung von Transkriptionsfaktoren in vitro direkt beeinflussen.“ Darüber hinaus zeigte das Stanford-Team mithilfe eines Algorithmus für maschinelles Lernen, dass die in ihren In-vitro-Experimenten beobachteten Effekte offenbar auch in lebenden Zellen (also in vivo) auftreten.
Jedoch müssen auch Robert Tjian, ein Biochemiker in Berkeley und Forscher am Howard Hughes Medical Institute, glaubt, dass es möglicherweise noch zu früh ist, um sicher zu sein, welchen Einfluss eine bestimmte STR-Transkriptionsfaktor-Kombination auf die Genexpression in echten Zellen hat.
Tjian, Xavier Darzacq und ihre Kollegen in dem Labor, das sie gemeinsam in Berkeley betreiben, sind sich einig, dass STRs offenbar eine Möglichkeit bieten, Transkriptionsfaktoren in der Nähe von Genregulationsstellen zu konzentrieren. Doch ohne zu wissen, wie nahe die Faktoren beieinander liegen müssen, um die Transkription zu aktivieren, ist es schwierig, die funktionelle Bedeutung dieses Ergebnisses zu verstehen. Tjian sagte, er würde gerne sehen, ob die Einführung eines STR in eine lebende Zelle die Expression eines Zielgens vorhersehbar beeinflusst. Derzeit, sagte er, sei er „nicht davon überzeugt, dass STRs notwendigerweise ein wichtiger Aspekt der [Regulations-]Mechanismen in vivo sein werden.“
Eine kombinatorische Grammatik
Ein anhaltendes Rätsel ist, wie ein solcher Mechanismus zuverlässig die Art der präzisen Genregulation ermöglicht, die Zellen benötigen, da sowohl die Stärke als auch die Selektivität der Transkriptionsfaktorbindung innerhalb der STR-Wells schwach sind. Fordyce glaubt, dass eine solche Spezifität des Einflusses viele Ursachen haben könnte – nicht nur von Unterschieden in den STR-Sequenzen, sondern auch von kooperativen Wechselwirkungen zwischen Transkriptionsfaktoren und anderen an der Regulierung beteiligten Proteinen.
Angesichts all dessen, sagte Horton, sei es nicht klar, ob es einfach sei, die Wirkung einer bestimmten STR-Transkriptionsfaktor-Kombination auf die Expression eines Gens vorherzusagen. Die Logik des Prozesses ist in der Tat unklar. Und die „Grammatik“ des Einflusses sei wahrscheinlich kombinatorischer Natur, fügte Horton hinzu: Das Ergebnis hängt von verschiedenen Kombinationen von Transkriptionsfaktoren und anderen Molekülen ab.
Das Stanford-Team geht davon aus, dass vielleicht 90 % der Transkriptionsfaktoren empfindlich auf STRs reagieren, es aber im menschlichen Genom viel mehr Arten von Transkriptionsfaktoren als Arten von STRs gibt. „Die Mutation einer STR-Sequenz könnte die Bindung von 20 verschiedenen Transkriptionsfaktoren in diesem Zelltyp beeinflussen und zu einer allgemeinen Abnahme der Transkription dieses benachbarten Gens führen, ohne dass ein spezifischer Transkriptionsfaktor impliziert wird“, sagte Horton.
Das Stanford-Team stimmt also mit Tjian darin überein, dass die Genregulation in lebenden Zellen nicht durch einen einzigen, einfachen Mechanismus gesteuert wird. Vielmehr können sich Transkriptionsfaktoren, ihre DNA-Bindungsstellen und andere regulatorische Moleküle zu dichten Ansammlungen zusammenschließen, die gemeinsam ihren Einfluss ausüben.
„Es gibt mittlerweile mehrere Beispiele, die die Idee stützen, dass DNA-Elemente Transkriptionsfaktoren so weit verdrängen können, dass sie mit Cofaktoren Kondensate bilden“, sagte er Richard Young, Zellbiologe am Whitehead Institute des Massachusetts Institute of Technology. Enhancer binden viele Transkriptionsfaktoren, um diese Überfüllung zu erzeugen. STRs können eine Zutat sein, die dazu beiträgt, dass sich Transkriptionsfaktoren in der Nähe eines Gens ansammeln, aber sie sind nicht die ganze Geschichte.
Warum Gene auf diese komplizierte Weise regulieren, anstatt sich auf die starken und spezifischen Wechselwirkungen zwischen regulatorischen Proteinen und DNA-Stellen zu verlassen, die in Prokaryoten vorherrschen? Es ist möglich, dass diese Unschärfe die Bildung großer, komplexer Metazoen überhaupt erst ermöglicht hat.
Um lebensfähige Arten zu sein, müssen Organismen in der Lage sein, sich zu entwickeln und sich an veränderte Umstände anzupassen. Wenn unsere Zellen auf ein riesiges, aber streng vorgeschriebenes Netzwerk genregulierender Interaktionen angewiesen wären, wäre es schwierig, Änderungen daran vorzunehmen, ohne den gesamten Apparat zu zerstören, so wie eine Schweizer Uhr kaputt geht, wenn wir etwas entfernen (oder auch nur geringfügig verschieben). seiner unzähligen Zahnräder. Wenn die regulatorischen molekularen Wechselwirkungen jedoch locker und eher unspezifisch sind, gibt es eine nützliche Lücke im System – so wie ein Ausschuss im Allgemeinen auch dann eine gute Entscheidung treffen kann, wenn eines seiner Mitglieder krank ist.
Fordyce weist darauf hin, dass es in Prokaryoten wie Bakterien für Transkriptionsfaktoren möglicherweise relativ einfach ist, ihre Bindungsstellen zu finden, da das zu durchsuchende Genom kleiner ist. Aber das wird schwieriger, je größer das Genom wird. In den großen Genomen von Eukaryoten „kann man das Risiko, vorübergehend an einer ‚falschen‘ Bindungsstelle stecken zu bleiben, nicht länger tolerieren“, sagte Fordyce, denn das würde die Fähigkeit beeinträchtigen, schnell auf sich ändernde Umweltbedingungen zu reagieren.
Darüber hinaus sind STRs selbst in hohem Maße entwicklungsfähig. Eine Verlängerung oder Verkürzung ihrer Sequenz oder eine Veränderung der Größe und Tiefe des „Transkriptionsfaktorbrunnens“ kann leicht durch Pannen bei der DNA-Replikation oder -Reparatur oder durch sexuelle Rekombination der Chromosomen auftreten. Für Fordyce deutet es darauf hin, dass STRs „daher als Rohmaterial für die Entwicklung neuer regulatorischer Elemente und die Feinabstimmung bestehender regulatorischer Module für sensible Transkriptionsprogramme dienen könnten“, wie etwa solche, die die Entwicklung von Tieren und Pflanzen steuern.
Die Macht schwacher Interaktionen
Solche Überlegungen veranlassen Molekularbiologen dazu, schwachen und relativ unselektiven Wechselwirkungen im Genom viel mehr Aufmerksamkeit zu schenken. Bei vielen davon handelt es sich um Proteine, die keine feste und präzise Struktur haben, sondern locker und schlaff sind – „intrinsisch ungeordnet“, wie Biochemiker es nennen. Wenn Proteine nur über starre Strukturdomänen funktionieren würden, erklärte Young, würde dies nicht nur die Entwicklungsfähigkeit von Regulierungssystemen einschränken, sondern auch die Art der dynamischen Regulierung, die im Leben zu beobachten ist. „Sie werden keinen lebenden Organismus – oder gar einen Virus – finden, der nur mit stabilen Strukturelementen wie denen einer Schweizer Uhr funktioniert“, sagte Young.
Vielleicht ist die Evolution einfach nur auf STRs als Bestandteil einer so komplexen, aber letztendlich effektiveren Lösung zur Genregulation in Eukaryoten gestoßen. STRs selbst können auf verschiedene Weise entstehen – zum Beispiel durch Fehler bei der DNA-Replikation oder durch die Aktivität von DNA-Segmenten, sogenannten transponierbaren Elementen, die im gesamten Genom Kopien von sich selbst erstellen.
„Es ist einfach so passiert, dass die daraus resultierenden schwachen Wechselwirkungen zwischen Proteinen und den repetitiven Sequenzen etwas waren, das … den Zellen, in denen es auftrat, einen selektiven Vorteil verschaffen konnte“, sagte Kuhlman. Seine Vermutung ist, dass diese Unschärfe wahrscheinlich den Eukaryoten aufgezwungen wurde, dass „sie sie später aber zu ihrem eigenen Vorteil ausnutzen konnten“. Bakterien und andere Prokaryoten können sich auf eine genau definierte „digitale“ Regulierungslogik verlassen, da ihre Zellen dazu neigen, nur in wenigen einfachen, unterschiedlichen Zuständen zu existieren, wie etwa Bewegung und Replikation.
Aber die verschiedenen Zellzustände von Metazoen seien „viel komplexer und ähneln manchmal einem Kontinuum“, sagte Suter, sodass sie durch eine unschärfere „analoge“ Regulierung besser bedient werden könnten.
„Die Genregulationssysteme in Bakterien und Eukaryoten scheinen sich erheblich voneinander unterschieden zu haben“, stimmte Tjian zu. Während Monod einmal bemerkt haben soll: „Was wahr ist für E. coli „Das trifft auf den Elefanten zu.“ Es scheint, dass das nicht immer so ist.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoData.Network Vertikale generative KI. Motiviere dich selbst. Hier zugreifen.
- PlatoAiStream. Web3-Intelligenz. Wissen verstärkt. Hier zugreifen.
- PlatoESG. Kohlenstoff, CleanTech, Energie, Umwelt, Solar, Abfallwirtschaft. Hier zugreifen.
- PlatoHealth. Informationen zu Biotechnologie und klinischen Studien. Hier zugreifen.
- Quelle: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- :hast
- :Ist
- :nicht
- :Wo
- ][P
- $UP
- 000
- 20
- 70
- a
- Fähigkeit
- Fähig
- Über uns
- abwesend
- reichlich
- Angesammelt
- Handlung
- Schauspielkunst
- Action
- aktivieren
- Aktivität
- Handlungen
- automatisch
- hinzufügen
- hinzugefügt
- benachbart
- Vorteil
- beeinflussen
- Affinität
- aufs Neue
- Übereinstimmung
- stimmt zu
- Algorithmus
- Alle
- allein
- entlang
- ebenfalls
- Obwohl
- immer
- unter
- verstärkt
- an
- und
- Tiere
- Ein anderer
- jedem
- auseinander
- SIND
- argumentieren
- entstehen
- um
- Artikel
- AS
- Aussehen
- angenommen
- At
- Aufmerksamkeit
- Autor
- Automatisiert
- ein Weg
- Zurück
- Bakterien
- Base
- BE
- weil
- werden
- war
- beginnen
- Sein
- Nutzen
- Berkeley
- BESTE
- Besser
- zwischen
- Big
- Großes Bild
- größer
- Größte
- binden
- Bindung
- Biologie
- Blockieren
- Blockiert
- blasen
- Auftrieb
- beide
- gebracht
- aber
- by
- Kalifornien
- namens
- kam
- CAN
- Zelle
- Die Zellen
- sicher
- Herausforderungen
- Änderungen
- Ändern
- Entscheidungen
- Chromosomen
- Umstände
- klassisch
- klassifizieren
- klar
- Menu
- eng
- Cluster
- zusammenarbeiten
- Kopien
- gemeinsam
- Kombination
- Kombinationen
- wie die
- Ausschuss
- gemeinsam
- Vergleich
- Komplex
- kompliziert
- Komponente
- Kompromiss
- Bedingungen
- Verbindungen
- Überlegungen
- Inhalt
- Continuum
- Smartgeräte App
- Steuerung
- Gespräche
- Genossenschaft
- könnte
- Paar
- Menschenmenge
- Jahrzehnte
- entscheidet
- Entscheidung
- Entscheidungen
- Decoding
- verringern
- Abhängig
- hängt
- Tiefe
- Detail
- detailliert
- Entwicklung
- abweichen
- Unterschiede
- anders
- schwer
- Rundfunk
- digital
- digitale Mode
- Direkt
- Entdeckung
- Krankheit
- deutlich
- verschieden
- Frau
- do
- Dock
- Tut nicht
- Domains
- Dominieren
- Nicht
- angetrieben
- synchronisiert
- im
- dynamisch
- Früh
- leicht
- Einfache
- bewirken
- Effektiv
- Effekten
- entweder
- Elemente
- Elefant
- anderswo
- entstehen
- zu steigern,
- Umwelt-
- Fehler
- Sogar
- Jedes
- Beweis
- Evolution
- entwickelt sich
- sich entwickelnden
- genau
- Beispiel
- Beispiele
- existieren
- vorhandenen
- erwarten
- Experimente
- erklärt
- Ausnutzen
- Ausdruck
- äußerst
- Tatsache
- Faktor
- Faktoren
- Fashion
- Bundes-
- wenige
- Feld
- gemustert
- Finden Sie
- Vorname
- passen
- fixiert
- Aussichten für
- gezwungen
- unten stehende Formular
- gefunden
- Französisch
- für
- funktional
- funktioniert
- Funktionen
- Bande
- sammeln
- Sammlung
- Versammlungen
- allgemein
- Genomik
- bekommen
- bekommt
- gegeben
- Kundenziele
- gehen
- gut
- regieren
- groß
- Gruppe an
- erraten
- Hälfte
- Griff
- passiert
- hart
- Schwerer
- Haben
- mit
- he
- hilft
- hier (auf dänisch)
- hoch
- seine
- Ultraschall
- aber
- HTML
- HTTPS
- riesig
- human
- Humans
- Idee
- identifiziert
- if
- Impact der HXNUMXO Observatorien
- in
- Einschließlich
- in der Tat
- Krankengymnastik
- beeinflussen
- Beeinflussung
- Information
- unbedeutend
- beantragen müssen
- Institut
- interagieren
- Interaktion
- Interaktionen
- in
- Einführung
- ausnahmslos
- beteiligen
- beteiligt
- beinhaltet
- IT
- SEINE
- selbst
- Jacob
- Puzzle
- Job
- nur
- Behalten
- Aufbewahrung
- Art
- Arten
- Wissend
- Labor
- grosse
- Nachname
- führenden
- lernen
- geführt
- weniger
- Lebensdauer
- Gefällt mir
- wahrscheinlich
- verknüpft
- wenig
- Leben
- Logik
- Lang
- länger
- sah
- Maschine
- Maschinelles Lernen
- gemacht
- Zeitschrift
- Dur
- um
- MACHT
- Weise
- viele
- Massachusetts
- Massachusetts Institute of Technology
- Spiel
- Ihres Materials
- mathematisch
- Materie
- Kann..
- vielleicht
- sinnvoll
- messen
- Messungen
- Mechanismus
- Mechanismen
- sowie medizinische
- Tagungen
- Mitglieder
- Methoden
- könnte
- MIT
- Model
- Module
- Molekular-
- Molekül
- mehr
- schlauer bewegen
- ziehen um
- viel
- mehrere
- sollen
- Myriade
- geheimnisvoll
- Mystery
- Natur
- In der Nähe von
- fast
- Notwendig
- Need
- erforderlich
- Netzwerk
- nicht
- Notizen
- jetzt an
- nuance
- und viele
- auftreten
- aufgetreten
- vorkommend
- of
- WOW!
- bieten
- vorgenommen,
- on
- einmal
- EINEM
- einzige
- or
- Andere
- UNSERE
- Ergebnis
- übrig
- Gesamt-
- besitzen
- verpackt
- Paarungen
- Paare
- Papier
- Parameter
- Teil
- Teilnehmer
- besondere
- passt
- Patches
- AUFMERKSAMKEIT
- vielleicht
- überredet
- physikalisch
- ein Bild
- Stücke
- Pflanzen
- Plato
- Datenintelligenz von Plato
- PlatoData
- Play
- Points
- Position
- möglich
- Werkzeuge
- größte treibende
- präzise
- vorhersagen
- vorhergesagt
- Präsenz
- Gegenwart
- Stolz
- wahrscheinlich
- Verfahren
- Prozessdefinierung
- produziert
- Produkt
- tiefgreifende
- Programme
- Projekt
- Proteine
- Proteine
- die
- bietet
- Beweis
- veröffentlicht
- setzen
- Rätsel
- Frage
- schnell
- ganz
- zufällig
- Bewerten
- lieber
- Roh
- leicht
- bereit
- echt
- Region
- Regionen
- Regulieren
- Rechtliches
- Regulierungsbehörden
- verhältnismäßig
- freigegeben
- verlassen
- sich auf
- bleiben
- bemerkt
- entfernen
- Knorpel zu reparieren,
- repetitiv
- Replikation
- Forscher
- diejenigen
- Reagieren
- Folge
- was zu
- Die Ergebnisse
- Reis
- Reiches
- Recht
- starr
- Risiko
- Flussufer
- RNA
- Rollen
- Zimmer
- regelmäßig
- Führen Sie
- Said
- gleich
- Wissenschaftler
- sicher
- sehen
- scheinen
- scheinbar
- scheint
- gesehen
- Segment
- Segmente
- Ergreifen
- selektiv
- empfindlich
- Satz
- September
- Reihenfolge
- brauchen
- serviert
- mehrere
- Sexuell
- seicht
- Form
- Short
- sollte
- erklären
- Bedeutung
- Einfacher
- da
- Single
- am Standort
- Seiten
- SIX
- Größe
- locker
- kleinere
- So
- Lösung
- einige
- irgendwie
- etwas
- manchmal
- Quellen
- Raumfahrt
- spezifisch
- Spezifität
- stabil
- Stanford
- Stanford Universität
- Staaten
- statistisch
- bleiben
- Schritt
- Kleben
- klebrig
- Stoppen
- Geschichte
- einfach
- Stärke
- stark
- starker
- strukturell
- Struktur
- Schüler und Studenten
- Studie
- Anschließend
- im Wesentlichen
- so
- Zucker
- Schlägt vor
- Suite
- Support
- sicher
- Umgebung
- Schweiz
- Schalter
- Schweiz
- System
- Systeme und Techniken
- Nehmen
- Tandem
- Target
- Ziele
- Aufgabe
- Team
- Technologie
- erzählt
- Neigen
- Zehn
- als
- zur Verbesserung der Gesundheitsgerechtigkeit
- Das
- ihr
- Sie
- sich
- dann
- Dort.
- deswegen
- Diese
- vom Nutzer definierten
- think
- Thinks
- fehlen uns die Worte.
- diejenigen
- obwohl?
- dachte
- Tausende
- Durch
- während
- So
- dicht
- Zeit
- mal
- zu
- gemeinsam
- auch
- Übersetzungen
- transparent
- Fang
- was immer dies auch sein sollte.
- Drehung
- XNUMX
- tippe
- Typen
- typisch
- ukr
- Letztlich
- unbedeckt
- verstehen
- Universität
- University of California
- unbekannt
- auf
- -
- benutzt
- nützlich
- gewöhnlich
- Werte
- verschiedene
- riesig
- sehr
- lebensfähig
- Virus
- Stimme
- Warten
- wurde
- Ansehen
- Wasser
- Weg..
- Wege
- we
- schwach
- webp
- GUT
- gut definiert
- Wells
- waren
- Was
- wann
- ob
- welche
- während
- WHO
- ganze
- ganz
- warum
- werden wir
- mit
- .
- ohne
- Arbeiten
- gearbeitet
- weltweit
- Wurm
- würde
- geschrieben
- schrieb
- noch
- Du
- jung
- Zephyrnet