Pharmazeutische Unternehmen, die eine Zulassung von Aufsichtsbehörden wie der US-amerikanischen Food & Drug Administration (FDA) oder der japanischen Pharmaceuticals and Medical Devices Agency (PMDA) für den Verkauf ihrer Medikamente auf dem Markt anstreben, müssen Nachweise vorlegen, um nachzuweisen, dass ihr Medikament sicher und wirksam für seinen Zweck ist verwenden. Ein Team aus Ärzten, Statistikern, Chemikern, Pharmakologen und anderen klinischen Wissenschaftlern überprüft die Daten zur Einreichung klinischer Studien und die vorgeschlagene Kennzeichnung. Wenn die Überprüfung ergibt, dass es genügend statistische Beweise dafür gibt, dass der gesundheitliche Nutzen des Medikaments die Risiken überwiegt, wird das Medikament zum Verkauf zugelassen.
Das Einreichungspaket für klinische Studien besteht aus tabellarischen Daten, Analysedaten, Studienmetadaten und statistischen Berichten, die aus statistischen Tabellen, Auflistungen und Abbildungen bestehen. Bei der US-amerikanischen FDA ist das Electronic Common Technical Document (eCTD) das Standardformat für die Übermittlung von Anträgen, Änderungen, Ergänzungen und Berichten an das Center for Biologics Evaluation and Research (CBER) und das Center for Drug Evaluation and Research ( CDER). Für die FDA und die japanische PMDA ist es gesetzlich vorgeschrieben, tabellierte Daten im CDISC Standard Data Tabulation Model (SDTM), Analysedaten im CDISC Analysis Dataset Model (ADaM) und Studienmetadaten in CDISC Define-XML (basierend auf dem Operational Data Model) einzureichen (ODM)).
In diesem Beitrag zeigen wir, wie wir RStudio verwenden können Amazon Sage Maker um solche behördlichen Einreichungsergebnisse zu erstellen. Dieser Beitrag beschreibt den Prozess der Einreichung klinischer Studien, wie wir Forschungsdaten klinischer Studien aufnehmen, die Daten tabellarisch darstellen und analysieren und dann statistische Berichte erstellen können – zusammenfassende Tabellen, Datenauflistungen und Zahlen (TLF). Diese Methode kann es pharmazeutischen Kunden ermöglichen, sich nahtlos mit klinischen Daten zu verbinden, die in ihrer AWS-Umgebung gespeichert sind, sie mit R zu verarbeiten und den Forschungsprozess für klinische Studien zu beschleunigen.
Entwicklungsprozess von Arzneimitteln
Der Arzneimittelentwicklungsprozess kann grob in fünf Hauptschritte unterteilt werden, wie in der folgenden Abbildung dargestellt.
Es dauert durchschnittlich 10 bis 15 Jahre und ungefähr 1 bis 3 Milliarden US-Dollar, bis ein Medikament von rund 10,000 potenziellen Molekülen erfolgreich zugelassen wird. In den frühen Phasen der Forschung (der Wirkstofffindungsphase) werden vielversprechende Wirkstoffkandidaten identifiziert, die in die präklinische Forschung übergehen. In der präklinischen Phase versuchen die Forscher, die Toxizität des Medikaments durch Vorführungen herauszufinden in vitro Experimente im Labor u in vivo Experimente an Tieren. Nach der präklinischen Erprobung gehen Medikamente in die Phase der klinischen Erprobung, in der sie am Menschen getestet werden müssen, um ihre Sicherheit und Wirksamkeit festzustellen. Die Forscher konzipieren klinische Studien und beschreiben den Studienplan im klinischen Studienprotokoll. Sie definieren die verschiedenen klinischen Forschungsphasen – von kleinen Phase-1-Studien zur Bestimmung der Arzneimittelsicherheit und -dosierung über größere Phase-2-Studien zur Bestimmung der Arzneimittelwirksamkeit und Nebenwirkungen bis hin zu noch größeren Phase-3- und -4-Studien zur Bestimmung der Arzneimittelwirksamkeit, -sicherheit und -dosis Überwachung von Nebenwirkungen. Nach erfolgreichen klinischen Studien am Menschen reicht der Arzneimittelsponsor einen New Drug Application (NDA) ein, um das Medikament zu vermarkten. Die Aufsichtsbehörden überprüfen alle Daten, arbeiten mit dem Sponsor an Informationen zur Verschreibungsetikettierung und genehmigen das Medikament. Nach der Zulassung des Medikaments überprüfen die Aufsichtsbehörden die Sicherheitsberichte nach dem Inverkehrbringen, um die Sicherheit des gesamten Produkts zu gewährleisten.
1997 wurde das Clinical Data Interchange Standards Consortium (CDISC), eine globale, gemeinnützige Organisation, bestehend aus Pharmaunternehmen, CROs, Biotech, akademischen Einrichtungen, Gesundheitsdienstleistern und Regierungsbehörden, als Freiwilligengruppe gegründet. CDISC hat Datenstandards veröffentlicht, um den Datenfluss von der Erfassung bis zur Übermittlung zu rationalisieren und den Datenaustausch zwischen Partnern und Anbietern zu erleichtern. CDISC hat die folgenden Standards veröffentlicht:
- CDASH (Harmonisierung klinischer Datenerfassungsstandards) – Standards für erhobene Daten
- SDTM (Studiendaten-Tabellenmodell) – Standards für die Übermittlung tabellarischer Daten
- ADaM (Analysedatenmodell) – Standards für Analysedaten
- SEND (Standard für den Austausch nichtklinischer Daten) – Standards für nichtklinische Daten
- PRM (Protokolldarstellungsmodell) – Standards für das Protokoll
Diese Standards können geschulten Gutachtern dabei helfen, Daten mithilfe von Standardwerkzeugen effektiver und schneller zu analysieren und dadurch die Zulassungszeiten für Arzneimittel zu verkürzen. Es ist eine gesetzliche Anforderung der US-amerikanischen FDA und der japanischen PMDA, alle tabellarischen Daten im SDTM-Format einzureichen.
R für Forschungseinreichungen für klinische Studien
SAS und R sind zwei der am häufigsten verwendeten statistischen Analysesoftware in der pharmazeutischen Industrie. Als die Entwicklung der SDTM-Standards von CDISC gestartet wurde, war SAS in der pharmazeutischen Industrie und bei der FDA nahezu universell im Einsatz. Allerdings gewinnt R heutzutage enorm an Popularität, da es Open Source ist und ständig neue Pakete und Bibliotheken hinzugefügt werden. Studenten verwenden R hauptsächlich während ihres Studiums und ihrer Forschung, und sie nehmen diese Vertrautheit mit R in ihre Arbeit mit. R bietet auch Unterstützung für neue Technologien wie fortschrittliche Deep-Learning-Integrationen.
Cloud-Anbieter wie AWS sind mittlerweile die Plattform der Wahl für Pharmakunden, um ihre Infrastruktur zu hosten. AWS bietet auch verwaltete Dienste wie SageMaker, mit denen Modelle für maschinelles Lernen (ML) mühelos erstellt, trainiert und bereitgestellt werden können. SageMaker ermöglicht auch den Zugriff auf die RStudio-IDE von überall über einen Webbrowser. Dieser Beitrag beschreibt, wie Statistikprogrammierer und Biostatistiker ihre klinischen Daten in die R-Umgebung einspeisen können, wie R-Code ausgeführt werden kann und wie Ergebnisse gespeichert werden. Wir stellen Codeausschnitte bereit, mit denen Datenwissenschaftler klinischer Studien XPT-Dateien in die R-Umgebung aufnehmen, R-Datenrahmen für SDTM und ADaM erstellen und schließlich TLF erstellen können, die in einem gespeichert werden können Amazon Simple Storage-Service (Amazon S3) Objektspeicher-Bucket.
RStudio auf SageMaker
Am 2. November 2021 hat AWS in Zusammenarbeit mit RStudio PBC angekündigt die allgemeine Verfügbarkeit von RStudio auf SageMaker, die branchenweit erste vollständig verwaltete RStudio Workbench-IDE in der Cloud. Sie können jetzt Ihre aktuelle RStudio-Lizenz mitbringen, um Ihre selbstverwalteten RStudio-Umgebungen in nur wenigen einfachen Schritten einfach zu SageMaker zu migrieren. Um mehr über diese spannende Zusammenarbeit zu erfahren, schauen Sie hier vorbei Ankündigung von RStudio auf Amazon SageMaker.
Neben der RStudio Workbench bietet die RStudio-Suite für R-Entwickler auch RStudio Connect und RStudio Package Manager. RStudio Connect wurde entwickelt, um Data Scientists die Veröffentlichung von Erkenntnissen, Dashboards und Webanwendungen zu ermöglichen. Es macht es einfach, ML- und Data-Science-Erkenntnisse aus der komplizierten Arbeit von Data Scientists zu teilen und sie Entscheidungsträgern zur Verfügung zu stellen. RStudio Connect macht auch das Hosten und Verwalten von Inhalten einfach und skalierbar für eine breite Nutzung.
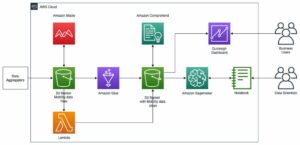
Lösungsüberblick
In den folgenden Abschnitten erörtern wir, wie wir Rohdaten aus einem Remote-Repository oder S3-Bucket in RStudio auf SageMaker importieren können. Auch eine direkte Verbindung ist möglich Relationaler Amazon-Datenbankdienst (Amazon RDS) und Data Warehouses wie Amazon RedShift (sehen Verbinden von R mit Amazon Redshift) direkt aus RStudio; Dies ist jedoch nicht Gegenstand dieses Beitrags. Nachdem Daten aus verschiedenen Quellen aufgenommen wurden, verarbeiten wir sie und erstellen R-Datenrahmen für eine Tabelle. Dann konvertieren wir den Tabellendatenrahmen in eine RTF-Datei und speichern die Ergebnisse wieder in einem S3-Bucket. Diese Ergebnisse können dann möglicherweise für Zwecke der Einreichung bei Behörden verwendet werden, vorausgesetzt, die in der Post verwendeten R-Pakete wurden für die Verwendung bei Einreichungen bei Behörden durch den Kunden validiert.
Richten Sie RStudio auf SageMaker ein
Anweisungen zum Einrichten von RStudio auf SageMaker in Ihrer Umgebung finden Sie unter Beginnen Sie mit RStudio auf SageMaker. Stellen Sie sicher, dass die Ausführungsrolle von RStudio auf SageMaker Zugriff zum Herunterladen und Hochladen von Daten in den S3-Bucket hat, in dem Daten gespeichert sind. Weitere Informationen zum Verwalten von R-Paketen und zum Veröffentlichen Ihrer Analyse mit RStudio auf SageMaker finden Sie unter Ankündigung von vollständig verwaltetem RStudio auf SageMaker für Data Scientists.
Nehmen Sie Daten in RStudio auf
In diesem Schritt nehmen wir Daten aus verschiedenen Quellen auf, um sie für unsere R-Sitzung verfügbar zu machen. Wir importieren Daten im SAS XPT-Format; Der Prozess ist jedoch ähnlich, wenn Sie Daten in anderen Formaten aufnehmen möchten. Einer der Vorteile der Verwendung von RStudio auf SageMaker besteht darin, dass SageMaker nativ auf die Daten zugreifen kann, wenn die Quelldaten in Ihren AWS-Konten gespeichert sind AWS Identity and Access Management and (IAM)-Rollen.
Greifen Sie auf Daten zu, die in einem Remote-Repository gespeichert sind
In diesem Schritt importieren wir ADaM-Daten aus der Das GitHub-Repository der FDA. Wir erstellen ein lokales Verzeichnis namens data in der RStudio-Umgebung, um die Daten zu speichern und demografische Daten herunterzuladen (dm.xpt) aus dem Remote-Repository. In diesem Zusammenhang bezieht sich das lokale Verzeichnis auf ein Verzeichnis, das auf Ihrem privaten Amazon EFS-Speicher erstellt wurde, der standardmäßig mit Ihrer R-Sitzungsumgebung verbunden ist. Siehe folgenden Code:
Wenn dieser Schritt abgeschlossen ist, können Sie sehen dm.xpt heruntergeladen werden, indem Sie zu navigieren Mappen, technische Daten, dm.xpt.
Greifen Sie auf Daten zu, die in Amazon S3 gespeichert sind
In diesem Schritt laden wir Daten herunter, die in einem S3-Bucket in unserem Konto gespeichert sind. Wir haben Inhalte aus dem GitHub-Repository der FDA in den genannten S3-Bucket kopiert aws-sagemaker-rstudio für dieses Beispiel. Siehe folgenden Code:
Wenn der Schritt abgeschlossen ist, können Sie sehen pp.xpt heruntergeladen werden, indem Sie zu navigieren Mappen, technische Daten, pp.xpt.
XPT-Daten verarbeiten
Jetzt, da wir SAS XPT-Dateien in der R-Umgebung verfügbar haben, müssen wir sie in R-Datenrahmen konvertieren und verarbeiten. Wir benutzen das haven Bibliothek zum Lesen von XPT-Dateien. Wir führen CDISC SDTM-Datensätze zusammen dm und pp um einen ADPP-Datensatz zu erstellen. Dann erstellen wir eine zusammenfassende Statistiktabelle unter Verwendung des ADPP-Datenrahmens. Die Übersichtstabelle wird dann im RTF-Format exportiert.
Zuerst werden XPT-Dateien mit dem gelesen read_xpt Funktion der Portbibliothek. Dann wird ein Analysedatensatz mit erstellt sqldf Funktion des sqldf Bibliothek. Siehe folgenden Code:
Dann wird ein Ausgangsdatenrahmen mit Funktionen aus erstellt Tplyr und dplyr Bibliotheken:
Der Ausgabedatenrahmen wird dann als RTF-Datei im Ausgabeordner in der RStudio-Umgebung gespeichert:
Hochladen von Ausgaben in Amazon S3
Nachdem die Ausgabe generiert wurde, legen wir die Daten zurück in einen S3-Bucket. Wir können dies erreichen, indem wir erneut eine SageMaker-Sitzung erstellen, wenn noch keine Sitzung aktiv ist, und den Inhalt des Ausgabeordners mithilfe von in einen S3-Bucket hochladen session$upload_data Funktion:
Mit diesen Schritten haben wir Daten aufgenommen, verarbeitet und die Ergebnisse hochgeladen, um sie den Regulierungsbehörden zur Verfügung zu stellen.
Aufräumen
Um unbeabsichtigte Kosten zu vermeiden, müssen Sie Ihre aktuelle Sitzung beenden. Wählen Sie oben rechts auf der Seite das Power-Symbol. Dadurch wird die zugrunde liegende Instanz automatisch angehalten, sodass keine unbeabsichtigten Rechenkosten anfallen.
Herausforderungen
Der Beitrag hat Schritte zum Erfassen von Rohdaten beschrieben, die in einem S3-Bucket oder aus einem Remote-Repository gespeichert sind. Es gibt jedoch viele andere Rohdatenquellen für eine klinische Studie, hauptsächlich eCRF-Daten (Electronic Case Report Forms), die in EDC-Systemen (Electronic Data Capture) wie Oracle Clinical, Medidata Rave, OpenClinica oder Snowflake gespeichert sind; Labordaten; Daten aus eCOA (klinische Ergebnisbewertung) und ePRO (elektronische Patient-Reported Outcomes); reale Daten von Apps und medizinischen Geräten; und elektronische Patientenakten (EHRs) in den Krankenhäusern. Bevor diese Daten für behördliche Einreichungen nutzbar gemacht werden können, ist eine erhebliche Vorverarbeitung erforderlich. Der Aufbau von Konnektoren zu verschiedenen Datenquellen und deren Sammlung in einem zentralen Datenspeicher (CDR) oder einem klinischen Datensee bei gleichzeitiger Aufrechterhaltung angemessener Zugriffskontrollen ist mit erheblichen Herausforderungen verbunden.
Eine weitere wichtige Herausforderung, die es zu bewältigen gilt, ist die Einhaltung gesetzlicher Vorschriften. Das Computersystem, das zum Erstellen von regulatorischen Einreichungsergebnissen verwendet wird, muss den entsprechenden Vorschriften entsprechen, wie z. B. 21 CFR Part 11, HIPAA, GDPR oder anderen GxP-Anforderungen oder ICH-Richtlinien. Dies bedeutet, dass Sie in einer validierten und qualifizierten Umgebung mit Kontrollen für Zugriff, Sicherheit, Backup und Überprüfbarkeit arbeiten. Dies bedeutet auch, dass alle R-Pakete, die zum Erstellen von regulatorischen Einreichungsergebnissen verwendet werden, vor der Verwendung validiert werden müssen.
Zusammenfassung
In diesem Beitrag haben wir gesehen, dass einige der wichtigsten Ergebnisse für eine eCTD-Einreichung CDISC SDTM, ADaM-Datensätze und TLF waren. In diesem Beitrag wurden die Schritte beschrieben, die zum Erstellen dieser behördlichen Einreichungsergebnisse erforderlich sind, indem zunächst Daten aus mehreren Quellen in RStudio auf SageMaker aufgenommen werden. Wir haben dann gesehen, wie wir die aufgenommenen Daten im XPT-Format verarbeiten können; Konvertieren Sie es in R-Datenrahmen, um SDTM, ADaM und TLF zu erstellen; und laden Sie die Ergebnisse schließlich in einen S3-Bucket hoch.
Wir hoffen, dass statistische Programmierer und Biostatistiker mit den in diesem Beitrag dargelegten allgemeinen Ideen den End-to-End-Prozess des Ladens, Verarbeitens und Analysierens von Forschungsdaten klinischer Studien in RStudio auf SageMaker leicht visualisieren und die Erkenntnisse verwenden können, um eine benutzerdefinierte zu definieren Workflow, der für Ihre behördlichen Einreichungen geeignet ist.
Können Sie sich weitere Anwendungsmöglichkeiten von RStudio vorstellen, um Forschern, Statistikern und R-Programmierern zu helfen, ihr Leben einfacher zu machen? Wir würden uns freuen, von Ihren Ideen zu hören! Und wenn Sie Fragen haben, teilen Sie diese bitte im Kommentarbereich mit.
Downloads
Weitere Informationen finden Sie unter den folgenden Links:
Über die Autoren
 Rohit Banga ist ein Global Clinical Development Industry Specialist mit Sitz in London, UK. Er ist ausgebildeter Biostatistiker und unterstützt Kunden aus den Bereichen Healthcare und LifeScience bei der Bereitstellung innovativer klinischer Entwicklungslösungen auf AWS. Er interessiert sich leidenschaftlich dafür, wie Data Science, KI/ML und neue Technologien zur Lösung realer Geschäftsprobleme in der Gesundheits- und LifeScience-Branche eingesetzt werden können. In seiner Freizeit fährt Rohit gerne Ski, grillt und verbringt Zeit mit Familie und Freunden.
Rohit Banga ist ein Global Clinical Development Industry Specialist mit Sitz in London, UK. Er ist ausgebildeter Biostatistiker und unterstützt Kunden aus den Bereichen Healthcare und LifeScience bei der Bereitstellung innovativer klinischer Entwicklungslösungen auf AWS. Er interessiert sich leidenschaftlich dafür, wie Data Science, KI/ML und neue Technologien zur Lösung realer Geschäftsprobleme in der Gesundheits- und LifeScience-Branche eingesetzt werden können. In seiner Freizeit fährt Rohit gerne Ski, grillt und verbringt Zeit mit Familie und Freunden.
 Georgios Schinas ist ein Specialist Solutions Architect für AI/ML in der EMEA-Region. Er hat seinen Sitz in London und arbeitet eng mit Kunden in Großbritannien und Irland zusammen. Georgios unterstützt Kunden bei der Entwicklung und Bereitstellung von Anwendungen für maschinelles Lernen in der Produktion auf AWS mit besonderem Interesse an MLOps-Praktiken und ermöglicht Kunden, maschinelles Lernen in großem Maßstab durchzuführen. In seiner Freizeit reist er gerne, kocht und verbringt Zeit mit Freunden und Familie.
Georgios Schinas ist ein Specialist Solutions Architect für AI/ML in der EMEA-Region. Er hat seinen Sitz in London und arbeitet eng mit Kunden in Großbritannien und Irland zusammen. Georgios unterstützt Kunden bei der Entwicklung und Bereitstellung von Anwendungen für maschinelles Lernen in der Produktion auf AWS mit besonderem Interesse an MLOps-Praktiken und ermöglicht Kunden, maschinelles Lernen in großem Maßstab durchzuführen. In seiner Freizeit reist er gerne, kocht und verbringt Zeit mit Freunden und Familie.
- AI
- Kunst
- KI-Kunstgenerator
- KI-Roboter
- Amazon Sage Maker
- Amazon einfacher Speicherdienst (S3)
- künstliche Intelligenz
- Zertifizierung für künstliche Intelligenz
- Künstliche Intelligenz im Bankwesen
- Roboter mit künstlicher Intelligenz
- Roboter mit künstlicher Intelligenz
- Software für künstliche Intelligenz
- AWS Maschinelles Lernen
- Blockchain
- Blockchain-Konferenz ai
- Einfallsreichtum
- dialogorientierte künstliche Intelligenz
- Krypto-Konferenz ai
- Dalls
- tiefe Lernen
- Google Ai
- Mittel (200)
- Biowissenschaften
- Maschinelles Lernen
- Plato
- platon ai
- Datenintelligenz von Plato
- Plato-Spiel
- PlatoData
- Platogaming
- Skala ai
- Syntax
- Zephyrnet