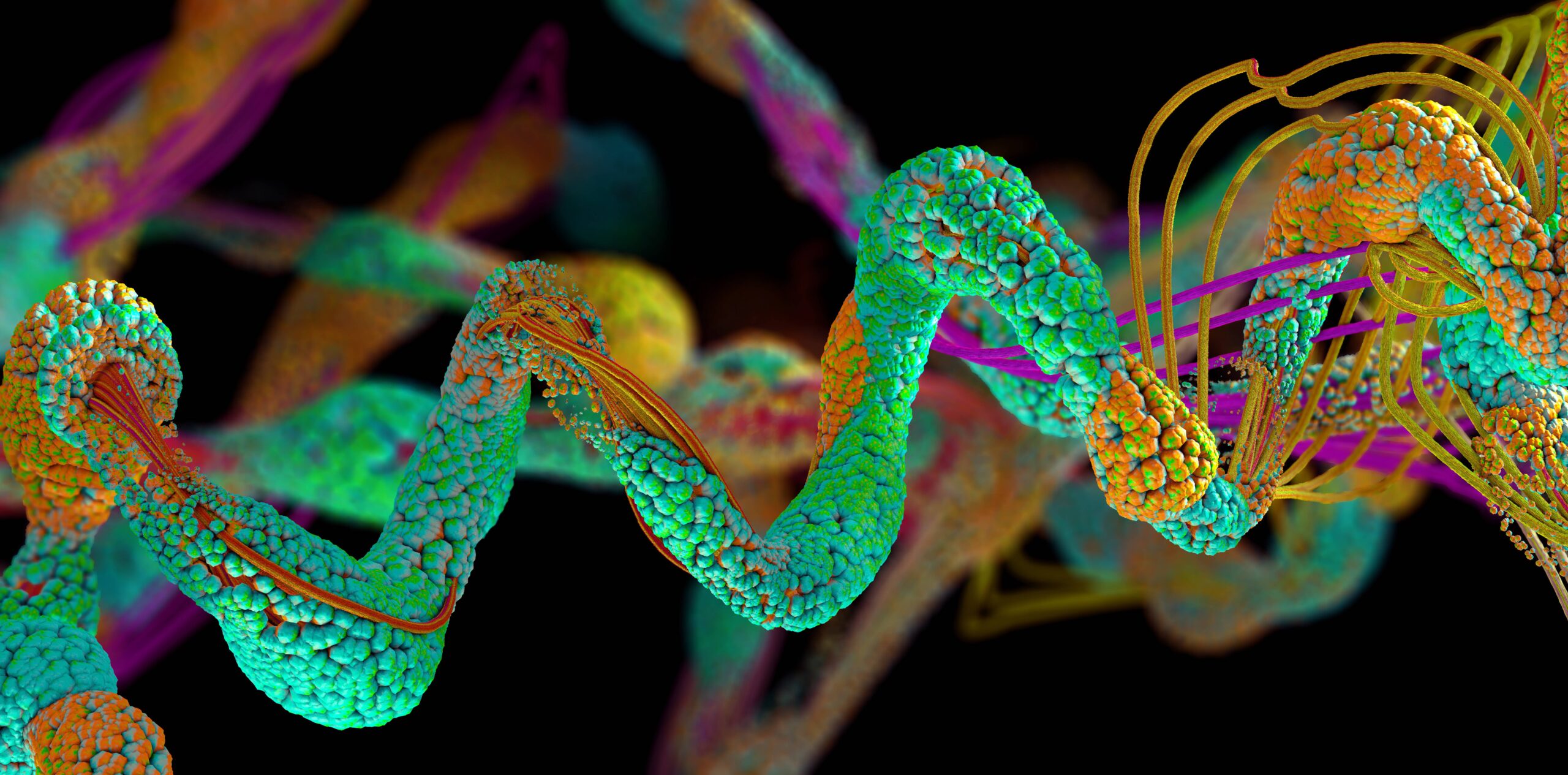
El modelo AlphaFold de plegamiento de proteínas impulsado por IA ha predicho más de 200 millones de proteínas, casi todas esas estructuras conocidas por la ciencia, dijo DeepMind el jueves.
Las proteínas son moléculas biológicas complejas producidas en organismos vivos a partir de instrucciones almacenadas en el ADN. Hechas de hasta 20 tipos de aminoácidos, estas cadenas a escala nanométrica realizan tareas celulares vitales para llevar a cabo todo tipo de funciones corporales. Conocer la forma tridimensional de las proteínas es importante, ya que su estructura física brinda pistas sobre cómo se comporta y para qué sirve, lo que nos ayuda a hacer cosas como desarrollar medicamentos y crear proteínas imitadoras para quienes carecen de ellas.
Algunas proteínas son útiles, como las involucradas en la digestión de los alimentos, mientras que otras pueden ser dañinas, como las involucradas en el crecimiento de tumores. Sin embargo, es difícil descifrar sus complicadas formas retorcidas. Los biólogos moleculares pueden pasar años realizando experimentos para descifrar la estructura de una proteína, y AlphaFold puede hacerlo en minutos, dependiendo del tamaño de la molécula, a partir de la composición de aminoácidos.
AlphaFold se entrenó en cientos de miles de estructuras de proteínas conocidas y aprendió las relaciones entre los aminoácidos constituyentes y las formas generales finales. Dada una secuencia de aminoácidos de entrada arbitraria, el modelo puede predecir una estructura de proteína 3D. Ahora, el modelo ha predicho casi todas las estructuras de proteínas conocidas por la ciencia.
En colaboración con el Instituto Europeo de Bioinformática, DeepMind ha ampliado su Base de datos de estructura de proteínas AlphaFold para contener más de 200 millones de formas 3D de proteínas de animales a plantas, de bacterias a virus: un aumento de más de 200 veces de casi un millón de moléculas a al menos 200 millones de moléculas en solo un año.
"Esperábamos que este recurso innovador ayudara a acelerar la investigación científica y el descubrimiento a nivel mundial, y que otros equipos pudieran aprender y aprovechar los avances que hicimos con AlphaFold para crear más avances", Demis Hassibis, cofundador y director ejecutivo de DeepMind, dijo en un comunicado el jueves.
“Esa esperanza se ha hecho realidad mucho más rápido de lo que nos habíamos atrevido a soñar. Solo doce meses después, más de medio millón de investigadores han accedido a AlphaFold y lo han utilizado para acelerar el progreso en importantes problemas del mundo real que van desde la contaminación plástica hasta la resistencia a los antibióticos”.
El registro ha pedido a DeepMind más comentarios.
AlphaFold también ha mostrado un gran potencial para diseñar nuevos fármacos. Las estructuras ayudan a los científicos a descubrir compuestos químicos que pueden unirse a proteínas objetivo para tratarlas o evitar que lleven a cabo funciones patológicas. Las empresas, incluida Insilco Medicine, han experimentado con el modelo para descubrir nuevas drogas; El CEO Alex Zhavoronkov dijo El registro que el proceso es mucho más complicado de lo que piensas y consta de varios pasos.
No está claro qué tan precisas son las predicciones de AlphaFold. La estructura en forma de cinta de una proteína a menudo cambia de forma cuando interactúa con un fármaco, algo en lo que AlphaFold no puede ayudar a los científicos, ya que no está entrenado en eso. Zhavoronkov dijo que el modelo es un "avance bastante notable", pero desconfiaba de toda la exageración.
“Hasta que veamos una estructura para un nuevo objetivo en una gran enfermedad obtenida a través de AlphaFold sin ningún experimento adicional, una molécula diseñada usando IA u otros métodos usando esta estructura predicha, sintetizada y probada hasta el final y luego publicada en una revista importante. – [podemos] entonces celebrar”.
Las grandes farmacéuticas quieren ver moléculas diseñadas con la ayuda de herramientas de inteligencia artificial como AlphaFold, que se prueben en ratones y humanos. “Los logros algorítmicos puros no son valiosos para las compañías farmacéuticas y especialmente para los pacientes”, agregó Zhavoronkov.
Fabio Urbina, científico principal de Collaboration Pharmaceuticals, una empresa emergente que utiliza algoritmos de aprendizaje automático para desarrollar medicamentos para enfermedades genéticas raras, dijo que AlphaFold aún no ha demostrado ser útil en su investigación. Urbina utiliza una técnica diferente y se centra más en la estructura de un nuevo fármaco potencial que en una proteína diana.
Todavía está por verse si las estructuras proteicas serán lo suficientemente útiles... para ayudarnos a descubrir nuevos fármacos potenciales para enfermedades raras.
“Esto se debe a algunas razones; las estructuras de proteínas para muchos objetivos de fármacos a menudo no estaban fácilmente disponibles para que las usaran los investigadores, y la información de proteínas no parecía ayudar a los primeros modelos de aprendizaje automático a mejorar su poder predictivo por un margen significativo”, dijo. El registro.
"Soy cautelosamente optimista de que AlphaFold esencialmente ha 'resuelto' el primer problema, pero aún está por verse si las estructuras de proteínas serán lo suficientemente útiles para nuestra aplicación posterior de mejorar el poder predictivo del aprendizaje automático para ayudarnos a descubrir nuevos fármacos potenciales". para enfermedades raras. Sin embargo, hemos visto cada vez más que la información estructural de las proteínas se tiene en cuenta como parte de los métodos de aprendizaje automático más nuevos, y hemos pensado en hacer lo mismo”.
Hacer disponible una base de datos con casi todas las estructuras de proteínas conocidas, como prometió DeepMind, significa que más científicos tendrán los recursos para experimentar y construir modelos de IA más potentes, dijo Urbina. “Soy cautelosamente optimista, pero con toda la biblioteca de estructuras de proteínas disponible, diría que hay una buena posibilidad de que las estructuras AlphaFold se incorporen a algunos de nuestros modelos de aprendizaje automático y, en última instancia, puedan ayudarnos a descubrir nuevas terapias. ” ®
- AI
- arte ai
- generador de arte ai
- robot ai
- inteligencia artificial
- certificación de inteligencia artificial
- inteligencia artificial en banca
- robots de inteligencia artificial
- robots de inteligencia artificial
- software de inteligencia artificial
- blockchain
- conferencia blockchain ai
- Coingenius
- inteligencia artificial conversacional
- criptoconferencia ai
- de dall
- deep learning
- google ai
- máquina de aprendizaje
- Platón
- platón ai
- Inteligencia de datos de Platón
- Juego de Platón
- PlatónDatos
- juego de platos
- escala ia
- sintaxis
- El registro
- zephyrnet