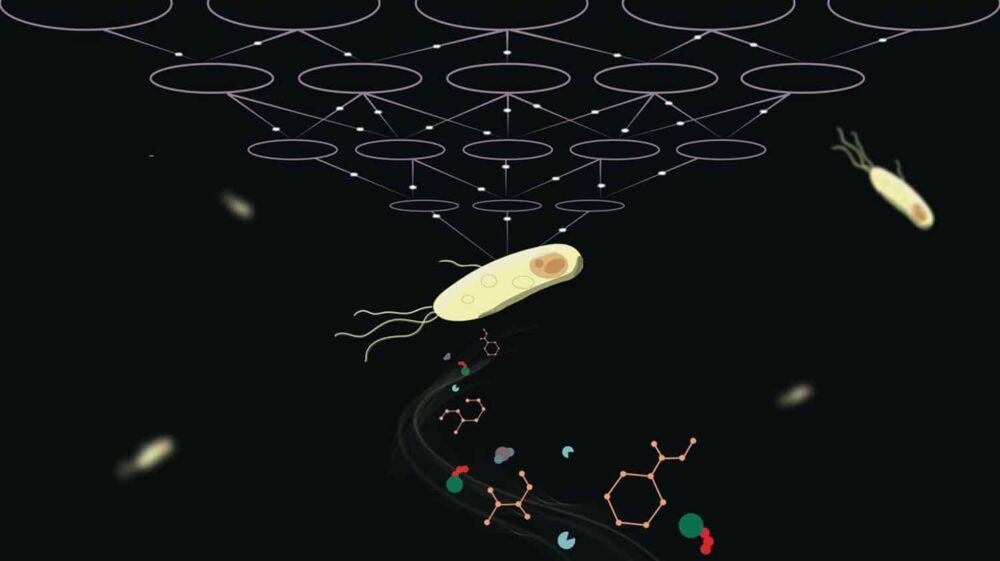
همه موجودات زنده به متابولیسم نیاز دارند. روشی که یک ارگانیسم مواد مغذی را متابولیزه می کند یک فرآیند پیچیده است و شبیه سازی فرآیندهای شیمیایی که زندگی را ادامه می دهد یک چالش دشوار است.
از نظر تئوری، این روش را می توان با معادلات ریاضی با پارامترهای خاص برای هر موجود زنده نشان داد. اما عملاً تعیین آن پارامترها به دلیل فقدان دادههای تجربی، امری پیچیده است.
عموماً دانشمندان برای یافتن این پارامترها به داده های تجربی و قدرت پردازش زیادی نیاز دارند. EPFL دانشمندان یک چارچوب محاسباتی مبتنی بر یادگیری عمیق را پیشنهاد کردند که خواص متابولیک دینامیکی مشاهده شده در سلول های. چارچوبی به نام REKINDLE میتواند راه را برای مدلسازی کارآمدتر و دقیقتر فرآیندهای متابولیک هموار کند.
Ljubisa Miskovic از آزمایشگاه بیوتکنولوژی سیستمهای محاسباتی EPFL و همکار این مطالعه گفت: REKINDLE به جامعه تحقیقاتی این امکان را می دهد که تلاش های محاسباتی در تولید مدل های جنبشی را با چندین مرتبه قدر کاهش دهند. همچنین با ادغام دادههای بیوشیمیایی در این مدلها، روشن کردن مشاهدات تجربی، و هدایت اکتشافات درمانی جدید و طرحهای بیوتکنولوژی، به فرضیههای جدید کمک میکند.»
سوبهام چودری، نویسنده اول این مطالعه، گفت: "هدف اصلی مدل سازی متابولیک، توصیف این است رفتار متابولیک سلولی تا حدی که درک و پیشبینی اثرات تغییرات در حالات سلولی و شرایط محیطی را میتوان به طور قابل اعتماد برای طیف وسیعی از مطالعات در سلامت، بیوتکنولوژی و سیستمها و زیستشناسی مصنوعی آزمایش کرد. ما امیدواریم که REKINDLE ساخت مدل های متابولیک را برای جامعه گسترده تر تسهیل کند.
این تکنیک کاربردهای بیوتکنولوژیکی مستقیمی دارد زیرا مدلهای جنبشی برای تحقیقات متعدد، از جمله مدلهای تولید زیستی، هدفگیری دارو، برهمکنشهای بین میکروبها و پاکسازی زیستی بسیار مهم هستند.
چودوری گفت:, REKINDLE از کتابخانههای استاندارد و پرکاربرد پایتون استفاده میکند که آن را در دسترس و آسان میکند. هدف اصلی ما از این مطالعه هموار کردن راه برای ایجاد این نوع تلاشهای مدلسازی منبع باز و در دسترس است تا هر کسی در جوامع زیستشناسی مصنوعی و سیستمی بتواند از آنها برای هدف تحقیقاتی خود استفاده کند، هر چه که باشد.
مرجع مجله:
- Choudhury، S.، Moret، M.، Salvy، P. و همکاران. بازسازی مدلهای جنبشی برای مطالعات دینامیکی متابولیسم با استفاده از شبکههای متخاصم مولد. نات ماخ اینتل 4، 710–719 (2022). DOI: 10.1038 / s42256-022-00519-y