La course pour résoudre toutes les structures protéiques vient d'accueillir un autre géant de la technologie : Meta AI.
Une branche de recherche de Meta, connue pour Facebook et Instagram, l'équipe est arrivée sur la scène de la prédiction de la forme des protéines avec un objectif ambitieux : déchiffrer la « matière noire » de l'univers des protéines. Souvent trouvées dans les bactéries, les virus et autres micro-organismes, ces protéines vivent dans notre environnement quotidien mais sont des mystères complets pour la science.
«Ce sont les structures que nous connaissons le moins. Ce sont des protéines incroyablement mystérieuses. Je pense qu'ils offrent le potentiel d'un grand aperçu de la biologie », a affirmé Valérie Plante. auteur principal, le Dr Alexander Rives à Nature.
En d'autres termes, ils sont un trésor d'inspiration pour la biotechnologie. Cachées dans leurs formes secrètes se trouvent des clés pour la conception biocarburants efficaces, antibiotiques, enzymes, ou même des organismes entièrement nouveaux. À leur tour, les données des prédictions de protéines pourraient entraîner davantage de modèles d'IA.
Au cœur de la nouvelle IA de Meta, baptisée ESMFold, se trouve un grand modèle de langage. Cela peut sembler familier. Ces algorithmes d'apprentissage automatique ont pris d'assaut le monde avec le chatbot vedette ChatGPT. Connu pour sa capacité à générer de beaux essais, poèmes et paroles avec des invites simples, ChatGPT et le récemment lancé GPT-4— sont formés avec des millions de textes accessibles au public. Finalement, l'IA apprend à prédire des lettres, des mots et même à écrire des paragraphes entiers et, dans le cas du chatbot similaire de Bing, à tenir conversations qui deviennent parfois légèrement énervants.
La nouvelle étude, publié dans Sciences, relie le modèle d'IA à la biologie. Les protéines sont composées de 20 « lettres ». Grâce à l'évolution, l'enchaînement des lettres permet de générer leurs formes ultimes. Si les grands modèles de langage peuvent facilement interpréter les 26 lettres de l'alphabet anglais en messages cohérents, pourquoi ne peuvent-ils pas également fonctionner pour les protéines ?
Spoiler : ils le font. ESM-2 a analysé environ 600 millions de prédictions de structure de protéines en seulement deux semaines à l'aide de 2,000 60 unités de traitement graphique (GPU). Par rapport aux tentatives précédentes, l'IA a rendu le processus jusqu'à XNUMX fois plus rapide. Les auteurs ont mis chaque structure dans l'Atlas métagénomique ESM, que vous pouvez explorer ici.
Pour le Dr Alfonso Valencia du Centre national de supercalcul de Barcelone (BCS), qui n'a pas participé aux travaux, la beauté de l'utilisation de grands systèmes linguistiques est un "simplicité conceptuelle.” Avec un développement plus poussé, l'IA peut prédire "la structure des protéines non naturelles, élargissant l'univers connu au-delà de ce que les processus évolutifs ont exploré".
Parlons évolution
ESMFold suit une directive simple : la séquence prédit la structure.
Revenons en arrière. Les protéines sont constituées de 20 acides aminés, chacun étant une « lettre », et enfilées comme des perles pointues sur une ficelle. Nos cellules les façonnent ensuite en traits délicats : certains ressemblent à des draps froissés, d'autres à une canne en bonbon tourbillonnante ou à des rubans lâches. Les protéines peuvent alors s'agripper les unes aux autres pour former un multiplex, par exemple, un tunnel qui traverse la membrane cellulaire du cerveau qui contrôle ses actions et contrôle à son tour notre façon de penser et de nous souvenir.
Les scientifiques savent depuis longtemps que les lettres d'acides aminés aident à façonner la structure finale d'une protéine. Semblables aux lettres ou aux caractères d'une langue, seuls certains d'entre eux, lorsqu'ils sont liés, ont un sens. Dans le cas des protéines, ces séquences les rendent fonctionnelles.
"Les propriétés biologiques d'une protéine contraignent les mutations à sa séquence qui sont sélectionnées au cours de l'évolution", ont déclaré les auteurs.
Semblable à la façon dont différentes lettres de l'alphabet convergent pour créer des mots, des phrases et des paragraphes sans ressembler à un charabia complet, les lettres protéinées font de même. Il existe une sorte de « dictionnaire évolutif » qui aide à enchaîner les acides aminés dans des structures que le corps peut comprendre.
"La logique de la succession des acides aminés dans les protéines connues est le résultat d'un processus évolutif qui les a amenés à avoir la structure spécifique avec laquelle ils remplissent une fonction particulière", a déclaré Valencia.
M. AI, fais de moi une protéine
Le dictionnaire relativement limité de la vie est bonne nouvelle pour les grands modèles de langage.
Ces modèles d'IA parcourent les textes facilement disponibles pour apprendre et construire des prédictions du mot suivant. Le résultat final, comme on le voit dans GPT-3 et ChatGPT, sont des conversations étonnamment naturelles et des images artistiques fantastiques.
Meta AI a utilisé le même concept, mais a réécrit le playbook pour les prédictions de la structure des protéines. Plutôt que d'alimenter l'algorithme avec des textes, ils ont donné au programme des séquences de protéines connues.
Le modèle d'IA, appelé modèle de langage des protéines transformatrices, a appris l'architecture générale des protéines en utilisant jusqu'à 15 milliards de "paramètres". Il a vu environ 65 millions de séquences de protéines différentes au total.
Dans leur prochaine étape, l'équipe a caché certaines lettres à l'IA, l'incitant à remplir les blancs. Dans ce qui équivaut à la saisie semi-automatique, le programme a finalement appris comment différents acides aminés se connectent (ou se repoussent). En fin de compte, l'IA a formé une compréhension intuitive des séquences de protéines évolutives et de la façon dont elles fonctionnent ensemble pour fabriquer des protéines fonctionnelles.
Dans l'inconnu
Comme preuve de concept, l'équipe a testé ESMFold à l'aide de deux ensembles de tests bien connus. L'un, CAMEO, concernait près de 200 structures ; l'autre, CASP14, a 51 formes de protéines publiées publiquement.
Dans l'ensemble, l'IA "fournit une précision de prédiction de structure de pointe", a déclaré l'équipe, "correspondant aux performances d'AlphaFold2 sur plus de la moitié des protéines". Il s'est également attaqué de manière fiable à de grands complexes protéiques, par exemple les canaux des neurones qui contrôlent leurs actions.
L'équipe a ensuite poussé son IA un peu plus loin, s'aventurant dans le monde de la métagénomique.
Les métagénomes sont ce à quoi ils ressemblent : un méli-mélo de matériel ADN. Normalement, ceux-ci proviennent de sources environnementales telles que la saleté sous vos pieds, l'eau de mer ou même des évents thermiques normalement inhospitaliers. La plupart des microbes ne peuvent pas être cultivés artificiellement dans les laboratoires, mais certains ont des super pouvoirs tels que la résistance à la chaleur volcanique, ce qui en fait une matière noire biologique encore à explorer.
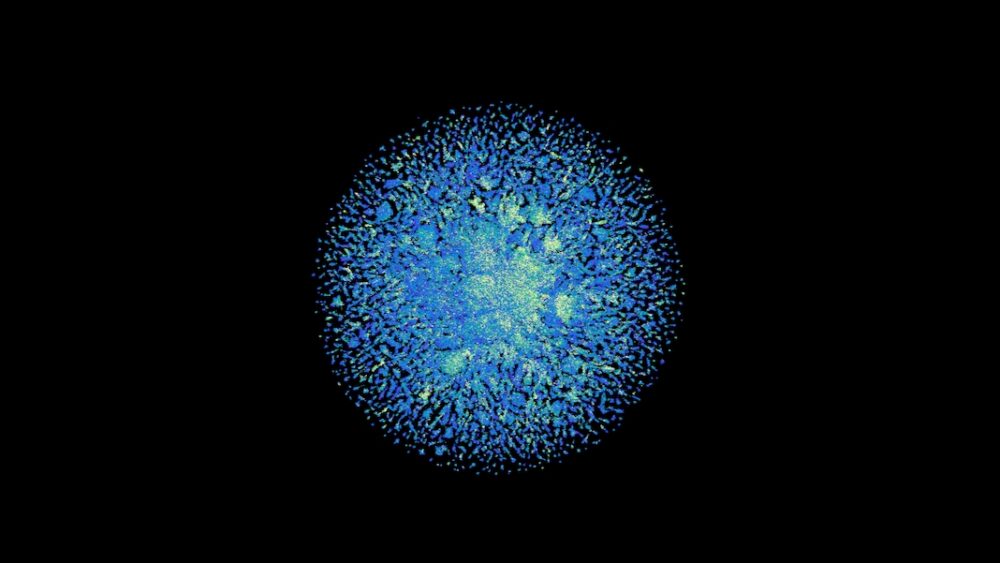
Au moment de la publication de l'article, l'IA avait prédit plus de 600 millions de ces protéines. Le nombre est maintenant supérieur à 700 millions avec la dernière version. Les prédictions sont venues rapidement et furieusement en environ deux semaines. En revanche, les tentatives de modélisation précédentes prenaient jusqu'à 10 minutes pour une seule protéine.
Environ un tiers des prédictions de protéines étaient de haute confiance, avec suffisamment de détails pour zoomer sur l'échelle atomique. Parce que les prédictions des protéines étaient basées uniquement sur leurs séquences, des millions d'"extraterrestres" sont apparus - des structures qui ne ressemblaient à rien dans les bases de données établies ou celles précédemment testées.
"Il est intéressant de noter que plus de 10% des prédictions concernent des protéines qui ne ressemblent en rien à d'autres protéines connues", a déclaré Valencia. Cela pourrait être dû à la magie des modèles de langage, qui sont beaucoup plus flexibles pour explorer - et potentiellement générer - des séquences inédites qui composent les protéines fonctionnelles. "Il s'agit d'un nouvel espace pour la conception de protéines avec de nouvelles séquences et propriétés biochimiques avec des applications en biotechnologie et en biomédecine", a-t-il déclaré.
Par exemple, ESMFold pourrait potentiellement aider à déterminer les conséquences des changements d'une seule lettre dans une protéine. Appelées mutations ponctuelles, ces modifications apparemment bénignes font des ravages dans le corps, provoquant des syndromes métaboliques dévastateurs, l'anémie falciforme et le cancer. Une IA maigre, moyenne et relativement simple apporte des résultats au laboratoire de recherche biomédicale moyen, tout en augmentant les prédictions de la forme des protéines grâce à la vitesse de l'IA.
La biomédecine mise à part, une autre idée fascinante est que les protéines peuvent aider à former de grands modèles de langage d'une manière que les textes ne peuvent pas. Comme l'a expliqué Valencia, "D'une part, les séquences de protéines sont plus abondantes que les textes, ont des tailles plus définies et un degré de variabilité plus élevé. D'autre part, les protéines ont un « sens » interne fort, c'est-à-dire une relation forte entre séquence et structure, un sens ou une cohérence beaucoup plus diffus dans les textes », reliant les deux domaines dans une boucle de rétroaction vertueuse.
Crédit image: Méta IA
- Contenu propulsé par le référencement et distribution de relations publiques. Soyez amplifié aujourd'hui.
- Platoblockchain. Intelligence métaverse Web3. Connaissance Amplifiée. Accéder ici.
- La source: https://singularityhub.com/2023/03/21/metas-new-ai-is-digging-into-the-most-mysterious-proteins-on-earth/
- :est
- $UP
- 000
- 10
- a
- capacité
- A Propos
- précision
- actes
- AI
- Alexandre
- algorithme
- algorithmes
- Alphabet
- ambitieux
- quantités
- ainsi que
- Une autre
- applications
- architecture
- SONT
- artistique
- AS
- At
- Tentatives
- auteur
- auteurs
- disponibles
- moyen
- Bactéries
- Barcelona
- basé
- BE
- Gardez
- pour créer les plus
- Beauté
- car
- jusqu'à XNUMX fois
- Au-delà
- Milliards
- biologie
- biomédical
- biotechnologie
- corps
- Cerveau
- ponts
- combler
- Apportez le
- construire
- by
- appelé
- CAN
- Cancer
- maisons
- causer
- Cellules
- Canaux centraux
- certaines
- Modifications
- Voies
- caractères
- Chatbot
- ChatGPT
- COHÉRENT
- comment
- par rapport
- complet
- comprendre
- concept
- confiance
- NOUS CONTACTER
- Conséquences
- contraste
- des bactéries
- contrôles
- converger
- conversations
- pourriez
- engendrent
- crédit
- Foncé
- La matière noire
- données
- bases de données
- Déchiffrer
- défini
- Degré
- Conception
- conception
- détail
- dévastateur
- Développement
- différent
- adn
- doublé
- chacun
- même
- Anglais
- assez
- Tout
- environnementales
- environnements
- établies
- Pourtant, la
- faire une éventuelle
- Chaque
- de tous les jours
- évolution
- exemple
- expansion
- expliqué
- explorez
- Exploré
- familier
- fascinant
- RAPIDE
- plus rapide
- Fonctionnalités:
- Réactions
- alimentation
- Pieds
- Des champs
- remplir
- finale
- flexible
- suit
- Pour
- formulaire
- formé
- trouvé
- De
- fonction
- fonctionnel
- plus
- la poursuite du développement
- Général
- générer
- géant
- objectif
- GPU
- saisir
- Graphique
- l'
- cultivé
- Half
- main
- Vous avez
- Cœur
- vous aider
- aide
- caché
- Haute
- augmentation
- Comment
- HTML
- HTTPS
- i
- idée
- satellite
- in
- incroyablement
- perspicacité
- Inspiration
- intéressant
- interne
- intuitif
- impliqué
- IT
- SES
- clés
- Savoir
- connu
- laboratoire
- Labs
- langue
- gros
- Nouveautés
- dernière version
- APPRENTISSAGE
- savant
- apprentissage
- LED
- comme
- limité
- Location
- Style
- ressembler
- Lounge
- click
- machine learning
- LES PLANTES
- la magie
- a prendre une
- Fabrication
- Matériel
- Matière
- sens
- messages
- Meta
- pourrait
- million
- des millions
- Minutes
- modèle
- numériques jumeaux (digital twin models)
- PLUS
- (en fait, presque toutes)
- mystérieux
- Nationales
- Nature
- Nature
- presque
- Neurones
- Nouveauté
- nouvelles
- next
- normalement
- of
- code
- on
- ONE
- Autre
- Autres
- global
- Papier
- particulier
- pour cent
- Effectuer
- performant
- Platon
- Intelligence des données Platon
- PlatonDonnées
- Point
- défaillances
- l'éventualité
- prévoir
- prédit
- prédiction
- Prédictions
- Prévoit
- précédent
- précédemment
- processus
- les process
- traitement
- Programme
- preuve
- preuve de concept
- propriétés
- Protéines
- Protéines
- publié
- mettre
- Race
- plutôt
- relation amoureuse
- relativement
- libérer
- rappeler
- un article
- résultat
- Résultats
- Rockstar
- grossièrement
- Saïd
- même
- Escaliers intérieurs
- mise à l'échelle
- scène
- Sciences
- choisi
- supérieur
- sens
- Séquence
- Sets
- Forme
- formes
- similaires
- étapes
- unique
- tailles
- RÉSOUDRE
- quelques
- Son
- Sources
- Space
- groupe de neurones
- vitesse
- state-of-the-art
- étapes
- tempête
- STRONG
- structure
- Étude
- tel
- Superinformatique
- Système
- discutons-en
- équipe
- technologie
- géant de la technologie
- tester
- Merci
- qui
- La
- le monde
- leur
- Les
- thermique
- Ces
- Troisièmement
- Avec
- fiable
- fois
- à
- ensemble
- Train
- qualifié
- TOUR
- ultime
- sous
- compréhension
- unités
- Univers
- virus
- Façon..
- Semaines
- a accueilli
- bien connu
- Quoi
- qui
- tout en
- WHO
- comprenant
- sans
- Word
- des mots
- activités principales
- travailler ensemble
- world
- écrire
- Vous n'avez
- Votre
- zéphyrnet
- zoom