Bevezetés
A 2000-es évek elején a Human Genome Project során felfedezett felfedezés, miszerint nekünk, embereknek csak körülbelül 20,000 XNUMX fehérjekódoló génünk van – körülbelül annyi, mint az apró talajlakó fonálféreg, és kevesebb, mint feleannyi, mint a rizsnövénynek –, sokkoló volt. . Ezt a büszkeségünkre mért csapást azonban tompította az az elképzelés, hogy az emberi genom gazdag szabályozási kapcsolatokban. Génjeink egy sűrű hálózatban lépnek kölcsönhatásba, amelyben a DNS-darabok és az általuk kódolt molekulák (RNS és fehérjék) szabályozzák más gének „kifejezését”, befolyásolva, hogy a megfelelő RNS-t és fehérjéket előállítsák-e. Az emberi genom megértéséhez meg kellett értenünk ezt a génszabályozási folyamatot.
Ez a feladat azonban sokkal nehezebbnek bizonyul, mint a genom szekvenciájának dekódolása.
Kezdetben azt gyanították, hogy a génszabályozás egyszerűen abból adódik, hogy az egyik géntermék egy másik gén be-/kikapcsolójaként működik, digitális módon. Az 1960-as években François Jacob és Jacques Monod francia biológusok először tisztázták génszabályozó folyamat mechanikai részletekben: In Escherichia coli Ha egy represszor fehérje a DNS egy bizonyos szegmenséhez kötődik, akkor blokkolja a szomszédos génkészletek transzkripcióját és transzlációját, amelyek a cukor-laktóz emésztésére szolgáló enzimeket kódolják. Ez a szabályozó áramkör, amelyet Monod és Jacob a lac operon, tiszta, átlátható logikája van.
De a génszabályozás összetett metazoánokban – az emberekhez hasonló állatokban, összetett eukarióta sejtekkel – általában nem így működik. Ehelyett molekulák csoportját foglalja magában, beleértve a fehérjéket, RNS-eket és a kromoszómából származó DNS-darabokat, amelyek valamilyen módon együttműködnek egy gén expressziójának szabályozásában.
Nem csak arról van szó, hogy az eukariótákban ennek a szabályozási folyamatnak több szereplője van, mint általában a baktériumokban és más egyszerű, prokarióta sejtekben; úgy tűnik, ez egy kategorikusan más folyamat, és egy homályosabb.
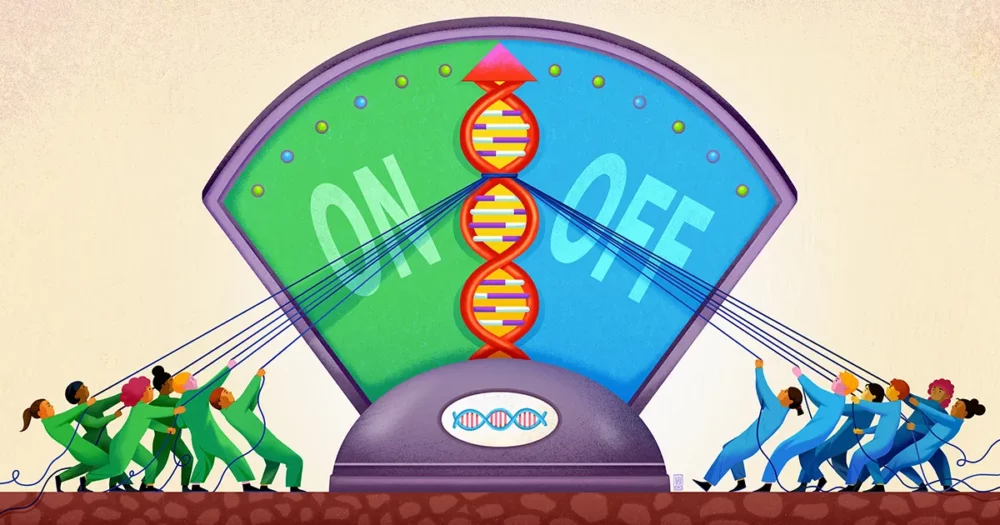
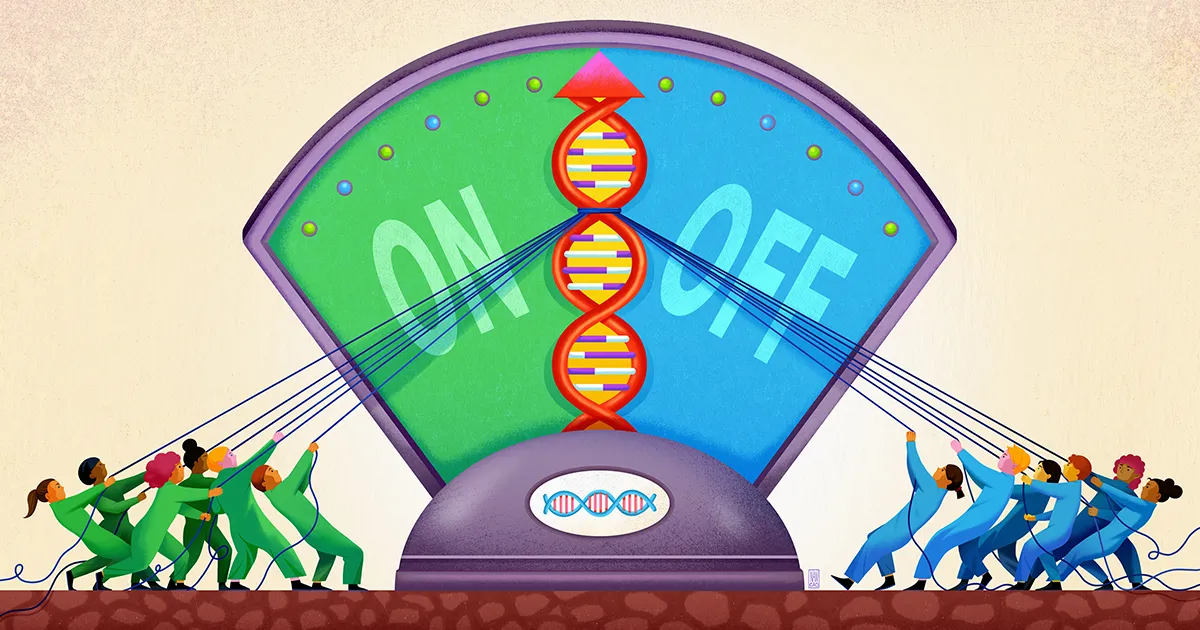
A Stanford Egyetem csapata, a biofizikus és biomérnök vezetésével Polly Fordyce, úgy tűnik, most feltárta a génszabályozás ezen elmosódott módjának egy összetevőjét. A munkájuk, amely tavaly szeptemberben jelent meg Tudomány, azt sugallja, hogy a gén közelében lévő DNS egyfajta sekély kútként működik a különféle szabályozó molekulák csapdába ejtésére, készen tartva őket a cselekvésre, hogy szükség esetén hozzászólhassanak a gén aktiválásáról szóló döntéshez.
Bevezetés
Ezek a szabályozó lyukak határozottan páratlan DNS-szakaszokból készülnek. Olyan szekvenciákból állnak, amelyekben egy rövid, egy-hat bázispár hosszúságú DNS-szakasz sokszor ismétlődik. Ezeknek a „rövid tandem ismétlődéseknek” (STR) több tíz példányát lehet összefűzni ezekben a sorozatokban, mint ugyanazt a kis „szót”, amelyet újra és újra leírnak.
Az STR-k bőségesek az emberi genomban: az összes DNS-ünk körülbelül 5%-át teszik ki. Valaha a „szemét” DNS klasszikus példáinak tartották őket, mivel egy ismétlődő DNS „szöveg”, amely csak STR-ekből áll, közel sem tud annyi értelmes információt tárolni, mint mondjuk a mondatot alkotó szabálytalan betűsorozat. cikk.
Ennek ellenére az STR-k nyilvánvalóan nem jelentéktelenek: összefüggésbe hozhatók olyan betegségekkel, mint a Huntington-kór, a spinobulbaris izomsorvadás, a Crohn-betegség és néhány rák. Az elmúlt néhány évtized során bizonyítékok gyűltek fel arra vonatkozóan, hogy ezek valamilyen módon fokozhatják vagy gátolhatják a génszabályozást. A rejtély az volt, hogyan lehetnek ilyen erősek ilyen kevés információtartalommal.
Komplex vezérlők összetett sejtekhez
Ahhoz, hogy megértsük, hogyan illeszkednek az STR-k a génszabályozás átfogó képébe, tegyünk egy lépést hátra. A géneket rutinszerűen olyan DNS-darabok szegélyezik, amelyek nem kódolnak RNS-t vagy fehérjét, de szabályozó funkciókkal rendelkeznek. A bakteriális géneknek vannak „promoter” régiói, ahol a polimeráz enzimek kötődhetnek, hogy megkezdjék a szomszédos DNS RNS-vé történő transzkripcióját. Rutinszerűen rendelkeznek „operátor” régiókkal is, ahol a represszor fehérjék kötődhetnek, hogy blokkolják a transzkripciót, kikapcsolva egy gént, mint a lac operon.
Az emberekben és más eukariótákban a szabályozó szekvenciák számosabbak, változatosabbak és zavarba ejtőek lehetnek. Az enhanszernek nevezett régiók például befolyásolják a gén átírásának valószínűségét. Az enhancerek gyakran a transzkripciós faktoroknak nevezett fehérjék célpontjai, amelyek kötődhetnek, hogy fokozzák vagy gátolják a génexpressziót. Furcsa módon egyes fokozók több tízezer bázispár távolságra vannak az általuk szabályozott génektől, és csak a DNS hurkjainak fizikai átrendeződése révén kerülnek közel hozzájuk egy csomagolt kromoszómában.
Az eukarióta génszabályozás jellemzően magában foglalja a DNS sokféle szabályozó blokkját, egy vagy több transzkripciós faktorral és más molekulákkal együtt, amelyek mindegyike egy gén körül gyűlik össze, mint egy bizottság, amelyet azért hívtak össze, hogy eldöntse, mit tegyen. Laza, sűrű fürtben gyűlnek össze.
Gyakran úgy tűnik, hogy a molekuláris résztvevők nem lépnek kölcsönhatásba a molekuláris biológiában megszokott rendkívül szelektív „zár és kulcs” párosításokon keresztül. Ehelyett sokkal kevésbé válogatósak, meglehetősen gyengén és nem szelektíven lépnek kapcsolatba, mintha kóborolnának és rövid beszélgetéseket folytatnának egymással.
Valójában rejtély volt, hogy a transzkripciós faktorok hogyan kötődnek a DNS-hez az eukariótákban. Régóta feltételezték, hogy a transzkripciós faktor egy részének szorosan meg kell egyeznie a DNS-ben található kötő „motívum” szekvenciával, például egy kirakós játék darabjaival. De bár azonosítottak néhány ilyen motívumot, jelenlétük nem mindig nagyon korrelál azzal, hogy a tudósok hol találják a DNS-hez tapadt transzkripciós faktorokat a sejtekben. Néha a transzkripciós faktorok a motívumok nélküli régiókban maradnak meg, míg egyes motívumok, amelyeknek úgy tűnik, hogy erősen kötniük kellene a transzkripciós faktorokat, üresek maradnak.
"Hagyományosan a genomikában a cél az volt, hogy a genomi helyeket [bináris] módon "kötött" vagy "nem kötött"" osztályba sorolják a transzkripciós faktorok segítségével" - mondta Fordyce. "De a kép ennél sokkal árnyaltabb." Úgy tűnik, hogy a génszabályozó „bizottságok” egyes tagjai nem mindig jelen vannak az üléseiken, vagy hiányoznak onnan, hanem eltérő valószínűséggel vannak jelen vagy sem.
A biofizikus szerint az eukarióták génszabályozásának tendenciája, hogy a nagy molekuláris komplexek sokféle, gyenge kölcsönhatásán alapul Thomas Kuhlman a Riverside-i Kaliforniai Egyetem munkatársa, aki írta egy kommentár a Fordyce labor papírján Tudomány. Mély rejtély, hogy ebből a látszólag kaotikus folyamatból hogyan születnek pontos döntések a gének be- és kikapcsolására vonatkozóan.
A döntési folyamat titokzatos, homályos logikáján túl az a kérdés is felmerül, hogy a bizottság összes tagja hogyan találja el az utat a megfelelő helyiségbe – és hogyan marad ott. A molekulák általában diffúzió útján mozognak a sejt körül, az összes többi környező molekulától, például víztől, és véletlenszerű irányokba vándorolnak. Arra számíthatunk, hogy ezek a laza bizottságok túl gyorsan válnak szét ahhoz, hogy elláthassák szabályozói munkájukat.
Fordyce és kollégái szerint itt lépnek be az STR-k. Az STR-ek feltűnően gyakoriak a DNS enhanszer helyein. Írásukban a kutatók azzal érvelnek, hogy az STR-k ragadós foltokként működnek, amelyek transzkripciós faktorokat idéznek elő, és megakadályozzák, hogy eltévedjenek.
A ragadósság finomhangolása
Fordyce csoportja szisztematikusan azt vizsgálta, hogy az STR-szekvenciák különbségei hogyan befolyásolják a transzkripciós faktorok kötőmotívumhoz való tapadását. Két tényezőt vizsgáltak – az egyiket az élesztőből, a másikat az emberből –, amelyek egy adott hatbázisú motívumhoz tapadnak. A kutatók mérték a kötődés erősségét (vagy affinitását), valamint azt a sebességet, amellyel a transzkripciós faktorok leragadnak és leragadnak (kinetika), amikor a motívumot véletlenszerű szekvencia helyett STR szegélyezi. Összehasonlításképpen megvizsgálták, hogy a faktorok milyen könnyen kötődnek önmagában az STR-hez és egy teljesen véletlenszerű DNS-szekvenciához.
"Az egyik legnagyobb kihívás ezen a területen az, hogy szétválasztjuk a számtalan változót, amelyek befolyásolják a [transzkripciós faktor] kötődését a genom egy adott helyén" - mondta. David Suter, a svájci Lausanne-i Szövetségi Technológiai Intézet molekuláris biológusa. A DNS alakja, más DNS-szegmensekhez való közelsége és a DNS-molekulák fizikai feszültsége egyaránt szerepet játszhat a transzkripciós faktorok megkötésében. Ezeknek a paramétereknek az értéke valószínűleg a genom minden pozíciójában, és talán sejttípusonként és egyetlen sejten belül is idővel egy adott pozícióban eltér. "Ez az ismeretlen változók hatalmas tere, amelyeket nagyon nehéz számszerűsíteni" - mondta Suter.
Bevezetés
Ezért olyan hasznosak a jól kontrollált kísérletek, mint a Stanford csapaté, tette hozzá Kuhlman. Általában, amikor a kutatóknak ilyen gyenge kölcsönhatásokat kell mérniük, két választásuk van: végezhetnek néhány nagyon részletes, rendkívül pontos mérést és általánosíthatnak belőlük, vagy végezhetnek sok gyors és piszkos mérést, és matematikailag összetettet használhatnak. statisztikai módszerek az eredmények levezetésére. Fordyce és kollégái azonban – mondta Kuhlman – egy automatizált, mikrofluidikus chip-alapú eljárást alkalmaztak, hogy precíz méréseket végezzenek nagy áteresztőképességű kísérletek során, „hogy mindkét világból a legjobbat kihozzák”.
A Stanford-csoport azt találta, hogy a különböző STR-szekvenciák akár 70-szeres mértékben is megváltoztathatják a transzkripciós faktorok DNS-hez való kötődési affinitását; néha nagyobb hatást gyakorolnak a transzkripciós faktor kötésére, mint magának a kötőmotívum szekvenciájának megváltoztatása. És a hatások eltérőek voltak az általuk vizsgált két különböző transzkripciós faktor esetében.
Tehát úgy tűnik, hogy az STR-k képesek finomhangolni a transzkripciós faktorok azon képességét, hogy egy DNS-helyen dokkoljanak, és így szabályozzák a gént. De pontosan hogyan?
Váróterem egy gén közelében
A kutatók arra jutottak, hogy a transzkripciós faktor DNS-hez kötődő része gyengén kölcsönhatásba léphet az STR-vel, ennek az affinitásnak a pontos erőssége az STR-szekvenciától függ. Mivel az ilyen kötés gyenge, nem lesz különösebb sajátossága. De ha egy transzkripciós faktort lazán megragad és felszabadít az STR újra és újra, a kumulatív hatás az, hogy a transzkripciós faktor a gén közelében marad, így nagyobb valószínűséggel kötődik biztonságosan a motívum régióhoz, ha szükséges.
Fordyce és munkatársai azt jósolták, hogy az STR-k így „lobbyként” vagy kútként működnek, ahol a transzkripciós faktorok összegyűlhetnek, bármilyen átmenetileg is, egy szabályozó kötőhely közelében. "Az STR ismétlődő jellege felerősíti bármely egyetlen kötőhely gyenge hatását, amelyből készült" - mondta. Connor Horton, a tanulmány első szerzője, aki jelenleg a Kaliforniai Egyetem doktorandusza, Berkeley.
Ezzel szemben, hozzátette, egyes STR-ek arra is hatást gyakorolhatnak, hogy elhúzzák a transzkripciós faktorokat a szabályozó szekvenciáktól, és szivacsszerűen felszívják a transzkripciós faktorokat máshol. Ily módon gátolhatják a génexpressziót.
Suter szerint a munka „meggyőzően mutatja, hogy az STR-k közvetlenül befolyásolják a transzkripciós faktorok kötődését in vitro”. Sőt, a Stanford csapata egy gépi tanulási algoritmust használt annak kimutatására, hogy az in vitro kísérleteikben látott hatások élő sejtekben (vagyis in vivo) is jelentkeznek.
De Robert Tjian, a Berkeley biokémikusa és a Howard Hughes Medical Institute kutatója úgy véli, még túl korai lehet tudni, hogy egy adott STR-transzkripciós faktor kombináció milyen hatással van a génexpresszióra a valódi sejtekben.
Tjian, Xavier Darzacq és a Berkeley-i laboratóriumban dolgozó kollégáik egyetértenek abban, hogy úgy tűnik, hogy az STR-k módot kínálnak a transzkripciós faktorok génszabályozó helyek közelében történő koncentrálására. Ennek ellenére nehéz megérteni ennek az eredménynek a funkcionális jelentőségét anélkül, hogy tudnánk, milyen közel kell lenniük a tényezőknek a transzkripció aktiválásához. Tjian azt mondta, szeretné látni, hogy egy STR élő sejtbe történő bejuttatása előre láthatóan befolyásolja-e a célgén expresszióját. Jelenleg „nincs meggyőződve arról, hogy az STR-k szükségszerűen a [szabályozási] mechanizmusok fő szempontjai lesznek in vivo”.
Kombinatorikus nyelvtan
Az egyik elhúzódó fejtörés az, hogy egy ilyen mechanizmus hogyan biztosítja megbízhatóan azt a típusú precíz génszabályozást, amelyre a sejteknek szüksége van, mivel az STR-lyukakon belüli transzkripciós faktor kötődés ereje és szelektivitása egyaránt gyenge. Fordyce úgy véli, hogy a hatás ilyen specifikussága számos forrásból származhat – nemcsak az STR-szekvenciák különbségeiből, hanem a transzkripciós faktorok és a szabályozásban részt vevő más fehérjék közötti kooperatív kölcsönhatásokból is.
Mindezek ismeretében Horton szerint nem világos, hogy egyértelmű lenne megjósolni egy adott STR-transzkripciós faktor kombináció hatását egy gén expressziójára. A folyamat logikája valóban homályos. A hatás „grammatikája” pedig valószínűleg kombinatorikus, tette hozzá Horton: Az eredmény a transzkripciós faktorok és más molekulák különböző kombinációitól függ.
A Stanford csapata úgy gondolja, hogy a transzkripciós faktorok talán 90%-a érzékeny az STR-ekre, de sokkal több fajta transzkripciós faktor van az emberi genomban, mint az STR-típusok. "Egy STR szekvencia mutációja 20 különböző transzkripciós faktor kötődését befolyásolhatja abban a sejttípusban, ami a közeli gén transzkripciójának általános csökkenéséhez vezet anélkül, hogy bármilyen specifikus transzkripciós faktort befolyásolna" - mondta Horton.
Valójában tehát a Stanford csapata egyetért Tjiannal abban, hogy az élő sejtekben a génszabályozást nem egyetlen egyszerű mechanizmus fogja vezérelni. Inkább a transzkripciós faktorok, DNS-kötőhelyeik és más szabályozó molekulák sűrű csoportokká gyűlhetnek össze, amelyek együttesen fejtik ki hatásukat.
"Ma már több példa is alátámasztja azt az elképzelést, hogy a DNS-elemek a transzkripciós faktorokat annyira összetorlhatják, hogy kondenzátumot képeznek a kofaktorokkal" Richard Young, a Massachusetts Institute of Technology Whitehead Intézetének sejtbiológusa. Az enhancerek sok transzkripciós faktort megkötnek a zsúfoltság előidézése érdekében. Az STR-k olyan összetevők lehetnek, amelyek elősegítik a transzkripciós faktorok összegyűjtését egy gén közelében, de nem ők jelentik a teljes történetet.
Miért kell ilyen bonyolult módon szabályozni a géneket, ahelyett, hogy a szabályozó fehérjék és a DNS-helyek közötti erős és specifikus kölcsönhatásokra hagyatkoznánk, amelyek dominálnak a prokariótákban? Lehetséges, hogy ez az elmosódottság az, ami egyáltalán lehetővé tette a nagy, összetett metazoánokat.
Ahhoz, hogy életképes faj legyen, az organizmusoknak képesnek kell lenniük fejlődni és alkalmazkodni a változó körülményekhez. Ha sejtjeink a génszabályozó kölcsönhatások valamilyen hatalmas, de szigorúan előírt hálózatára támaszkodnának, nehéz lenne bármiféle változtatást végrehajtani rajta anélkül, hogy az egész szerkezetet megzavarnánk, ahogyan egy svájci óra is felkapaszkodik, ha eltávolítunk (vagy akár kissé el is toljuk) számtalan fogaskerekéből. Ha azonban a szabályozó molekuláris kölcsönhatások lazák és meglehetősen nem specifikusak, akkor a rendszerben hasznos lazaság van – ahogy egy bizottság általában akkor is jó döntést tud hozni, ha valamelyik tagja beteg.
Fordyce megjegyzi, hogy a prokariótákban, például a baktériumokban, a transzkripciós faktorok viszonylag könnyen megtalálhatják kötőhelyeiket, mivel a keresendő genom kisebb. De ez egyre nehezebbé válik, ahogy a genom nő. Az eukarióták nagy genomjában „többé nem tudja elviselni azt a kockázatot, hogy átmenetileg egy „rossz” kötési helyen ragadjon meg” – mondta Fordyce, mert ez veszélyeztetné a változó környezeti feltételekre való gyors reagálás képességét.
Sőt, maguk az STR-k is nagyon fejleszthetők. A szekvenciájuk meghosszabbítása vagy lerövidülése, vagy a „transzkripciós faktor lyuk” méretének és mélységének megváltozása könnyen bekövetkezhet a DNS-replikáció vagy -javítás hibáiból, vagy a kromoszómák szexuális rekombinációjából. Fordyce szerint ez azt sugallja, hogy az STR-k „ezért nyersanyagként szolgálhatnak új szabályozási elemek kidolgozásához és a meglévő szabályozási modulok finomhangolásához az érzékeny transzkripciós programokhoz”, például az állatok és növények fejlődését szabályozókhoz.
A gyenge kölcsönhatások ereje
Az ilyen megfontolások arra késztetik a molekuláris biológusokat, hogy sokkal nagyobb figyelmet fordítsanak a genom gyenge és viszonylag nem szelektív kölcsönhatásaira. Ezek közül sok olyan fehérjéket foglal magában, amelyek ahelyett, hogy rögzített és precíz szerkezetük lenne, hanem lazák és hajlékonyak – a biokémikusok megfogalmazása szerint „lényegében rendezetlenek”. Ha a fehérjék csak merev szerkezeti tartományokon keresztül működnének, magyarázta Young, az nemcsak azt korlátozná, hogy a szabályozórendszerek milyen jól fejlődhetnek, hanem az életben tapasztalható dinamikus szabályozások fajtáit is. „Nem fogsz találni olyan élő szervezetet – vagy akár vírust –, amely csak olyan stabil szerkezeti elemekkel működne, mint egy svájci karórában” – mondta Young.
Talán az evolúció csak ráakadt az STR-ekre, mint az eukarióták génszabályozásának ilyen összetett, de végül hatékonyabb megoldására. Maguk az STR-k többféle módon keletkezhetnek – például a DNS-replikáció hibáiból vagy a transzponálható elemeknek nevezett DNS-szakaszok aktivitásából, amelyek másolatokat készítenek magukról a genomban.
"Csak úgy történt, hogy a fehérjék és az ismétlődő szekvenciák közötti kialakuló gyenge kölcsönhatások olyan dolgok voltak, amelyek … szelektív előnyt jelenthetnek a sejtek számára, ahol ez előfordult" - mondta Kuhlman. Úgy sejti, hogy ezt a homályosságot valószínűleg az eukariótákra kényszerítették, de „később ki tudták használni [ezt] saját hasznukra”. A baktériumok és más prokarióták jól meghatározott „digitális” szabályozási logikára támaszkodhatnak, mivel sejtjeik általában csak néhány egyszerű, elkülönülő állapotban léteznek, például mozognak és replikálódnak.
De a metazoánok különböző sejtállapotai „sokkal összetettebbek, és néha közel állnak egy kontinuumhoz” – mondta Suter, ezért jobban szolgálja őket a fuzzierabb „analóg” szabályozás.
"Úgy tűnik, hogy a baktériumok és eukarióták génszabályozó rendszerei jelentősen eltértek egymástól" - értett egyet Tjian. Míg Monod állítólag egyszer megjegyezte, hogy „amire igaz E. coli igaz az elefántra” – úgy tűnik, ez nem mindig így van.
- SEO által támogatott tartalom és PR terjesztés. Erősödjön még ma.
- PlatoData.Network Vertical Generative Ai. Erősítse meg magát. Hozzáférés itt.
- PlatoAiStream. Web3 Intelligence. Felerősített tudás. Hozzáférés itt.
- PlatoESG. Carbon, CleanTech, Energia, Környezet, Nap, Hulladékgazdálkodás. Hozzáférés itt.
- PlatoHealth. Biotechnológiai és klinikai vizsgálatok intelligencia. Hozzáférés itt.
- Forrás: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- :van
- :is
- :nem
- :ahol
- ][p
- $ UP
- 000
- 20
- 70
- a
- képesség
- Képes
- Rólunk
- hiányzó
- bőséges
- Felgyülemlett
- törvény
- ható
- Akció
- aktív
- tevékenység
- cselekmények
- alkalmazkodni
- hozzá
- hozzáadott
- szomszédos
- Előny
- érint
- affinitás
- újra
- egyeztetett
- egyetért
- algoritmus
- Minden termék
- kizárólag
- mentén
- Is
- Bár
- mindig
- között
- felerősíti
- an
- és a
- állatok
- Másik
- bármilyen
- külön
- VANNAK
- érvel
- felmerülhet
- körül
- cikkben
- AS
- megjelenés
- feltételezte
- At
- figyelem
- szerző
- Automatizált
- el
- vissza
- Baktériumok
- bázis
- BE
- mert
- válik
- óta
- kezdődik
- hogy
- haszon
- Berkeley
- BEST
- Jobb
- között
- Nagy
- Nagy kép
- nagyobb
- Legnagyobb
- kötődik
- kötés
- biológia
- Blokk
- Blocks
- fúj
- fellendítésére
- mindkét
- hozott
- de
- by
- Kalifornia
- hívott
- jött
- TUD
- sejt
- Cellák
- bizonyos
- kihívások
- Változások
- változó
- választás
- Kromoszómák
- körülmények
- klasszikus
- osztályoz
- világos
- világosan
- közel
- szorosan
- Fürt
- együttműködik
- munkatársai
- együttesen
- kombináció
- kombinációk
- hogyan
- bizottság
- Közös
- összehasonlítás
- bonyolult
- bonyolult
- összetevő
- kompromisszum
- Körülmények
- kapcsolatok
- megfontolások
- tartalom
- Folytonosság
- ellenőrzés
- ellenőrzések
- beszélgetések
- szövetkezet
- tudott
- Pár
- tömeg
- évtizedek
- dönt
- döntés
- határozatok
- Dekódolás
- csökkenés
- attól
- függ
- mélység
- részlet
- részletes
- Fejlesztés
- különbözik
- különbségek
- különböző
- nehéz
- Diffusion
- digitális
- digitális divat
- közvetlenül
- felfedezés
- betegség
- különböző
- számos
- dna
- do
- Dokk
- Nem
- domainek
- dominálnak
- ne
- hajtott
- szinkronizált
- alatt
- dinamikus
- Korai
- könnyen
- könnyű
- hatás
- Hatékony
- hatások
- bármelyik
- elemek
- elefánt
- máshol
- felmerül
- növelése
- környezeti
- hibák
- Még
- Minden
- bizonyíték
- evolúció
- fejlődik
- fejlődik
- pontosan
- példa
- példák
- létezik
- létező
- vár
- kísérletek
- magyarázható
- Exploit
- kifejezés
- rendkívüli módon
- tény
- tényező
- tényezők
- Divat
- Szövetségi
- kevés
- mező
- mintás
- Találjon
- vezetéknév
- megfelelő
- rögzített
- A
- kényszerű
- forma
- talált
- francia
- ból ből
- funkcionális
- működése
- funkciók
- Banda
- gyűjt
- gyűjtése
- összejövetelek
- általában
- genomika
- kap
- jelentkeznek
- adott
- cél
- megy
- jó
- irányító
- nagy
- Csoport
- Szerintem
- fél
- fogantyú
- történt
- Kemény
- nehezebb
- Legyen
- tekintettel
- he
- segít
- neki
- nagyon
- övé
- tart
- Hogyan
- azonban
- HTML
- HTTPS
- hatalmas
- emberi
- Az emberek
- ötlet
- azonosított
- if
- Hatás
- in
- Beleértve
- valóban
- egyéni
- befolyás
- befolyásoló
- információ
- jelentéktelen
- helyette
- Intézet
- kölcsönhatásba
- kölcsönható
- kölcsönhatások
- bele
- bevezetéséről
- változatlanul
- vonja
- részt
- jár
- IT
- ITS
- maga
- Jacob
- lombfűrész
- Munka
- éppen
- Tart
- tartás
- Kedves
- fajta
- Ismerve
- labor
- nagy
- keresztnév
- vezető
- tanulás
- Led
- kevesebb
- élet
- mint
- Valószínű
- összekapcsolt
- kis
- élő
- logika
- Hosszú
- hosszabb
- nézett
- gép
- gépi tanulás
- készült
- magazin
- fontos
- csinál
- KÉSZÍT
- mód
- sok
- Massachusetts
- Massachusetts Institute of Technology
- Mérkőzés
- anyag
- matematikailag
- Anyag
- Lehet..
- talán
- jelentőségteljes
- intézkedés
- mérések
- mechanizmus
- mechanizmusok
- orvosi
- találkozók
- Partnerek
- mód
- esetleg
- MIT
- Mód
- Modulok
- molekuláris
- molekula
- több
- mozog
- mozgó
- sok
- többszörös
- kell
- számtalan
- titokzatos
- Rejtély
- Természet
- Közel
- közel
- szükségszerűen
- Szükség
- szükséges
- hálózat
- nem
- Megjegyzések
- Most
- árnyalt
- számos
- előfordul
- történt
- előforduló
- of
- kedvezmény
- ajánlat
- gyakran
- on
- egyszer
- ONE
- csak
- or
- Más
- mi
- ki
- Eredmény
- felett
- átfogó
- saját
- csomagolt
- párosítások
- párok
- Papír
- paraméterek
- rész
- résztvevők
- különös
- múlt
- Patches
- Fizet
- talán
- rábeszélte
- fizikai
- kép
- darabok
- Telephelyek (Plants)
- Plató
- Platón adatintelligencia
- PlatoData
- játszani
- játékos
- pont
- pozíció
- lehetséges
- hatalom
- erős
- pontos
- előre
- jósolt
- jelenlét
- be
- büszkeség
- valószínűleg
- eljárás
- folyamat
- gyárt
- Termékek
- mélységes
- Programok
- program
- Fehérje
- Fehérjék
- ad
- biztosít
- bizonyítja
- közzétett
- tesz
- kirakós játék
- kérdés
- gyorsan
- egészen
- véletlen
- Arány
- Inkább
- Nyers
- készségesen
- kész
- igazi
- vidék
- régiók
- Szabályoz
- Szabályozás
- szabályozók
- viszonylag
- felszabaduló
- támaszkodnak
- támaszkodva
- marad
- jegyezte
- eltávolítása
- javítás
- ismétlő
- replikáció
- kutatók
- azok
- Reagálni
- eredményez
- kapott
- Eredmények
- Rizs
- Gazdag
- jobb
- merev
- Kockázat
- Folyóparti
- RNS
- Szerep
- Szoba
- rutinszerűen
- futás
- Mondott
- azonos
- azt mondják
- tudósok
- biztosan
- lát
- látszik
- látszólag
- Úgy tűnik,
- látott
- részes
- szegmensek
- Ragadd
- szelektív
- érzékeny
- mondat
- szeptember
- Sorozat
- szolgál
- szolgált
- számos
- Szexuális
- sekély
- Alak
- rövid
- kellene
- előadás
- jelentőség
- Egyszerű
- óta
- egyetlen
- weboldal
- Webhely (ek)
- SIX
- Méret
- laza
- kisebb
- So
- megoldások
- néhány
- valahogy
- valami
- néha
- Források
- Hely
- különleges
- sajátosság
- stabil
- Stanford
- Stanford Egyetem
- Államok
- statisztikai
- tartózkodás
- Lépés
- ragasztás
- ragadós
- megáll
- Történet
- egyértelmű
- erő
- erős
- erősen
- szerkezeti
- struktúra
- diák
- Tanulmány
- Később
- lényegesen
- ilyen
- cukor
- javasolja,
- kíséret
- támogatás
- biztos
- környező
- Svájci
- kapcsoló
- svájc
- rendszer
- Systems
- Vesz
- Tandem
- cél
- célok
- Feladat
- csapat
- Technológia
- megmondja
- Inkább
- tíz
- mint
- hogy
- A
- azok
- Őket
- maguk
- akkor
- Ott.
- ebből adódóan
- Ezek
- ők
- dolgok
- Szerintem
- Azt hiszi
- ezt
- azok
- bár?
- gondoltam
- ezer
- Keresztül
- egész
- Így
- szorosan
- idő
- alkalommal
- nak nek
- együtt
- is
- Fordítás
- átlátszó
- lószerszámdíszítés
- igaz
- Turning
- kettő
- típus
- típusok
- jellemzően
- ukr
- Végül
- fedetlen
- megért
- egyetemi
- University of California
- ismeretlen
- upon
- használ
- használt
- hasznos
- rendszerint
- Értékek
- különféle
- Hatalmas
- nagyon
- életképes
- vírus
- Hang
- Várakozás
- volt
- Nézz
- Víz
- Út..
- módon
- we
- gyenge
- webp
- JÓL
- jól definiált
- Wells
- voltak
- Mit
- amikor
- vajon
- ami
- míg
- WHO
- egész
- teljesen
- miért
- lesz
- val vel
- belül
- nélkül
- Munka
- dolgozott
- világ
- féreg
- lenne
- írott
- írt
- még
- te
- fiatal
- zephyrnet