Wprowadzenie
Odkrycie w ramach projektu Human Genome Project na początku XXI wieku, że my, ludzie, mamy tylko około 2000 20,000 genów kodujących białka – mniej więcej tyle, co maleńki nicień żyjący w glebie i mniej niż połowę mniej niż roślina ryżowa – było szokiem . Uderzenie w naszą dumę zostało jednak złagodzone przez koncepcję, że ludzki genom jest bogaty w powiązania regulacyjne. Nasze geny oddziałują na siebie w gęstej sieci, w której fragmenty DNA i cząsteczki, które kodują (RNA i białka) kontrolują „ekspresję” innych genów, wpływając na to, czy wytwarzają one odpowiednie RNA i białka. Aby zrozumieć ludzki genom, musieliśmy zrozumieć proces regulacji genów.
To zadanie okazuje się jednak znacznie trudniejsze niż rozszyfrowanie sekwencji genomu.
Początkowo podejrzewano, że regulacja genów polega po prostu na tym, że jeden produkt genowy działa jako włącznik/wyłącznik innego genu, w sposób cyfrowy. W latach sześćdziesiątych francuscy biolodzy François Jacob i Jacques Monod po raz pierwszy wyjaśnili proces regulacji genów w szczegółach mechanicznych: In Escherichia coli bakterii, gdy białko represorowe wiąże się z pewnym segmentem DNA, blokuje transkrypcję i translację sąsiadującego zestawu genów kodujących enzymy trawiące laktozę. Ten obwód regulacyjny, który Monod i Jacob nazwali jezioro operon, ma zgrabną, przejrzystą logikę.
Jednak regulacja genów u złożonych metazoanów – zwierząt takich jak ludzie, ze złożonymi komórkami eukariotycznymi – na ogół nie działa w ten sposób. Zamiast tego obejmuje zbiór cząsteczek, w tym białka, RNA i fragmenty DNA z całego chromosomu, które w jakiś sposób współpracują, aby kontrolować ekspresję genu.
Nie chodzi tylko o to, że w procesie regulacyjnym u eukariontów bierze udział więcej uczestników, niż zwykle obserwuje się w przypadku bakterii i innych prostych komórek prokariotycznych; wydaje się, że jest to proces całkowicie odmienny i bardziej mglisty.
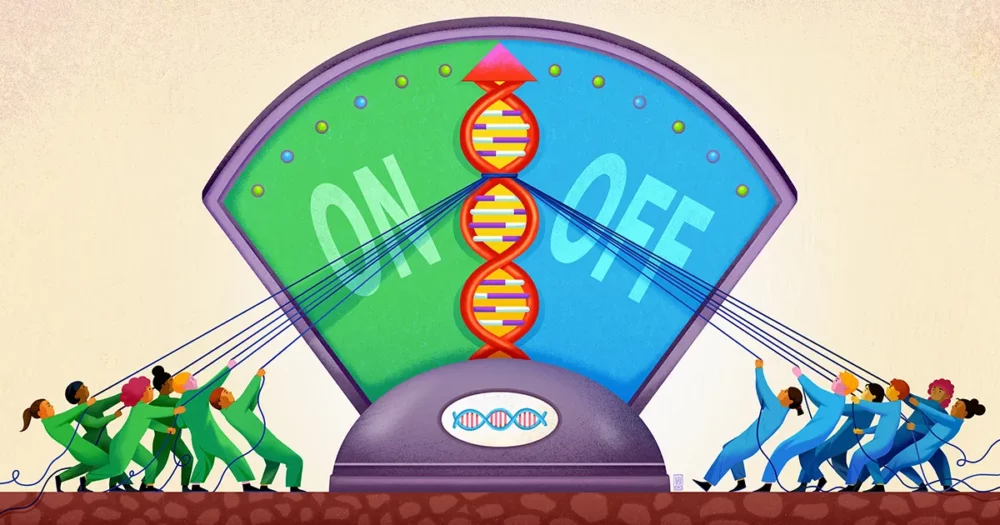
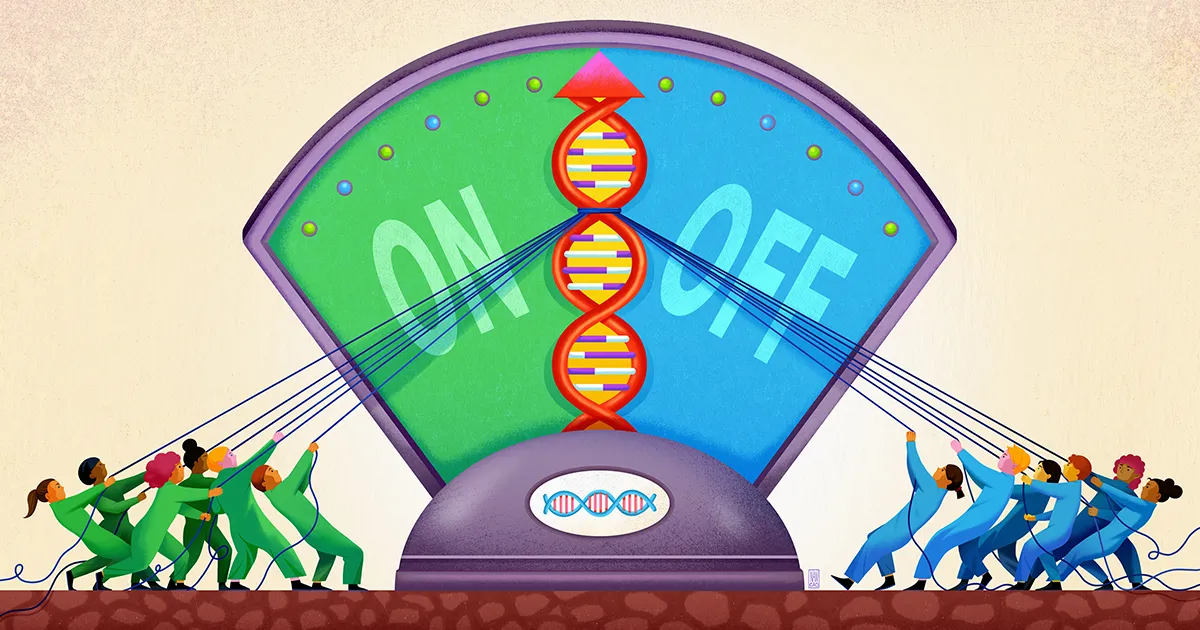
Zespół z Uniwersytetu Stanforda, kierowany przez biofizyka i bioinżyniera Polly Fordyce, wydaje się teraz, że odkrył składnik tego rozmytego trybu regulacji genów. Ich praca, opublikowane we wrześniu ubiegłego roku w nauka, sugeruje, że DNA w pobliżu genu działa jak rodzaj płytkiej studni, w której wychwytuje się różne cząsteczki regulatorowe, utrzymując je w gotowości do działania, aby w razie potrzeby mogły zabrać głos w decyzji o aktywacji genu.
Wprowadzenie
Te studzienki regulacyjne składają się z zdecydowanie dziwnych odcinków DNA. Składają się z sekwencji, w których krótki odcinek DNA o długości od jednej do sześciu par zasad powtarza się wielokrotnie. Dziesiątki kopii tych „krótkich powtórzeń tandemowych” (STR) można połączyć w te sekwencje, jak to samo małe „słowo” pisane w kółko.
STR są liczne w ludzkim genomie: stanowią około 5% całego naszego DNA. Kiedyś uważano je za klasyczne przykłady „śmieciowego” DNA, ponieważ powtarzający się „tekst” DNA składający się wyłącznie z STR nie może zawierać tak wielu znaczących informacji, jak, powiedzmy, nieregularna sekwencja liter tworzących zdanie w tym artykuł.
A jednak STR wyraźnie nie są bez znaczenia: powiązano je z takimi dolegliwościami, jak choroba Huntingtona, rdzeniowo-opuszkowy zanik mięśni, choroba Leśniowskiego-Crohna i niektóre nowotwory. W ciągu ostatnich kilku dekad zgromadzono dowody na to, że mogą one w jakiś sposób wzmacniać lub hamować regulację genów. Tajemnicą było to, jak mogli być tak potężni przy tak małej zawartości informacji.
Złożone kontrole dla złożonych komórek
Aby zrozumieć, jak STR wpisują się w szerszy obraz regulacji genów, cofnijmy się o krok. Geny są rutynowo otoczone fragmentami DNA, które nie kodują RNA ani białka, ale pełnią funkcje regulacyjne. Geny bakteryjne mają regiony „promotorowe”, z którymi mogą wiązać się enzymy polimerazy, rozpoczynając transkrypcję sąsiedniego DNA na RNA. Rutynowo posiadają także regiony „operatorowe”, gdzie białka represorowe mogą wiązać się, blokując transkrypcję, wyłączając gen, jak w jezioro operon.
U ludzi i innych eukariontów sekwencje regulatorowe mogą być liczniejsze, różnorodne i kłopotliwe. Na przykład regiony zwane wzmacniaczami wpływają na prawdopodobieństwo transkrypcji genu. Wzmacniacze są często celem dla białek zwanych czynnikami transkrypcyjnymi, które mogą wiązać się w celu zwiększenia lub zahamowania ekspresji genów. Co dziwne, niektóre wzmacniacze znajdują się dziesiątki tysięcy par zasad od genów, które regulują, i zbliżają się do nich jedynie w wyniku fizycznej rearanżacji pętli DNA w upakowanym chromosomie.
Regulacja genów eukariotycznych zazwyczaj obejmuje wiele różnorodnych bloków regulacyjnych DNA, wraz z jednym lub większą liczbą czynników transkrypcyjnych i innymi cząsteczkami, a wszystko to gromadzi się wokół genu niczym komitet zwołany w celu podjęcia decyzji, co powinien zrobić. Gromadzą się w luźnym, gęstym skupisku.
Często wydaje się, że uczestnicy molekularni nie wchodzą w interakcje poprzez wysoce selektywne pary „zamek i klucz”, powszechne w biologii molekularnej. Zamiast tego są znacznie mniej wybredni, oddziałując raczej słabo i nieselektywnie, jakby błąkali się i nawiązywali między sobą krótkie rozmowy.
W rzeczywistości sposób, w jaki czynniki transkrypcyjne wiążą się z DNA u eukariontów, pozostaje tajemnicą. Długo zakładano, że jakaś część czynnika transkrypcyjnego musi ściśle odpowiadać wiążącej sekwencji „motywu” w DNA, podobnie jak elementy układanki. Chociaż zidentyfikowano kilka takich motywów, ich obecność nie zawsze dobrze koreluje z miejscem, w którym naukowcy znajdują czynniki transkrypcyjne przyczepione do DNA w komórkach. Czasami czynniki transkrypcyjne pozostają w regionach bez żadnych motywów, podczas gdy niektóre motywy, które wydają się, jakby powinny silnie wiązać czynniki transkrypcyjne, pozostają puste.
„Tradycyjnie w genomice celem była klasyfikacja miejsc genomowych w sposób [binarny] jako „związane” lub „niezwiązane”” na podstawie czynników transkrypcyjnych, powiedział Fordyce. „Ale obraz jest o wiele bardziej złożony”. Wydaje się, że poszczególni członkowie tych „komitetów” ds. regulacji genów nie zawsze są obecni na swoich spotkaniach lub nieobecni, ale raczej mają różne prawdopodobieństwo obecności lub nie.
Tendencja regulacji genów u eukariontów do opierania się na tak wielu różnorodnych słabych interakcjach między dużymi kompleksami molekularnymi „jest jedną z rzeczy, która sprawia, że niezwykle trudno jest sobie z tym poradzić teoretycznie” – powiedział biofizyk Tomasz Kuhlman z Uniwersytetu Kalifornijskiego w Riverside, który napisał komentarz w artykule laboratorium Fordyce nauka. To wielka zagadka, jak z tego pozornie chaotycznego procesu wyłaniają się precyzyjne decyzje dotyczące włączania i wyłączania genów.
Oprócz tajemniczej, niejasnej logiki procesu decyzyjnego pojawia się również pytanie, w jaki sposób wszyscy członkowie komisji w ogóle trafiają do odpowiedniego pokoju i pozostają tam. Cząsteczki zazwyczaj poruszają się po komórce na drodze dyfuzji, są atakowane przez wszystkie inne otaczające cząsteczki, takie jak woda, i wędrują w przypadkowych kierunkach. Można by się spodziewać, że te luźne komitety zbyt szybko się rozdzielą, aby wykonać swoje zadania regulacyjne.
To właśnie, zdaniem Fordyce i jej współpracowników, właśnie tu z pomocą przychodzą STR. STR są uderzająco powszechne w miejscach wzmacniacza w DNA. W swoim artykule naukowcy argumentują, że STR działają jak lepkie plastry, które skupiają czynniki transkrypcyjne i zapobiegają ich przemieszczaniu się.
Dostrajanie lepkości
Grupa Fordyce'a systematycznie badała, w jaki sposób różnice w sekwencji STR wpływają na przyleganie czynników transkrypcyjnych do motywu wiążącego. Przyjrzeli się dwóm czynnikom – jednemu pochodzącemu od drożdży, drugiemu od człowieka – które wiążą się z konkretnym motywem sześciu zasad. Naukowcy zmierzyli zarówno siłę (lub powinowactwo) tego wiązania, jak i szybkość, z jaką czynniki transkrypcyjne ulegają zablokowaniu i odklejeniu (kinetyka), gdy motyw jest otoczony przez STR zamiast losowej sekwencji. Dla porównania sprawdzili, jak łatwo czynniki wiążą się z samym STR i całkowicie losową sekwencją DNA.
„Jednym z największych wyzwań w tej dziedzinie jest rozwikłanie niezliczonych zmiennych, które wpływają na wiązanie [czynnika transkrypcyjnego] w określonej pozycji genomu” – powiedział Dawid Suter, biolog molekularny w Szwajcarskim Federalnym Instytucie Technologii w Lozannie w Szwajcarii. Kształt DNA, bliskość innych segmentów DNA i napięcie fizyczne w cząsteczkach DNA mogą odgrywać rolę w wiązaniu czynników transkrypcyjnych. Wartości tych parametrów prawdopodobnie różnią się w każdej pozycji w genomie, a być może także między typami komórek oraz w obrębie pojedynczej komórki w czasie w danej pozycji. „To ogromna przestrzeń nieznanych zmiennych, które bardzo trudno jest określić ilościowo” – powiedział Suter.
Wprowadzenie
Dlatego dobrze kontrolowane eksperymenty, takie jak te przeprowadzone przez zespół ze Stanforda, są tak przydatne, dodał Kuhlman. Zwykle, gdy badacze muszą zmierzyć takie słabe interakcje, mają dwie możliwości: mogą dokonać kilku bardzo szczegółowych, niezwykle precyzyjnych pomiarów i wyciągnąć z nich uogólnienia, lub mogą wykonać wiele szybkich i brudnych pomiarów i zastosować matematycznie złożone metody statystyczne do wnioskowania wyników. Kuhlman powiedziała jednak, że Fordyce i jej współpracownicy wykorzystali zautomatyzowaną procedurę opartą na chipach mikroprzepływowych do wykonywania precyzyjnych pomiarów podczas eksperymentów o dużej przepustowości, „aby uzyskać to, co najlepsze z obu światów”.
Zespół ze Stanforda odkrył, że różne sekwencje STR mogą zmieniać powinowactwo wiązania czynników transkrypcyjnych z DNA aż 70-krotnie; czasami mają większy wpływ na wiązanie czynnika transkrypcyjnego niż zmiana sekwencji samego motywu wiążącego. Efekty były różne w przypadku dwóch różnych czynników transkrypcyjnych, które badali.
Zatem wydaje się, że STR są w stanie dostroić zdolność czynników transkrypcyjnych do dokowania w miejscu DNA i w ten sposób regulować gen. Ale jak dokładnie?
Poczekalnia w pobliżu genu
Naukowcy ustalili, że część czynnika transkrypcyjnego, która wiąże DNA, może słabo oddziaływać z STR, a dokładna siła tego powinowactwa zależy od sekwencji STR. Ponieważ takie wiązanie jest słabe, nie będzie miało dużej specyficzności. Jeśli jednak czynnik transkrypcyjny jest luźno chwytany i uwalniany przez STR raz za razem, skumulowany efekt polega na utrzymaniu czynnika transkrypcyjnego w pobliżu genu, dzięki czemu jest bardziej prawdopodobne, że w razie potrzeby zwiąże się bezpiecznie z regionem motywu.
Fordyce i jej współpracownicy przewidzieli, że STR działają zatem jak „lobby” lub studnia, w której czynniki transkrypcyjne mogą gromadzić się, choć przejściowo, w pobliżu miejsca wiązania regulatora. „Powtarzający się charakter STR wzmacnia słaby efekt dowolnego pojedynczego miejsca wiązania, z którego jest zbudowany” – powiedział Connora Hortona, pierwszy autor badania, który jest obecnie doktorantem na Uniwersytecie Kalifornijskim w Berkeley.
Dodał, że i odwrotnie, niektóre STR mogą również odciągać czynniki transkrypcyjne od sekwencji regulatorowych, chłonąc je gdzie indziej niczym gąbka. W ten sposób mogą hamować ekspresję genów.
Praca, stwierdził Suter, „pokazuje w przekonujący sposób, że STR bezpośrednio wpływają na wiązanie czynników transkrypcyjnych in vitro”. Co więcej, zespół ze Stanford wykorzystał algorytm uczenia maszynowego, aby wykazać, że efekty zaobserwowane w eksperymentach in vitro wydają się występować również w żywych komórkach (czyli in vivo).
Ale Roberta Tjiana, biochemik z Berkeley i badacz z Instytutu Medycznego Howarda Hughesa, uważa, że może być za wcześnie, aby mieć pewność, jaki wpływ ma dana kombinacja STR i czynników transkrypcyjnych na ekspresję genów w prawdziwych komórkach.
Tjian, Xavier Darzacq i ich koledzy z laboratorium, które wspólnie prowadzą w Berkeley, zgadzają się, że STR wydają się umożliwiać koncentrację czynników transkrypcyjnych w pobliżu miejsc regulacyjnych genów. Jednak bez wiedzy, jak blisko muszą znajdować się czynniki, aby aktywować transkrypcję, trudno jest zrozumieć funkcjonalne znaczenie tego wyniku. Tjian powiedział, że chciałby sprawdzić, czy wprowadzenie STR do żywej komórki w przewidywalny sposób wpływa na ekspresję genu docelowego. Obecnie, stwierdził, „nie jest przekonany, że STR koniecznie będą głównym aspektem mechanizmów [regulacyjnych] in vivo”.
Gramatyka kombinatoryczna
Jedną z utrzymujących się zagadek jest to, w jaki sposób taki mechanizm niezawodnie zapewnia rodzaj precyzyjnej regulacji genów, której potrzebują komórki, ponieważ zarówno siła, jak i selektywność wiązania czynników transkrypcyjnych w dołkach STR są słabe. Fordyce uważa, że taka specyfika wpływu może mieć wiele źródeł — nie tylko różnice w sekwencjach STR, ale także kooperacyjne interakcje między czynnikami transkrypcyjnymi i innymi białkami zaangażowanymi w regulację.
Biorąc to wszystko pod uwagę, stwierdził Horton, nie jest jasne, czy przewidywanie wpływu danej kombinacji STR i czynników transkrypcyjnych na ekspresję genu będzie proste. Logika tego procesu jest rzeczywiście niejasna. Horton dodał, że „gramatyka” wpływu jest prawdopodobnie kombinatoryczna: Wynik zależy od różnych kombinacji czynników transkrypcyjnych i innych cząsteczek.
Zespół ze Stanforda uważa, że być może 90% czynników transkrypcyjnych jest wrażliwych na STR, ale w ludzkim genomie istnieje o wiele więcej typów czynników transkrypcyjnych niż typów STR. „Mutowanie sekwencji STR może wpływać na wiązanie 20 różnych czynników transkrypcyjnych w komórkach tego typu, prowadząc do ogólnego zmniejszenia transkrypcji pobliskiego genu bez wpływu na jakikolwiek konkretny czynnik transkrypcyjny” – powiedział Horton.
W efekcie zespół ze Stanford zgadza się z Tjianem, że regulacja genów w żywych komórkach nie będzie sterowana jednym, prostym mechanizmem. Przeciwnie, czynniki transkrypcyjne, ich miejsca wiązania DNA i inne cząsteczki regulatorowe mogą łączyć się w gęste zbiorowiska, które wspólnie wywierają swój wpływ.
„Obecnie istnieje wiele przykładów potwierdzających tezę, że elementy DNA mogą tłoczyć czynniki transkrypcyjne do punktu, w którym tworzą kondensaty z kofaktorami” – mówi Richarda Younga, biolog komórkowy w Whitehead Institute of Massachusetts Institute of Technology. Wzmacniacze wiążą wiele czynników transkrypcyjnych, aby wytworzyć to stłoczenie. STR mogą być składnikiem, który pomaga zebrać czynniki transkrypcyjne w skupieniu w pobliżu genu, ale to nie będzie cała historia.
Po co regulować geny w tak skomplikowany sposób, zamiast polegać na rodzaju silnych i specyficznych interakcji między białkami regulatorowymi a miejscami DNA, które dominują u prokariotów? Możliwe, że właśnie taka niejasność w ogóle umożliwiła powstanie dużych, złożonych metazoanów.
Aby organizmy mogły przetrwać jako gatunki, muszą być w stanie ewoluować i dostosowywać się do zmieniających się warunków. Gdyby nasze komórki opierały się na jakiejś ogromnej, ale ściśle określonej sieci interakcji regulacyjnych genów, trudno byłoby dokonać w niej jakichkolwiek zmian bez zakłócania całego urządzenia, tak jak szwajcarski zegarek się zużyje, jeśli usuniemy (lub chociaż nieznacznie przesuniemy) jakikolwiek niezliczonych kół zębatych. Jeśli jednak regulacyjne interakcje molekularne są luźne i raczej niespecyficzne, w systemie występuje pożyteczny luz – tak jak komitet może ogólnie podjąć dobrą decyzję, nawet jeśli jeden z jego członków jest chory.
Fordyce zauważa, że u prokariotów, takich jak bakterie, czynniki transkrypcyjne mogą stosunkowo łatwo znaleźć swoje miejsca wiązania, ponieważ przeszukiwany genom jest mniejszy. Ale staje się to trudniejsze w miarę powiększania się genomu. W dużych genomach eukariontów „nie można już tolerować ryzyka, że przejściowo utkniesz w„ złym ” miejscu wiązania” – stwierdził Fordyce, ponieważ zagroziłoby to możliwości szybkiego reagowania na zmieniające się warunki środowiskowe.
Co więcej, same STR są wysoce ewolucyjne. Wydłużenie lub skrócenie ich sekwencji lub zmiana wielkości i głębokości „studni czynnika transkrypcyjnego” może łatwo nastąpić w wyniku niepowodzeń w replikacji lub naprawie DNA lub w wyniku rekombinacji płciowej chromosomów. Fordyce sugeruje, że STR „mogą zatem służyć jako surowiec do opracowywania nowych elementów regulacyjnych i dostrajania istniejących modułów regulacyjnych na potrzeby wrażliwych programów transkrypcyjnych”, takich jak te regulujące rozwój zwierząt i roślin.
Siła słabych interakcji
Takie rozważania skłaniają biologów molekularnych do zwracania znacznie większej uwagi na słabe i stosunkowo nieselektywne interakcje w genomie. Wiele z nich obejmuje białka, które zamiast mieć stałą i precyzyjną strukturę, są luźne i wiotkie – „wewnętrznie nieuporządkowane”, jak to ujęli biochemicy. Young wyjaśnił, że gdyby białka działały wyłącznie poprzez sztywne domeny strukturalne, ograniczałoby to nie tylko skuteczność ewolucji systemów regulacyjnych, ale także rodzaj dynamicznej regulacji obserwowanej w życiu. „Nie znajdziesz żywego organizmu — ani nawet wirusa — działającego wyłącznie z stabilnymi elementami strukturalnymi, takimi jak te w szwajcarskim zegarku” – powiedział Young.
Być może ewolucja po prostu natknęła się na STR jako składnik tak złożonego, ale ostatecznie skuteczniejszego rozwiązania regulacji genów u eukariontów. Same STR mogą powstawać na kilka sposobów — na przykład w wyniku błędów w replikacji DNA lub aktywności segmentów DNA zwanych elementami transpozycyjnymi, które tworzą kopie w całym genomie.
„Tak się złożyło, że powstałe w rezultacie słabe interakcje między białkami i powtarzającymi się sekwencjami mogły… zapewnić selektywną przewagę komórkom, w których to nastąpiło” – powiedział Kuhlman. Domyśla się, że eukariontom prawdopodobnie narzucono tę niejasność, ale „potem udało im się ją wykorzystać dla własnych korzyści”. Bakterie i inne prokarioty mogą polegać na dobrze zdefiniowanej „cyfrowej” logice regulacyjnej, ponieważ ich komórki zwykle istnieją tylko w kilku prostych, odrębnych stanach, takich jak poruszanie się i replikacja.
Jednak różne stany komórkowe metazoanów są „znacznie bardziej złożone i czasami bliskie kontinuum” – powiedział Suter, dlatego lepiej służy im bardziej rozmyta „analogowa” regulacja.
„Wydaje się, że systemy regulacji genów u bakterii i eukariontów znacznie się od siebie różnią” – zgodził się Tjian. Chociaż Monod podobno zauważył kiedyś, że „to, co jest prawdą E. coli to prawda w przypadku słonia”, wydaje się, że nie zawsze tak jest.
- Dystrybucja treści i PR oparta na SEO. Uzyskaj wzmocnienie już dziś.
- PlatoData.Network Pionowe generatywne AI. Wzmocnij się. Dostęp tutaj.
- PlatoAiStream. Inteligencja Web3. Wiedza wzmocniona. Dostęp tutaj.
- PlatonESG. Węgiel Czysta technologia, Energia, Środowisko, Słoneczny, Gospodarowanie odpadami. Dostęp tutaj.
- Platon Zdrowie. Inteligencja w zakresie biotechnologii i badań klinicznych. Dostęp tutaj.
- Źródło: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- :ma
- :Jest
- :nie
- :Gdzie
- ][P
- $W GÓRĘ
- 000
- 20
- 70
- a
- zdolność
- Zdolny
- O nas
- nieobecny
- obfity
- Zgromadzone
- działać
- gra aktorska
- Działania
- aktywowany
- działalność
- Dzieje Apostolskie
- przystosować
- Dodaj
- w dodatku
- sąsiadujący
- Korzyść
- oddziaływać
- powinowactwo
- ponownie
- uzgodniony
- Zgadza się
- algorytm
- Wszystkie kategorie
- sam
- wzdłuż
- również
- Chociaż
- zawsze
- wśród
- wzmacnia
- an
- i
- zwierzęta
- Inne
- każdy
- osobno
- SĄ
- argumentować
- powstać
- na około
- artykuł
- AS
- aspekt
- przypuszczalny
- At
- Uwaga
- autor
- zautomatyzowane
- z dala
- z powrotem
- Bakteria
- baza
- BE
- bo
- stają się
- być
- rozpocząć
- jest
- korzyści
- Berkeley
- BEST
- Ulepsz Swój
- pomiędzy
- Duży
- Duże zdjęcie
- większe
- Najwyższa
- związania
- wiążący
- biologia
- Blokować
- Bloki
- wiać
- podnieść
- obie
- przyniósł
- ale
- by
- California
- nazywa
- oprawa ołowiana witrażu
- CAN
- komórka
- Komórki
- pewien
- wyzwania
- Zmiany
- wymiana pieniędzy
- wybory
- Chromosomy
- okoliczności
- klasyczny
- Klasyfikuj
- jasny
- wyraźnie
- Zamknij
- dokładnie
- Grupa
- współpracować
- koledzy
- zbiorowo
- połączenie
- kombinacje
- jak
- komisja
- wspólny
- porównanie
- kompleks
- skomplikowane
- składnik
- kompromis
- Warunki
- połączenia
- Rozważania
- zawartość
- Kontinuum
- kontrola
- kontroli
- rozmowy
- spółdzielnia
- mógłby
- Para
- tłum
- lat
- zdecydować
- decyzja
- Decyzje
- Rozszyfrowanie
- spadek
- W zależności
- zależy
- głębokość
- detal
- szczegółowe
- oprogramowania
- różnić się
- Różnice
- różne
- trudny
- Transmitowanie
- cyfrowy
- moda cyfrowa
- bezpośrednio
- odkrycie
- choroba
- odrębny
- inny
- DNA
- do
- Dok
- Nie
- domeny
- Zdominować
- nie
- napędzany
- dubbingowane
- podczas
- dynamiczny
- Wcześnie
- z łatwością
- łatwo
- efekt
- Efektywne
- ruchomości
- bądź
- Elementy
- słoń
- gdzie indziej
- wyłaniać się
- wzmacniać
- środowiskowy
- Błędy
- Parzyste
- Każdy
- dowód
- ewolucja
- ewoluuje
- ewoluuje
- dokładnie
- przykład
- przykłady
- istnieć
- Przede wszystkim system został opracowany
- oczekiwać
- eksperymenty
- wyjaśnione
- Wykorzystać
- wyrażenie
- niezwykle
- fakt
- czynnik
- Czynniki
- Moda
- Federalny
- kilka
- pole
- wzorzysty
- Znajdź
- i terminów, a
- dopasować
- ustalony
- W razie zamówieenia projektu
- wymuszony
- Nasz formularz
- znaleziono
- francuski
- od
- funkcjonalny
- funkcjonowanie
- Funkcje
- Gang
- zbierać
- zebranie
- spotkania
- ogólnie
- genomika
- otrzymać
- dostaje
- dany
- cel
- będzie
- dobry
- rządzić
- wspaniały
- Zarządzanie
- zgadnąć
- Pół
- uchwyt
- się
- Ciężko
- trudniej
- Have
- mający
- he
- pomaga
- jej
- wysoko
- jego
- przytrzymaj
- W jaki sposób
- Jednak
- HTML
- HTTPS
- olbrzymi
- człowiek
- Ludzie
- pomysł
- zidentyfikowane
- if
- Rezultat
- in
- Włącznie z
- rzeczywiście
- indywidualny
- wpływ
- wpływanie
- Informacja
- nieistotny
- zamiast
- Instytut
- interakcji
- interakcji
- Interakcje
- najnowszych
- wprowadzenie
- niezmiennie
- angażować
- zaangażowany
- dotyczy
- IT
- JEGO
- samo
- Jakub
- jigsaw
- Praca
- właśnie
- Trzymać
- konserwacja
- Uprzejmy
- rodzaje
- Wiedząc
- laboratorium
- duży
- Nazwisko
- prowadzący
- nauka
- Doprowadziło
- mniej
- życie
- lubić
- Prawdopodobnie
- powiązany
- mało
- życie
- logika
- długo
- dłużej
- wyglądał
- maszyna
- uczenie maszynowe
- zrobiony
- magazyn
- poważny
- robić
- WYKONUJE
- sposób
- wiele
- massachusetts
- Instytut Technologii w Massachusetts
- Mecz
- materiał
- matematycznie
- Materia
- Może..
- może
- wymowny
- zmierzyć
- Pomiary
- mechanizm
- Mechanizmy
- medyczny
- Spotkania
- Użytkownicy
- metody
- może
- MIT
- Moda
- Moduły
- Cząsteczkowa
- cząsteczka
- jeszcze
- ruch
- przeniesienie
- dużo
- wielokrotność
- musi
- miriada
- tajemniczy
- Tajemnica
- Natura
- Blisko
- prawie
- koniecznie
- Potrzebować
- potrzebne
- sieć
- Nie
- Uwagi
- już dziś
- niuansowany
- liczny
- występować
- miejsce
- występujący
- of
- poza
- oferta
- często
- on
- pewnego razu
- ONE
- tylko
- or
- Inne
- ludzkiej,
- na zewnątrz
- Wynik
- koniec
- ogólny
- własny
- zatłoczony
- parowania
- par
- Papier
- parametry
- część
- Uczestnicy
- szczególny
- Przeszłość
- Łatki
- Zapłacić
- może
- przekonany
- fizyczny
- obraz
- sztuk
- Rośliny
- plato
- Analiza danych Platona
- PlatoDane
- Grać
- gracze
- punkt
- position
- możliwy
- power
- mocny
- precyzyjny
- przewidzieć
- Przewiduje
- obecność
- teraźniejszość
- pycha
- prawdopodobnie
- procedura
- wygląda tak
- produkować
- Produkt
- głęboki
- Programy
- projekt
- Białko
- Białka
- zapewniać
- zapewnia
- udowodnienie
- opublikowany
- położyć
- puzzle
- pytanie
- szybko
- całkiem
- przypadkowy
- Kurs
- raczej
- Surowy
- łatwo
- gotowy
- real
- region
- regiony
- Regulować
- Regulacja
- regulacyjne
- stosunkowo
- wydany
- polegać
- opierając się
- pozostawać
- zauważyłem
- usunąć
- naprawa
- powtarzalne
- replikacja
- Badacze
- osób
- Odpowiadać
- dalsze
- wynikły
- Efekt
- Ryż
- Bogaty
- prawo
- sztywny
- Ryzyko
- Nadrzeczny
- RNA
- Rola
- Pokój
- rutynowo
- run
- Powiedział
- taki sam
- powiedzieć
- Naukowcy
- bezpiecznie
- widzieć
- wydać się
- pozornie
- wydaje
- widziany
- segment
- Segmenty
- Chwycić
- selektywny
- wrażliwy
- wyrok
- wrzesień
- Sekwencja
- służyć
- służył
- kilka
- Seksualny
- płytki
- Shape
- Short
- powinien
- pokazać
- znaczenie
- Prosty
- ponieważ
- pojedynczy
- witryna internetowa
- Witryny
- SIX
- Rozmiar
- luźny
- mniejszy
- So
- rozwiązanie
- kilka
- jakoś
- coś
- czasami
- Źródła
- Typ przestrzeni
- specyficzny
- specyficzność
- stabilny
- Stanford
- Uniwersytet Stanford
- Zjednoczone
- statystyczny
- pobyt
- Ewolucja krok po kroku
- klejący
- lepki
- Stop
- Historia
- bezpośredni
- jest determinacja.
- silny
- strongly
- strukturalny
- Struktura
- student
- Badanie
- Następnie
- w zasadzie
- taki
- cukier
- Wskazuje
- apartament
- wsparcie
- pewnie
- otaczający
- Szwajcarski
- Przełącznik
- Szwajcaria
- system
- systemy
- Brać
- Tandem
- cel
- cele
- Zadanie
- zespół
- Technologia
- mówi
- Tendencję
- kilkadziesiąt
- niż
- że
- Połączenia
- ich
- Im
- sami
- następnie
- Tam.
- w związku z tym
- Te
- one
- rzeczy
- myśleć
- Myśli
- to
- tych
- chociaż?
- myśl
- tysiące
- Przez
- poprzez
- A zatem
- ciasno
- czas
- czasy
- do
- razem
- także
- Tłumaczenie
- przezroczysty
- odłowu
- prawdziwy
- Obrócenie
- drugiej
- rodzaj
- typy
- zazwyczaj
- UCR
- Ostatecznie
- odkryte
- zrozumieć
- uniwersytet
- University of California
- nieznany
- na
- posługiwać się
- używany
- użyteczny
- zazwyczaj
- Wartości
- różnorodny
- Naprawiono
- początku.
- wykonalne
- wirus
- Głos
- Czekanie
- była
- Oglądaj
- Woda
- Droga..
- sposoby
- we
- słaby
- webp
- DOBRZE
- dobrze zdefiniowane
- Wells
- były
- Co
- jeśli chodzi o komunikację i motywację
- czy
- który
- Podczas
- KIM
- cały
- całkowicie
- dlaczego
- będzie
- w
- w ciągu
- bez
- Praca
- pracował
- świat
- robak
- by
- napisany
- napisał
- jeszcze
- You
- młody
- zefirnet