- 7 lipca 2014 r.
- Wasilis Wryniotis
- . 1 komentarz
W poprzednich artykułach szczegółowo omówiliśmy Modele mieszanek procesowych Dirichleta i jak można je wykorzystać w analizie skupień. W tym artykule przedstawimy implementację Java dwa różne modele DPMM: wielowymiarowy model normalnej mieszaniny Dirichleta, który można wykorzystać do grupowania danych Gaussa oraz model wielomianowej mieszaniny Dirichleta, który służy do grupowania dokumentów. Kod Java jest open-source na licencji GPL v3 i można go bezpłatnie pobrać z Github.
Aktualizacja: Platforma uczenia maszynowego Datumbox jest teraz open-source i bezpłatna pobieranie. Zapoznaj się z pakietem com.datumbox.framework.machinelearning.clustering, aby zobaczyć implementację modeli mieszania procesów Dirichleta w Javie.
Implementacja modelu mieszaniny procesów Dirichleta w Javie
Kod implementuje model Dirichlet Process Mixture Model z Gibbs Sampler i używa Apache Commons Math 3.3 jako biblioteki macierzy. Jest licencjonowany na licencji GPLv3, więc możesz go swobodnie używać, modyfikować i rozpowszechniać swobodnie, a implementację Java możesz pobrać ze strony Github. Zauważ, że możesz znaleźć wszystkie teoretyczne części metody klastrowania w poprzednich 5 artykułach i szczegółowe komentarze Javadoc dotyczące implementacji w kodzie źródłowym.
Poniżej podajemy opis wysokiego poziomu kodu:
1. Klasa DPMM
DPMM jest klasą abstrakcyjną i działa jak baza dla różnych modeli, implementuje Chiński proces restauracyjny i zawiera Zwinięty próbnik Gibbsa. Posiada publiczną metodę cluster(), która odbiera zbiór danych jako Listę Punktów i odpowiada za wykonanie analizy skupień. Inne przydatne metody tej klasy to getPointAssignments(), która służy do pobierania przypisań klastrów po zakończeniu klastrowania, oraz getClusterList(), która służy do pobierania listy zidentyfikowanych klastrów. DPMM zawiera klaster statycznej klasy abstrakcyjnej zagnieżdżonej; zawiera kilka abstrakcyjnych metod dotyczących zarządzania punktami i estymacji a posteriori pdf, które są wykorzystywane do estymacji przypisań do klastrów.
2. Gaussowska klasa DPMM
GaussianDPMM jest implementacją wielowymiarowego modelu normalnej mieszaniny Dirichleta i rozszerza klasę DPMM. Zawiera wszystkie metody, które są wymagane do oszacowania prawdopodobieństw przy założeniu Gaussa. Ponadto zawiera statyczną klasę zagnieżdżoną Cluster, która implementuje wszystkie abstrakcyjne metody klasy DPMM.Cluster.
3. Wielomianowa klasa DPMM
MultinomialDPMM implementuje model Dirichleta-Multinomial Mixture i rozszerza klasę DPMM. Podobnie jak klasa GaussianDPMM zawiera wszystkie metody wymagane do oszacowania prawdopodobieństw przy założeniu Multinomial-Dirichlet oraz zawiera statyczną klasę zagnieżdżoną Cluster, która implementuje abstrakcyjne metody DPMM.Cluster.
4. Klasa SRS
Klasa SRS służy do wykonywania prostego próbkowania losowego z tabeli częstotliwości. Jest używany przez Gibbs Sampler do oszacowania nowych przypisań klastrów na każdym etapie procesu iteracyjnego.
5. Klasa punktowa
Klasa Point służy jako krotka przechowująca dane rekordu wraz z jego identyfikatorem.
6. Biblioteka matematyczna Apache Commons
Biblioteka Apache Commons Math 3.3 służy do mnożenia macierzy i jest jedyną zależnością naszej implementacji.
7. DPMMPrzykładowa klasa
Ta klasa zawiera przykłady użycia implementacji Java.
Korzystanie z implementacji Java
Użytkownik kodu ma możliwość konfiguracji wszystkich parametrów modeli mieszanin, w tym typów modeli i hiperparametrów. W poniższym fragmencie kodu możemy zobaczyć, jak algorytm jest inicjowany i wykonywany:
List<Point> pointList = new ArrayList<>();
//add records in pointList
//Dirichlet Process parameter
Integer dimensionality = 2;
double alpha = 1.0;
//Hyper parameters of Base Function
int kappa0 = 0;
int nu0 = 1;
RealVector mu0 = new ArrayRealVector(new double[]{0.0, 0.0});
RealMatrix psi0 = new BlockRealMatrix(new double[][]{{1.0,0.0},{0.0,1.0}});
//Create a DPMM object
DPMM dpmm = new GaussianDPMM(dimensionality, alpha, kappa0, nu0, mu0, psi0);
int maxIterations = 100;
int performedIterations = dpmm.cluster(pointList, maxIterations);
//get a list with the point ids and their assignments
Map<Integer, Integer> zi = dpmm.getPointAssignments();
Poniżej możemy zobaczyć wyniki działania algorytmu na syntetycznym zbiorze danych składającym się z 300 punktów danych. Punkty zostały pierwotnie wygenerowane przez 3 różne rozkłady: N([10,50], I), N([50,10], I) oraz N([150,100], I).
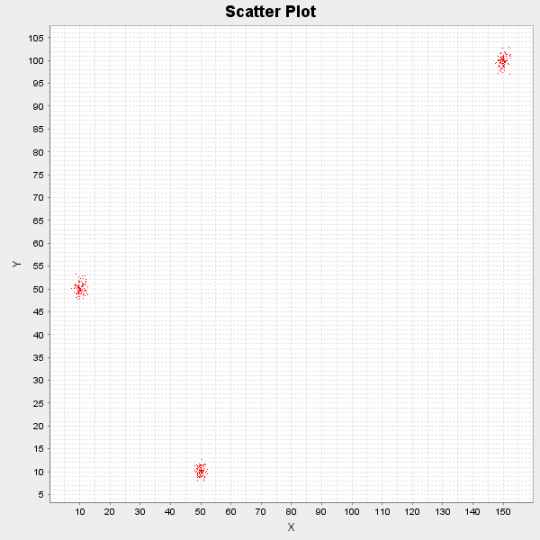
Rysunek 1: Wykres punktowy zestawu danych demonstracyjnych
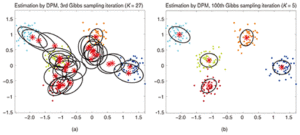
Algorytm po uruchomieniu przez 10 iteracji zidentyfikował następujące 3 centra skupień: [10.17, 50.11], [49.99, 10.13] i [149.97, 99.81]. Wreszcie, ponieważ traktujemy wszystko w sposób bayesowski, jesteśmy w stanie nie tylko dostarczyć jednopunktowe estymacje centrów skupień, ale także ich rozkład prawdopodobieństwa, korzystając z funkcji formuła 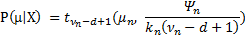 .
.
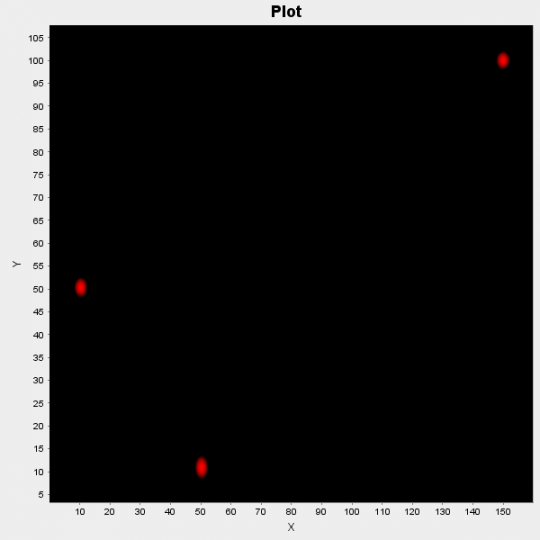
Rysunek 2: Wykres punktowy prawdopodobieństw centrów klastrów
Na powyższym rysunku wykreślamy te prawdopodobieństwa; czerwone obszary wskazują na wysokie prawdopodobieństwo bycia w centrum klastra, a czarne na niskie prawdopodobieństwo.
Aby użyć implementacji Java w rzeczywistych aplikacjach, musisz napisać zewnętrzny kod, który konwertuje oryginalny zbiór danych do wymaganego formatu. Ponadto dodatkowy kod może być konieczny, jeśli chcesz zwizualizować wyjście, jak widać powyżej. Na koniec zwróć uwagę, że do projektu dołączona jest biblioteka Apache Commons Math, a zatem do uruchomienia wersji demonstracyjnych nie jest wymagana żadna dodatkowa konfiguracja.
Jeśli korzystasz z wdrożenia w ciekawym projekcie, napisz do nas, a my przedstawimy Twój projekt na naszym blogu. Również jeśli artykuł Ci się spodobał, poświęć chwilę i udostępnij go na Twitterze lub Facebooku.
- AI
- ai sztuka
- generator sztuki ai
- masz robota
- sztuczna inteligencja
- certyfikacja sztucznej inteligencji
- robot sztucznej inteligencji
- roboty sztucznej inteligencji
- oprogramowanie sztucznej inteligencji
- blockchain
- konferencja blockchain ai
- pomysłowość
- sztuczna inteligencja konwersacyjna
- konferencja kryptograficzna
- Dall's
- Skrzynka odniesienia
- głęboka nauka
- google to
- uczenie maszynowe
- Uczenie maszynowe i statystyki
- plato
- Platon Ai
- Analiza danych Platona
- Gra Platona
- PlatoDane
- platogaming
- Programowanie
- skala ai
- składnia
- zefirnet