Introdução
A descoberta durante o Projeto Genoma Humano, no início dos anos 2000, de que nós, humanos, temos apenas cerca de 20,000 genes codificadores de proteínas – quase o mesmo número do minúsculo verme nematóide que vive no solo e menos da metade do número da planta do arroz – foi um choque. . Esse golpe no nosso orgulho foi amenizado pela ideia de que o genoma humano é rico em conexões regulatórias. Nossos genes interagem em uma rede densa, na qual pedaços de DNA e as moléculas que eles codificam (RNA e proteínas) controlam a “expressão” de outros genes, influenciando se eles produzem seus respectivos RNA e proteínas. Para compreender o genoma humano, precisávamos compreender este processo de regulação genética.
Essa tarefa, porém, está se mostrando muito mais difícil do que decodificar a sequência do genoma.
Inicialmente, suspeitava-se que a regulação genética era uma simples questão de um produto genético agir como um interruptor liga/desliga para outro gene, de forma digital. Na década de 1960, os biólogos franceses François Jacob e Jacques Monod elucidaram pela primeira vez um processo regulador genético em detalhes mecanísticos: Em Escherichia coli Nas bactérias, quando uma proteína repressora se liga a um determinado segmento de DNA, ela bloqueia a transcrição e tradução de um conjunto adjacente de genes que codificam enzimas para digerir o açúcar lactose. Este circuito regulador, que Monod e Jacob apelidaram de laca operon, tem uma lógica clara e transparente.
Mas a regulação genética em metazoários complexos – animais como os humanos, com células eucarióticas complexas – geralmente não parece funcionar desta forma. Em vez disso, envolve um grupo de moléculas, incluindo proteínas, RNAs e pedaços de DNA de um cromossomo, que de alguma forma colaboram para controlar a expressão de um gene.
Não é apenas que esse processo regulatório em eucariontes tenha mais participantes do que normalmente visto em bactérias e outras células procarióticas simples; parece ser um processo categoricamente diferente e mais confuso.
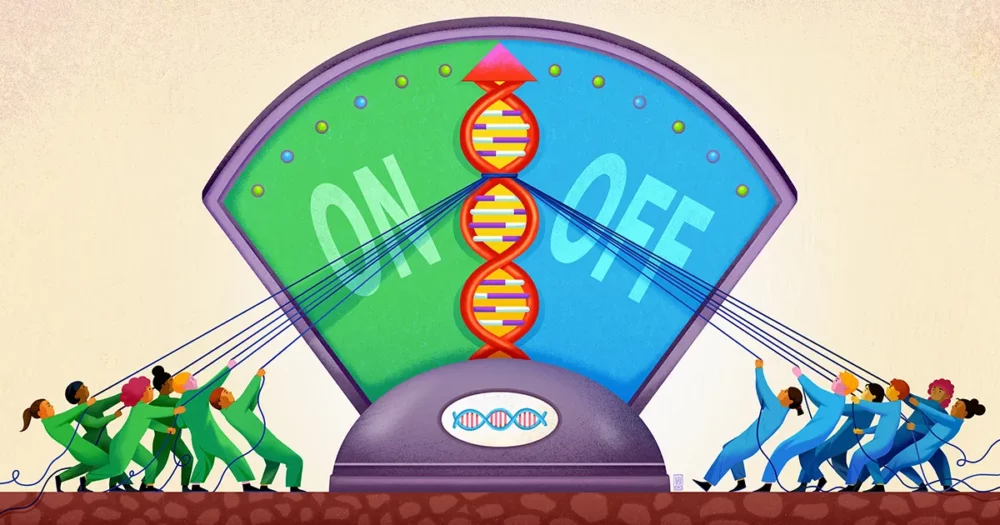
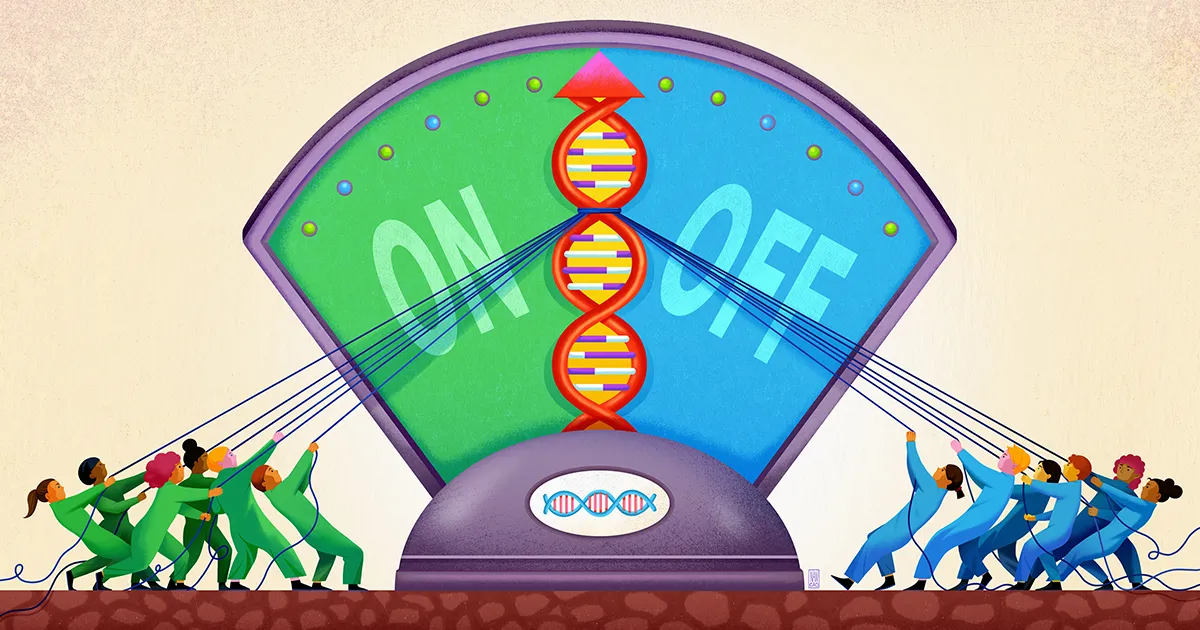
Uma equipe da Universidade de Stanford, liderada pelo biofísico e bioengenheiro Polly Fordyce, parece agora ter descoberto um componente desse modo confuso de regulação genética. Trabalho deles, publicado em setembro passado em Ciência, sugere que o DNA próximo a um gene atua como uma espécie de poço raso para capturar diversas moléculas reguladoras, mantendo-as prontas para a ação para que, quando necessário, possam dar voz à decisão sobre ativar ou não o gene.
Introdução
Esses poços reguladores são feitos de trechos decididamente estranhos de DNA. Eles consistem em sequências nas quais um pequeno trecho de DNA, de um a seis pares de bases, se repete muitas vezes. Dezenas de cópias dessas “repetições curtas em tandem” (STRs) podem ser encadeadas nessas sequências, como a mesma pequena “palavra” escrita repetidamente.
Os STRs são abundantes no genoma humano: compreendem cerca de 5% de todo o nosso DNA. Antigamente, eles eram considerados exemplos clássicos de DNA “lixo”, porque um “texto” repetitivo de DNA composto apenas de STRs não consegue conter tanta informação significativa quanto, digamos, a sequência irregular de letras que compõem uma frase neste texto. artigo.
E, no entanto, os STRs não são claramente insignificantes: têm sido associados a doenças como a doença de Huntington, a atrofia muscular espinobulbar, a doença de Crohn e alguns cancros. Nas últimas décadas, acumularam-se evidências de que eles podem de alguma forma aumentar ou inibir a regulação genética. O mistério era como eles podiam ser tão poderosos com tão pouco conteúdo informativo.
Controles Complexos para Células Complexas
Para entender como os STRs se enquadram no panorama geral da regulação genética, vamos dar um passo atrás. Os genes são rotineiramente flanqueados por pedaços de DNA que não codificam RNA ou proteínas, mas têm funções reguladoras. Os genes bacterianos possuem regiões “promotoras” onde as enzimas polimerases podem se ligar para iniciar a transcrição do DNA adjacente em RNA. Eles também possuem rotineiramente regiões “operadoras”, onde proteínas repressoras podem se ligar para bloquear a transcrição, desligando um gene, como no laca operon.
Em humanos e outros eucariotos, as sequências reguladoras podem ser mais numerosas, variadas – e desconcertantes. Regiões chamadas intensificadores, por exemplo, afetam a probabilidade de um gene ser transcrito. Os intensificadores são frequentemente alvos de proteínas chamadas fatores de transcrição, que podem se ligar para aumentar ou inibir a expressão genética. Estranhamente, alguns intensificadores estão a dezenas de milhares de pares de bases de distância dos genes que regulam e só são aproximados deles através do rearranjo físico das alças de DNA num cromossomo compactado.
A regulação genética eucariótica normalmente envolve esses diversos blocos reguladores de DNA, juntamente com um ou mais fatores de transcrição e outras moléculas, todos reunidos em torno de um gene como um comitê reunido para decidir o que deve fazer. Eles se reúnem em um aglomerado denso e solto.
Muitas vezes, os participantes moleculares também não parecem interagir através dos pares altamente seletivos de “fechadura e chave” comuns na biologia molecular. Em vez disso, são muito menos exigentes, interagindo de forma bastante fraca e não seletiva, como se estivessem vagando e iniciando breves conversas entre si.
Na verdade, a forma como os fatores de transcrição se ligam ao DNA em eucariontes tem sido um mistério. Durante muito tempo presumiu-se que alguma parte de um factor de transcrição devia corresponder estreitamente a uma sequência de “motivo” de ligação no ADN, como as peças de um puzzle. Mas embora alguns desses motivos tenham sido identificados, a sua presença nem sempre se correlaciona muito bem com o local onde os cientistas encontram factores de transcrição aderidos ao ADN nas células. Às vezes, os fatores de transcrição permanecem em regiões sem quaisquer motivos, enquanto alguns motivos que parecem dever ligar-se fortemente aos fatores de transcrição permanecem vazios.
“Tradicionalmente na genómica, o objectivo tem sido classificar os sítios genómicos de uma forma [binária] como 'ligados' ou 'não ligados'” por factores de transcrição, disse Fordyce. “Mas a imagem é muito mais sutil do que isso.” Os membros individuais desses “comités” de regulação genética não parecem estar invariavelmente presentes ou ausentes nas suas reuniões, mas têm diferentes probabilidades de estar presentes ou não.
A tendência da regulação genética em eucariontes de depender de tantas interações fracas diversas entre grandes complexos moleculares “é uma das coisas que torna notoriamente difícil entender teoricamente”, disse o biofísico. Thomas Kuhlman da Universidade da Califórnia, Riverside, que escreveu um comentário no artigo do laboratório Fordyce para Ciência. É um enigma profundo como, deste processo aparentemente caótico, emergem decisões precisas sobre ligar e desligar genes.
Além da misteriosa lógica confusa desse processo de decisão, há também a questão de como todos os membros do comitê encontram o caminho para a sala certa – e então permanecem lá. As moléculas geralmente se movem ao redor da célula por difusão, atingidas por todas as outras moléculas circundantes, como a água, e vagando em direções aleatórias. Poderíamos esperar que estes comités frouxos se separassem demasiado rapidamente para realizarem o seu trabalho regulamentar.
É aí, pensam Fordyce e seus colegas, que entram os STRs. Os STRs são surpreendentemente comuns em locais intensificadores do DNA. No seu artigo, os investigadores argumentam que os STRs actuam como manchas pegajosas que reúnem factores de transcrição e impedem que se desviem.
Ajustando a aderência
O grupo de Fordyce investigou sistematicamente como as diferenças na sequência STR influenciam a adesão dos fatores de transcrição a um motivo de ligação. Eles analisaram dois fatores – um de levedura e outro de humanos – que seguem um motivo específico de seis bases. Os pesquisadores mediram a força (ou afinidade) dessa ligação e a taxa na qual os fatores de transcrição ficam presos e descolados (cinética) quando o motivo é flanqueado por um STR em vez de uma sequência aleatória. Para efeito de comparação, observaram a rapidez com que os factores se ligam apenas ao STR e a uma sequência de ADN totalmente aleatória.
“Um dos maiores desafios deste campo é desemaranhar a miríade de variáveis que impactam a ligação [do fator de transcrição] em uma posição específica do genoma”, disse David Suter, biólogo molecular do Instituto Federal Suíço de Tecnologia de Lausanne, na Suíça. A forma do DNA, a proximidade de outros segmentos de DNA e a tensão física nas moléculas de DNA podem desempenhar um papel na ligação do fator de transcrição. Os valores destes parâmetros provavelmente diferem em todas as posições do genoma, e talvez também entre tipos de células e dentro de uma única célula ao longo do tempo em uma determinada posição. “Este é um vasto espaço de variáveis desconhecidas que são muito difíceis de quantificar”, disse Suter.
Introdução
É por isso que experimentos bem controlados como os da equipe de Stanford são tão úteis, acrescentou Kuhlman. Normalmente, quando os pesquisadores precisam medir interações fracas como essas, eles têm duas opções: podem fazer algumas medições muito detalhadas e extremamente precisas e generalizar a partir delas, ou podem fazer muitas medições rápidas e sujas e usar medidas matematicamente complexas. métodos estatísticos para deduzir resultados. Mas Fordyce e seus colegas, disse Kuhlman, usaram um procedimento automatizado baseado em chip microfluídico para fazer medições precisas durante experimentos de alto rendimento “para obter o melhor dos dois mundos”.
A equipe de Stanford descobriu que diferentes sequências STR podem alterar as afinidades de ligação dos fatores de transcrição ao DNA em até 70 vezes; às vezes eles têm mais impacto na ligação do fator de transcrição do que na alteração da sequência do próprio motivo de ligação. E os efeitos foram diferentes para os dois fatores de transcrição analisados.
Assim, os STRs parecem capazes de ajustar a capacidade dos fatores de transcrição de se acoplarem a um local de DNA e, assim, regularem um gene. Mas como, exatamente?
Uma sala de espera perto de um gene
Os investigadores concluíram que a parte de um factor de transcrição que se liga ao ADN pode interagir fracamente com um STR, com a força exacta dessa afinidade dependendo da sequência do STR. Como essa ligação é fraca, ela não terá muita especificidade. Mas se um fator de transcrição for vagamente captado e liberado por um STR repetidas vezes, o efeito cumulativo é manter o fator de transcrição na vizinhança do gene, de modo que seja mais provável que ele se ligue com segurança à região do motivo, se necessário.
Fordyce e os seus colegas previram que os STRs actuam assim como um “lobby” ou poço onde os factores de transcrição podem reunir-se, ainda que transitoriamente, perto de um local de ligação regulamentar. “A natureza repetitiva de um STR amplifica o efeito fraco de qualquer sítio de ligação único de que é feito”, disse Connor Horton, o primeiro autor do estudo, que agora é estudante de doutorado na Universidade da Califórnia, Berkeley.
Por outro lado, acrescentou ele, alguns STRs também podem agir para afastar os fatores de transcrição das sequências reguladoras, absorvendo os fatores de transcrição em outros lugares como uma esponja. Desta forma, eles podem inibir a expressão genética.
O trabalho, disse Suter, “mostra de forma convincente que os STRs impactam diretamente a ligação dos fatores de transcrição in vitro”. Além do mais, a equipe de Stanford usou um algoritmo de aprendizado de máquina para mostrar que os efeitos observados em seus experimentos in vitro também parecem ocorrer em células vivas (ou seja, in vivo).
BUT Robert Tjian, bioquímico de Berkeley e investigador do Howard Hughes Medical Institute, acha que pode ser muito cedo para ter certeza da influência que uma determinada combinação de fator de transcrição STR tem na expressão genética em células reais.
Tjian, Xavier Darzacq e seus colegas no laboratório que dirigem juntos em Berkeley concordam que os STRs parecem oferecer uma maneira de concentrar os fatores de transcrição perto dos locais reguladores dos genes. No entanto, sem saber quão próximos os fatores precisam estar para ativar a transcrição, é difícil compreender o significado funcional desse resultado. Tjian disse que gostaria de ver se a introdução de um STR em uma célula viva influencia previsivelmente a expressão de um gene alvo. Actualmente, disse ele, “não está convencido de que as COS serão necessariamente um aspecto importante dos mecanismos [regulatórios] in vivo”.
Uma gramática combinatória
Um enigma persistente é como tal mecanismo fornece de forma confiável o tipo de regulação genética precisa que as células necessitam, uma vez que tanto a força quanto a seletividade da ligação do fator de transcrição dentro dos poços STR são fracas. Fordyce acredita que tal especificidade de influência poderia vir de muitas fontes – não apenas de diferenças nas sequências STR, mas também de interações cooperativas entre fatores de transcrição e outras proteínas envolvidas na regulação.
Considerando tudo isso, disse Horton, não está claro se será fácil prever o efeito de uma determinada combinação de fator de transcrição STR na expressão de um gene. A lógica do processo é realmente confusa. E a “gramática” da influência é provavelmente combinatória, acrescentou Horton: O resultado depende de diferentes combinações de fatores de transcrição e outras moléculas.
A equipe de Stanford acredita que talvez 90% dos fatores de transcrição sejam sensíveis aos STRs, mas que existem muito mais tipos de fatores de transcrição no genoma humano do que tipos de STRs. “A mutação de uma sequência STR pode afetar a ligação de 20 fatores de transcrição diferentes nesse tipo de célula, levando a uma diminuição geral na transcrição desse gene próximo, sem implicar qualquer fator de transcrição específico”, disse Horton.
Assim, na verdade, a equipe de Stanford concorda com Tjian que a regulação genética nas células vivas não será impulsionada por um mecanismo único e simples. Em vez disso, os factores de transcrição, os seus locais de ligação ao ADN e outras moléculas reguladoras podem reunir-se em grupos densos que exercem a sua influência colectivamente.
“Existem agora vários exemplos que apoiam a ideia de que os elementos do DNA podem aglomerar os fatores de transcrição a ponto de formarem condensados com cofatores”, disse Richard Young, biólogo celular do Instituto Whitehead do Instituto de Tecnologia de Massachusetts. Os intensificadores ligam muitos fatores de transcrição para produzir esse apinhamento. Os STRs podem ser um ingrediente que ajuda a reunir fatores de transcrição para se agruparem perto de um gene, mas não serão tudo.
Por que regular os genes desta maneira complicada, em vez de confiar no tipo de interações fortes e específicas entre proteínas reguladoras e sítios de DNA que dominam nos procariontes? É possível que tal imprecisão tenha sido o que tornou possíveis metazoários grandes e complexos.
Para serem espécies viáveis, os organismos precisam ser capazes de evoluir e se adaptar às novas circunstâncias. Se nossas células dependessem de alguma rede enorme, mas rigorosamente prescrita, de interações reguladoras genéticas, seria difícil fazer quaisquer alterações nela sem interromper toda a engenhoca, assim como um relógio suíço irá travar se removermos (ou mesmo deslocarmos ligeiramente) qualquer de suas inúmeras engrenagens. Contudo, se as interacções moleculares regulamentares forem frouxas e bastante inespecíficas, existe uma folga útil no sistema - tal como um comité pode geralmente chegar a uma boa decisão mesmo que um dos seus membros esteja doente.
Fordyce observa que em procariontes como bactérias, pode ser relativamente fácil para os fatores de transcrição encontrarem seus locais de ligação porque o genoma a ser pesquisado é menor. Mas isso fica mais difícil à medida que o genoma aumenta. Nos grandes genomas dos eucariontes, “não se pode mais tolerar o risco de ficar temporariamente preso num local de ligação 'errado'”, disse Fordyce, porque isso comprometeria a capacidade de responder rapidamente às mudanças nas condições ambientais.
Além disso, os próprios STRs são altamente evolutivos. Um alongamento ou encurtamento de sua sequência, ou uma alteração no tamanho e profundidade do “poço do fator de transcrição”, pode ocorrer facilmente através de acidentes na replicação ou reparo do DNA, ou através da recombinação sexual dos cromossomos. Para Fordyce, sugere que os STRs “podem, portanto, servir como matéria-prima para a evolução de novos elementos regulatórios e para o ajuste fino dos módulos regulatórios existentes para programas transcricionais sensíveis”, como aqueles que regem o desenvolvimento de animais e plantas.
O poder das interações fracas
Tais considerações estão levando os biólogos moleculares a prestar muito mais atenção às interações fracas e relativamente não seletivas no genoma. Muitas delas envolvem proteínas que, em vez de terem uma estrutura fixa e precisa, são soltas e flexíveis – “intrinsecamente desordenadas”, como dizem os bioquímicos. Se as proteínas funcionassem apenas através de domínios estruturais rígidos, explicou Young, isso restringiria não apenas a evolução dos sistemas reguladores, mas também os tipos de regulação dinâmica observados na vida. “Você não encontrará um organismo vivo – ou mesmo um vírus – funcionando apenas com elementos estruturais estáveis como os de um relógio suíço”, disse Young.
Talvez a evolução tenha apenas tropeçado nos STRs como um componente de uma solução tão complexa, mas em última análise mais eficaz, para a regulação genética em eucariotas. Os próprios STRs podem surgir de várias maneiras – por exemplo, através de erros na replicação do DNA ou da atividade de segmentos de DNA chamados elementos transponíveis que fazem cópias de si mesmos em todo o genoma.
“Acontece que as interações fracas emergentes resultantes entre as proteínas e as sequências repetitivas eram algo que poderia… fornecer vantagem seletiva às células onde ocorriam”, disse Kuhlman. O seu palpite é que esta imprecisão foi provavelmente imposta aos eucariotas, mas que “eles foram posteriormente capazes de explorá-la para seu próprio benefício”. As bactérias e outros procariontes podem contar com uma lógica reguladora “digital” bem definida porque as suas células tendem a existir apenas em alguns estados simples e distintos, como movimentar-se e replicar-se.
Mas os diferentes estados celulares dos metazoários são “muito mais complexos e por vezes próximos de um continuum”, disse Suter, por isso são melhor servidos por uma regulação “analógica” mais difusa.
“Os sistemas reguladores genéticos em bactérias e eucariontes parecem ter divergido substancialmente”, concordou Tjian. Embora se diga que Monod uma vez observou que “o que é verdade para E. coli é verdade para o elefante”, parece que nem sempre é assim.
- Conteúdo com tecnologia de SEO e distribuição de relações públicas. Seja amplificado hoje.
- PlatoData.Network Gerativa Vertical Ai. Capacite-se. Acesse aqui.
- PlatoAiStream. Inteligência Web3. Conhecimento Amplificado. Acesse aqui.
- PlatãoESG. Carbono Tecnologia Limpa, Energia, Ambiente, Solar, Gestão de resíduos. Acesse aqui.
- PlatoHealth. Inteligência em Biotecnologia e Ensaios Clínicos. Acesse aqui.
- Fonte: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- :tem
- :é
- :não
- :onde
- ][p
- $UP
- 000
- 20
- 70
- a
- habilidade
- Capaz
- Sobre
- ausente
- abundante
- Acumulado
- Aja
- atuação
- Açao Social
- ativado
- atividade
- atos
- adaptar
- adicionar
- adicionado
- adjacente
- Vantagem
- afetar
- afinidade
- novamente
- concordaram
- concorda
- algoritmo
- Todos os Produtos
- sozinho
- juntamente
- tb
- Apesar
- sempre
- entre
- amplifica
- an
- e
- animais
- Outro
- qualquer
- à parte
- SOMOS
- argumentar
- surgir
- por aí
- artigo
- AS
- aspecto
- assumiu
- At
- por WhatsApp.
- autor
- Automatizado
- longe
- em caminho duplo
- Bactérias
- base
- BE
- Porque
- tornam-se
- sido
- começar
- ser
- beneficiar
- Berkeley
- MELHOR
- Melhor
- entre
- Grande
- Big Picture
- maior
- O maior
- vincular
- obrigatório
- biologia
- Bloquear
- Blocos
- soprar
- impulsionar
- ambos
- Trazido
- mas a
- by
- Califórnia
- chamado
- veio
- CAN
- célula
- Células
- certo
- desafios
- Alterações
- mudança
- escolhas
- Cromossomos
- circunstâncias
- clássico
- classificar
- remover filtragem
- claramente
- Fechar
- de perto
- Agrupar
- colaborar
- colegas
- coletivamente
- combinação
- combinações
- como
- comitê
- comum
- comparação
- integrações
- complicado
- componente
- compromisso
- condições
- Coneções
- Considerações
- conteúdo
- Continuum
- ao controle
- controles
- conversas
- cooperativo
- poderia
- Casal
- multidão
- décadas
- decidir
- decisão
- decisões
- decodificação
- diminuir
- Dependendo
- depende
- profundidade
- detalhe
- detalhado
- Desenvolvimento
- diferir
- diferenças
- diferente
- difícil
- Distribuição
- digital
- moda digital
- diretamente
- descoberta
- Doença
- distinto
- diferente
- dna
- do
- Dock
- Não faz
- domínios
- Dominar
- não
- dirigido
- apelidado
- durante
- dinâmico
- Cedo
- facilmente
- fácil
- efeito
- Eficaz
- efeitos
- ou
- elementos
- elefante
- em outro lugar
- emergem
- aumentar
- ambiental
- erros
- Mesmo
- Cada
- evidência
- evolução
- evolui
- evolução
- exatamente
- exemplo
- exemplos
- existir
- existente
- esperar
- experimentos
- explicado
- Explorar
- expressão
- extremamente
- fato
- fator
- fatores
- Moda
- Federal
- poucos
- campo
- figurado
- Encontre
- Primeiro nome
- caber
- fixado
- Escolha
- forçado
- formulário
- encontrado
- Francês
- da
- funcional
- funcionamento
- funções
- Gangue
- reunir
- coleta
- reuniões
- geralmente
- genómica
- ter
- fica
- dado
- meta
- vai
- Bom estado, com sinais de uso
- governando
- ótimo
- Grupo
- adivinhar
- Metade
- manipular
- aconteceu
- Queijos duros
- mais duro
- Ter
- ter
- he
- ajuda
- sua experiência
- altamente
- sua
- segurar
- Como funciona o dobrador de carta de canal
- Contudo
- HTML
- HTTPS
- enorme
- humano
- Humanos
- idéia
- identificado
- if
- Impacto
- in
- Incluindo
- de fato
- Individual
- influência
- influenciando
- INFORMAÇÕES
- insignificante
- em vez disso
- Instituto
- interagir
- interagindo
- interações
- para dentro
- introduzindo
- invariavelmente
- envolver
- envolvido
- envolve
- IT
- ESTÁ
- se
- jacob
- serra de vaivém
- Trabalho
- apenas por
- Guarda
- manutenção
- Tipo
- tipos
- Conhecimento
- laboratório
- grande
- Sobrenome
- principal
- aprendizagem
- levou
- menos
- vida
- como
- Provável
- ligado
- pequeno
- vida
- lógica
- longo
- mais
- olhou
- máquina
- aprendizado de máquina
- moldadas
- revista
- principal
- fazer
- FAZ
- maneira
- muitos
- massachusetts
- Instituto de Tecnologia de Massachusetts
- Match
- material
- matematicamente
- Importância
- Posso..
- talvez
- significativo
- a medida
- medições
- mecanismo
- mecanismos
- médico
- reuniões
- Membros
- métodos
- poder
- MIT
- Moda
- Módulos
- molecular
- molécula
- mais
- mover
- em movimento
- muito
- múltiplo
- devo
- miríade
- misterioso
- Mistério
- Natureza
- Perto
- quase
- necessariamente
- você merece...
- necessário
- rede
- não
- Notas
- agora
- matizado
- numeroso
- ocorrer
- ocorreu
- ocorrendo
- of
- WOW!
- oferecer
- frequentemente
- on
- uma vez
- ONE
- só
- or
- Outros
- A Nossa
- Fora
- Resultado
- Acima de
- global
- próprio
- embalado
- pares
- pares
- Papel
- parâmetros
- parte
- participantes
- particular
- passado
- Patches
- Pagar
- possivelmente
- persuadido
- físico
- fotografia
- peças
- Plants
- platão
- Inteligência de Dados Platão
- PlatãoData
- Jogar
- players
- ponto
- posição
- possível
- poder
- poderoso
- preciso
- predizer
- previsto
- presença
- presente
- orgulho
- provavelmente
- procedimentos
- processo
- produzir
- Produto
- profundo
- Programas
- projeto
- Proteína
- Proteínas
- fornecer
- fornece
- provando
- publicado
- colocar
- quebra-cabeça
- questão
- rapidamente
- bastante
- acaso
- Taxa
- em vez
- Cru
- prontamente
- pronto
- reais
- região
- regiões
- Regular
- Regulamento
- reguladores
- relativamente
- liberado
- depender
- contando
- permanecem
- comentou
- remover
- reparar
- repetitivo
- réplica
- pesquisadores
- aqueles
- Responder
- resultar
- resultando
- Resultados
- arroz
- Rico
- certo
- rígido
- Risco
- Ribeira
- RNA
- Tipo
- Quarto
- rotineiramente
- Execute
- Dito
- mesmo
- dizer
- cientistas
- firmemente
- Vejo
- parecem
- aparentemente
- parece
- visto
- segmento
- segmentos
- Aproveitar
- seletivo
- sensível
- sentença
- Setembro
- Seqüência
- servir
- servido
- vários
- Sexual
- raso
- Shape
- Baixo
- rede de apoio social
- mostrar
- significado
- simples
- desde
- solteiro
- local
- Locais
- SIX
- Tamanho
- folga
- menor
- So
- solução
- alguns
- de alguma forma
- algo
- às vezes
- Fontes
- Espaço
- específico
- especificidade
- estável
- Stanford
- Universidade de Stanford
- Unidos
- estatístico
- ficar
- Passo
- aderindo
- pegajoso
- Dê um basta
- História
- franco
- força
- mais forte,
- discordaram
- estrutural
- estrutura
- estudante
- Estudo
- Subseqüentemente
- substancialmente
- tal
- açúcar
- Sugere
- suíte
- ajuda
- certo
- Em torno da
- suíço
- Interruptor
- Suíça
- .
- sistemas
- Tire
- Tandem
- Target
- tem como alvo
- Tarefa
- Profissionais
- Tecnologia
- conta
- Tender
- dezenas
- do que
- que
- A
- deles
- Eles
- si mesmos
- então
- Lá.
- assim sendo
- Este
- deles
- coisas
- think
- Acha
- isto
- aqueles
- Apesar?
- pensamento
- milhares
- Através da
- todo
- Assim
- hermeticamente
- tempo
- vezes
- para
- juntos
- também
- Tradução
- transparente
- prendendo
- verdadeiro
- Passando
- dois
- tipo
- tipos
- tipicamente
- Reino Unido
- Em última análise
- descoberto
- compreender
- universidade
- Universidade da Califórnia
- desconhecido
- sobre
- usar
- usava
- útil
- geralmente
- Valores
- vário
- Grande
- muito
- viável
- vírus
- voz
- Esperando
- foi
- Assistir
- Água
- Caminho..
- maneiras
- we
- fraco
- webp
- BEM
- bem definido
- Wells
- foram
- O Quê
- quando
- se
- qual
- enquanto
- QUEM
- inteiro
- totalmente
- porque
- precisarão
- de
- dentro
- sem
- Atividades:
- trabalhou
- do mundo
- verme
- seria
- escrito
- escreveu
- ainda
- Vocês
- jovem
- zefirnet