Введение
Открытие, сделанное в ходе проекта «Геном человека» в начале 2000-х годов, что у нас, людей, есть только около 20,000 XNUMX генов, кодирующих белок — примерно столько же, сколько у крошечного почвенного червя-нематоды, и менее половины от количества у растения риса — стало шоком. . Однако этот удар по нашей гордости был смягчен идеей о том, что геном человека богат регуляторными связями. Наши гены взаимодействуют в плотной сети, в которой фрагменты ДНК и молекулы, которые они кодируют (РНК и белки), контролируют «экспрессию» других генов, влияя на то, создают ли они соответствующие РНК и белки. Чтобы понять геном человека, нам нужно было понять этот процесс регуляции генов.
Однако эта задача оказывается намного сложнее, чем расшифровка последовательности генома.
Первоначально предполагалось, что регуляция генов представляет собой простой процесс, когда один генный продукт действует как переключатель включения/выключения другого гена цифровым способом. В 1960-х годах французские биологи Франсуа Жакоб и Жак Моно впервые выяснили. процесс регуляции генов в механистических деталях: В Кишечная палочка У бактерий, когда белок-репрессор связывается с определенным сегментом ДНК, он блокирует транскрипцию и трансляцию соседнего набора генов, которые кодируют ферменты для переваривания сахарной лактозы. Этот регуляторный контур, который Моно и Джейкоб назвали лак оперон имеет четкую и прозрачную логику.
Но регуляция генов у сложных многоклеточных животных — животных, подобных человеку, со сложными эукариотическими клетками — обычно не работает таким образом. Вместо этого в нем участвует группа молекул, включая белки, РНК и фрагменты ДНК со всей хромосомы, которые каким-то образом взаимодействуют, контролируя экспрессию гена.
Дело не только в том, что в этом регуляторном процессе у эукариот задействовано больше участников, чем обычно наблюдается у бактерий и других простых прокариотических клеток; кажется, это совершенно другой процесс, и более туманный.
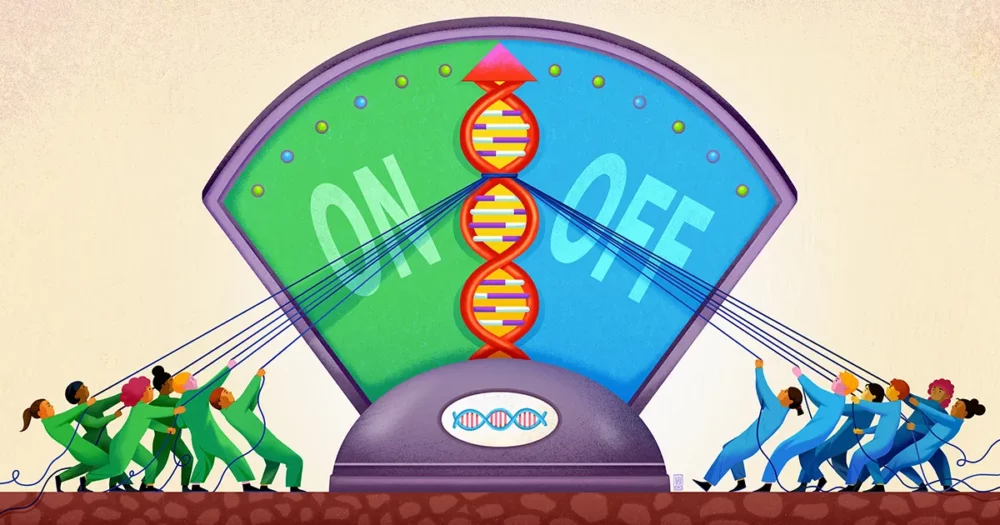
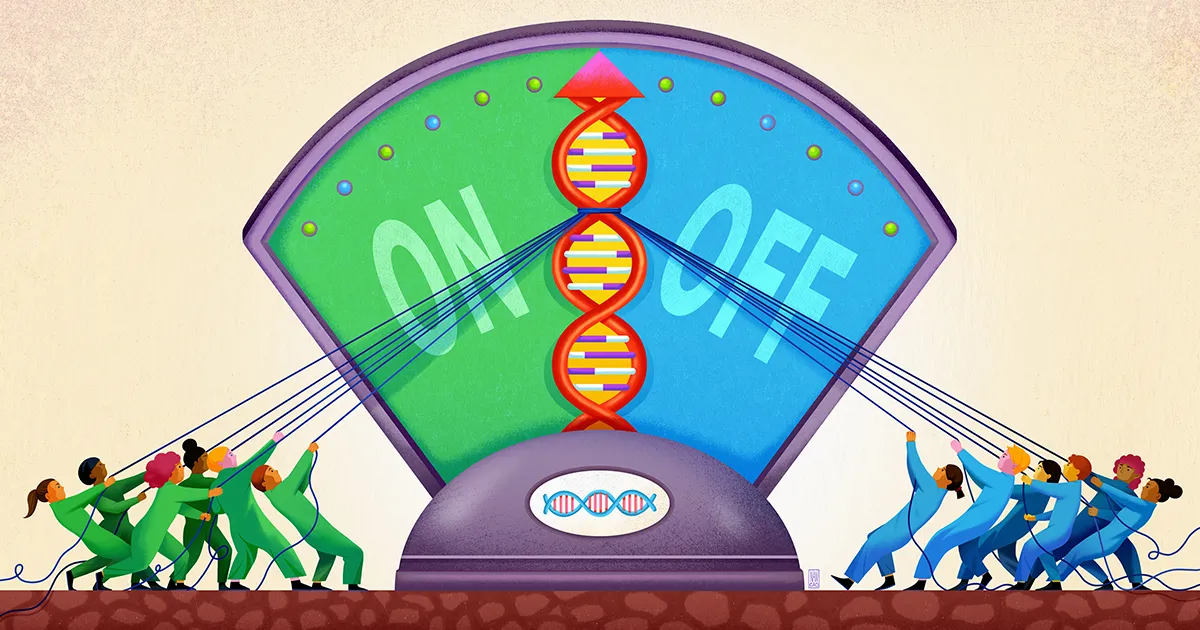
Команда Стэнфордского университета под руководством биофизика и биоинженера Полли Фордайс, похоже, теперь открыли компонент этого нечеткого способа регуляции генов. Их работа, опубликованный в сентябре прошлого года в Наука, предполагает, что ДНК рядом с геном действует как своего рода неглубокий колодец для улавливания различных регуляторных молекул, поддерживая их готовность к действию, чтобы при необходимости они могли добавить свой голос к решению о том, активировать ли ген.
Введение
Эти регуляторные лунки состоят из совершенно необычных участков ДНК. Они состоят из последовательностей, в которых короткий участок ДНК длиной от одной до шести пар оснований повторяется много раз. Десятки копий этих «коротких тандемных повторов» (STR) могут быть связаны в эти последовательности, как одно и то же маленькое «слово», написанное снова и снова.
STR широко распространены в геноме человека: они составляют около 5% всей нашей ДНК. Когда-то их считали классическими примерами «мусорной» ДНК, потому что повторяющийся «текст» ДНК, состоящий только из STR, не может содержать почти столько же значимой информации, как, скажем, нерегулярная последовательность букв, составляющих предложение в этом случае. статья.
И все же STR явно не являются незначительными: они связаны с такими заболеваниями, как болезнь Хантингтона, спинобульбарная мышечная атрофия, болезнь Крона и некоторые виды рака. За последние пару десятилетий накопились доказательства того, что они могут каким-то образом усиливать или подавлять регуляцию генов. Загадка заключалась в том, как они могли быть такими могущественными, имея так мало информации.
Комплексное управление сложными ячейками
Чтобы понять, как STR вписываются в общую картину регуляции генов, давайте сделаем шаг назад. Гены обычно окружены участками ДНК, которые не кодируют РНК или белок, но выполняют регуляторные функции. Бактериальные гены имеют «промоторные» области, с которыми могут связываться ферменты полимеразы, чтобы начать транскрипцию соседней ДНК в РНК. У них также обычно есть «операторные» области, где белки-репрессоры могут связываться и блокировать транскрипцию, отключая ген, как в лак оперон.
У людей и других эукариот регуляторные последовательности могут быть более многочисленными, разнообразными и запутанными. Например, регионы, называемые энхансерами, влияют на вероятность транскрипции гена. Энхансеры часто являются мишенью для белков, называемых факторами транскрипции, которые могут связываться, усиливая или ингибируя экспрессию генов. Как ни странно, некоторые энхансеры находятся на расстоянии десятков тысяч пар оснований от генов, которые они регулируют, и приближаются к ним только в результате физической перестройки петель ДНК в упакованной хромосоме.
Регуляция генов эукариот обычно включает в себя множество разнообразных регуляторных блоков ДНК, а также один или несколько факторов транскрипции и других молекул, которые собираются вокруг гена, как комитет, созванный для решения того, что ему следует делать. Они собираются в рыхлую, плотную группу.
Зачастую молекулярные участники не взаимодействуют посредством высокоселективных пар «замок и ключ», обычных в молекулярной биологии. Вместо этого они гораздо менее разборчивы, взаимодействуют довольно слабо и неизбирательно, как бы бродят вокруг и заводят друг с другом короткие разговоры.
Фактически, то, как факторы транскрипции связываются с ДНК у эукариот, остается загадкой. Долгое время считалось, что некоторая часть транскрипционного фактора должна точно соответствовать связывающей последовательности «мотива» в ДНК, подобно кусочкам головоломки. Но хотя некоторые такие мотивы были идентифицированы, их присутствие не всегда хорошо коррелирует с тем, где ученые находят факторы транскрипции, прикрепляющиеся к ДНК в клетках. Иногда факторы транскрипции задерживаются в регионах без каких-либо мотивов, в то время как некоторые мотивы, которые, как кажется, должны прочно связывать факторы транскрипции, остаются пустыми.
«Традиционно в геномике целью было классифицировать геномные сайты [бинарным] способом как «связанные» или «несвязанные» по факторам транскрипции», — сказал Фордайс. «Но картина гораздо более тонкая». Отдельные члены этих «комитетов» по регулированию генов, похоже, не всегда присутствуют или отсутствуют на их заседаниях, а скорее имеют разные вероятности присутствия или отсутствия.
Тенденция регуляции генов у эукариот полагаться на такое большое количество разнообразных слабых взаимодействий между большими молекулярными комплексами «является одной из вещей, из-за которой чрезвычайно сложно разобраться теоретически», — сказал биофизик. Томас Кульман из Калифорнийского университета в Риверсайде, написавший комментарий на бумаге лаборатории Фордайса для Наука. Это глубокая загадка, как из этого, казалось бы, хаотического процесса возникают точные решения о включении и выключении генов.
Помимо загадочной нечеткой логики этого процесса принятия решений, существует также вопрос о том, как все члены комитета вообще находят дорогу в нужную комнату и затем остаются там. Молекулы обычно движутся вокруг клетки за счет диффузии, подвергаясь воздействию всех других окружающих молекул, таких как вода, и блуждая в случайных направлениях. Мы могли бы ожидать, что эти разрозненные комитеты слишком быстро разойдутся и не смогут выполнять свою регулирующую работу.
Именно здесь, по мнению Фордайс и ее коллег, и появляются STR. STR поразительно распространены в энхансерных участках ДНК. В своей статье исследователи утверждают, что STR действуют как липкие пятна, которые собирают факторы транскрипции и не дают им сбиться с пути.
Точная настройка липкости
Группа Фордайса систематически исследовала, как различия в последовательности STR влияют на прикрепление факторов транскрипции к мотиву связывания. Они рассмотрели два фактора — один из дрожжей, другой из людей — которые придерживаются определенного шестиосновного мотива. Исследователи измерили как силу (или аффинность) этого связывания, так и скорость, с которой факторы транскрипции застревают и открепляются (кинетика), когда мотив окружен STR, а не случайной последовательностью. Для сравнения они изучили, насколько легко факторы связываются только с STR и с совершенно случайной последовательностью ДНК.
«Одна из самых больших проблем в этой области — распутать множество переменных, которые влияют на связывание [фактора транскрипции] в определенном положении генома», — сказал он. Дэвид Сутер, молекулярный биолог из Швейцарского федерального технологического института в Лозанне, Швейцария. Форма ДНК, близость к другим сегментам ДНК и физическое напряжение в молекулах ДНК — все это может играть роль в связывании транскрипционных факторов. Значения этих параметров, вероятно, различаются в каждой позиции генома, а также, возможно, между типами клеток и внутри одной клетки с течением времени в данной позиции. «Это огромное пространство неизвестных переменных, которые очень сложно измерить количественно», — сказал Сутер.
Введение
Вот почему так полезны хорошо контролируемые эксперименты, подобные экспериментам Стэнфордской команды, добавил Кульман. Обычно, когда исследователям необходимо измерить подобные слабые взаимодействия, у них есть два варианта: они могут провести несколько очень подробных, чрезвычайно точных измерений и обобщить их, или они могут провести множество быстрых и грязных измерений и использовать математически сложные измерения. статистические методы для получения результатов. Но Фордайс и ее коллеги, по словам Кульмана, использовали автоматизированную процедуру на основе микрофлюидных чипов для проведения точных измерений во время высокопроизводительных экспериментов, «чтобы получить лучшее от обоих миров».
Команда из Стэнфорда обнаружила, что различные последовательности STR могут изменять аффинность связывания транскрипционных факторов с ДНК в 70 раз; иногда они оказывают большее влияние на связывание транскрипционных факторов, чем на изменение последовательности самого мотива связывания. И эффекты были разными для двух разных факторов транскрипции, которые они изучали.
Таким образом, STR, по-видимому, способны точно настраивать способность факторов транскрипции стыковаться с участком ДНК и, таким образом, регулировать ген. Но как именно?
Зал ожидания возле гена
Исследователи предположили, что часть транскрипционного фактора, который связывает ДНК, может слабо взаимодействовать с STR, причем точная сила этого сродства зависит от последовательности STR. Поскольку такая привязка слаба, она не будет иметь особой специфичности. Но если транскрипционный фактор свободно захватывается и высвобождается STR снова и снова, кумулятивный эффект заключается в том, что транскрипционный фактор удерживается вблизи гена, чтобы он с большей вероятностью надежно связывался с областью мотива, если это необходимо.
Фордайс и ее коллеги предсказали, что STRs, таким образом, действуют как «лобби» или колодец, где факторы транскрипции могут собираться, пусть и временно, вблизи сайта связывания регулятора. «Повторяющаяся природа STR усиливает слабый эффект любого единственного сайта связывания, из которого он состоит», — сказал он. Коннор Хортон, первого автора исследования, который сейчас является аспирантом Калифорнийского университета в Беркли.
И наоборот, добавил он, некоторые STR могут также оттягивать факторы транскрипции от регуляторных последовательностей, впитывая факторы транскрипции в другие места, как губка. Таким образом, они могут ингибировать экспрессию генов.
По словам Сатера, эта работа «убедительно показывает, что STR напрямую влияют на связывание факторов транскрипции in vitro». Более того, команда из Стэнфорда использовала алгоритм машинного обучения, чтобы показать, что эффекты, наблюдаемые в их экспериментах in vitro, похоже, происходят и в живых клетках (то есть in vivo).
Но Роберт Тиан, биохимик из Беркли и исследователь из Медицинского института Говарда Хьюза, считает, что, возможно, еще слишком рано говорить наверняка, какое влияние оказывает данная комбинация STR-транскрипционных факторов на экспрессию генов в реальных клетках.
Тцзян, Ксавье Дарзак и их коллеги из лаборатории в Беркли, которую они вместе возглавляют, согласны с тем, что STR, по-видимому, предлагают способ концентрации факторов транскрипции вблизи регуляторных участков генов. Однако, не зная, насколько близкими должны быть факторы для активации транскрипции, трудно понять функциональное значение этого результата. Тиан сказал, что ему хотелось бы посмотреть, повлияет ли введение STR в живую клетку предсказуемым образом на экспрессию целевого гена. В настоящее время, по его словам, он «не убежден в том, что СПО обязательно станут важным аспектом [регуляторных] механизмов in vivo».
Комбинаторная грамматика
Одна из загадок заключается в том, как такой механизм надежно обеспечивает тот тип точной регуляции генов, который необходим клеткам, поскольку как сила, так и селективность связывания транскрипционных факторов в лунках STR слабы. Фордайс считает, что такая специфичность влияния может исходить из многих источников — не только из-за различий в последовательностях STR, но также из-за кооперативных взаимодействий между факторами транскрипции и другими белками, участвующими в регуляции.
Учитывая все это, сказал Хортон, неясно, будет ли легко предсказать влияние конкретной комбинации STR-транскрипционных факторов на экспрессию гена. Логика процесса действительно нечеткая. И «грамматика» влияния, вероятно, является комбинаторной, добавил Хортон: «Результат зависит от различных комбинаций факторов транскрипции и других молекул».
Команда Стэнфорда считает, что, возможно, 90% факторов транскрипции чувствительны к STR, но в геноме человека гораздо больше типов транскрипционных факторов, чем типов STR. «Мутация последовательности STR может повлиять на связывание 20 различных факторов транскрипции в этом типе клеток, что приведет к общему снижению транскрипции близлежащего гена без вовлечения какого-либо конкретного фактора транскрипции», — сказал Хортон.
Таким образом, команда из Стэнфорда согласна с Тианом в том, что регуляция генов в живых клетках не будет управляться одним простым механизмом. Скорее, транскрипционные факторы, их сайты связывания с ДНК и другие регуляторные молекулы могут собираться в плотные группы, которые коллективно оказывают свое влияние.
«В настоящее время существует множество примеров, подтверждающих идею о том, что элементы ДНК могут сгущать факторы транскрипции до такой степени, что они образуют конденсаты с кофакторами», — сказал он. Ричард Янг, клеточный биолог из Института Уайтхеда Массачусетского технологического института. Энхансеры связывают многие факторы транскрипции, создавая такую скученность. STR могут быть ингредиентом, который помогает факторам транскрипции группироваться возле гена, но это еще не все.
Зачем регулировать гены таким сложным способом, вместо того чтобы полагаться на сильные и специфические взаимодействия между регуляторными белками и участками ДНК, которые доминируют у прокариот? Возможно, именно такая нечеткость вообще сделала возможным появление крупных сложных многоклеточных животных.
Чтобы быть жизнеспособными видами, организмы должны иметь возможность развиваться и адаптироваться к меняющимся обстоятельствам. Если бы наши клетки полагались на некую огромную, но строго предписанную сеть регуляторных взаимодействий генов, было бы трудно внести в нее какие-либо изменения, не разрушив всю эту штуковину, точно так же, как швейцарские часы заклинят, если мы удалим (или даже слегка сместим) любую из его бесчисленных зубчатых колес. Однако если регуляторные молекулярные взаимодействия неопределенны и довольно неспецифичны, в системе есть полезная слабость — точно так же, как комитет обычно может прийти к правильному решению, даже если один из его членов заболел.
Фордайс отмечает, что у прокариот, таких как бактерии, факторам транскрипции может быть относительно легко найти свои сайты связывания, поскольку геном, который нужно искать, меньше. Но это становится сложнее по мере увеличения генома. В больших геномах эукариот «вы больше не можете терпеть риск того, что вы временно застрянете в «неправильном» месте связывания», — сказал Фордайс, потому что это поставит под угрозу способность быстро реагировать на изменяющиеся условия окружающей среды.
Более того, сами СПО легко развиваются. Удлинение или укорочение их последовательности, а также изменение размера и глубины «ямы фактора транскрипции» могут легко произойти в результате сбоев в репликации или репарации ДНК или в результате половой рекомбинации хромосом. По мнению Фордайса, это предполагает, что STR «могут служить сырьем для разработки новых регуляторных элементов и тонкой настройки существующих регуляторных модулей для чувствительных программ транскрипции», например, тех, которые управляют развитием животных и растений.
Сила слабых взаимодействий
Подобные соображения заставляют молекулярных биологов уделять гораздо больше внимания слабым и относительно неизбирательным взаимодействиям в геноме. Многие из них включают белки, которые вместо того, чтобы иметь фиксированную и точную структуру, являются рыхлыми и гибкими — «внутренне неупорядоченными», как выражаются биохимики. Если бы белки действовали только через жесткие структурные домены, объяснил Янг, это ограничивало бы не только развитие регуляторных систем, но и виды динамической регуляции, наблюдаемые в жизни. «Вы не найдете ни живого организма, ни даже вируса, функционирующего только со стабильными структурными элементами, как в швейцарских часах», — сказал Янг.
Возможно, эволюция просто наткнулась на STR как на компонент такого сложного, но в конечном итоге более эффективного решения регуляции генов у эукариот. Сами STR могут возникать несколькими способами — например, из-за ошибок репликации ДНК или активности сегментов ДНК, называемых мобильными элементами, которые копируют сами себя по всему геному.
«Так уж получилось, что возникающие в результате возникающие слабые взаимодействия между белками и повторяющимися последовательностями могли… обеспечить селективное преимущество клеткам, в которых это произошло», — сказал Кульман. Он предполагает, что эта нечеткость, вероятно, была навязана эукариотам, но «впоследствии они смогли использовать [ее] для своей выгоды». Бактерии и другие прокариоты могут полагаться на четко определенную «цифровую» регуляторную логику, поскольку их клетки, как правило, существуют только в нескольких простых, различных состояниях, таких как перемещение и репликация.
Но различные состояния клеток у многоклеточных животных «гораздо более сложны и иногда близки к континууму», сказал Сатер, поэтому им лучше обслуживать более нечеткую «аналоговую» регуляцию.
«Системы регуляции генов у бактерий и эукариот, похоже, действительно существенно разошлись», — согласился Тиан. Хотя Моно, как говорят, однажды заметил, что «то, что верно для E. палочки верно для слона», похоже, это не всегда так.
- SEO-контент и PR-распределение. Получите усиление сегодня.
- PlatoData.Network Вертикальный генеративный ИИ. Расширьте возможности себя. Доступ здесь.
- ПлатонАйСтрим. Интеллект Web3. Расширение знаний. Доступ здесь.
- ПлатонЭСГ. Углерод, чистые технологии, Энергия, Окружающая среда, Солнечная, Управление отходами. Доступ здесь.
- ПлатонЗдоровье. Биотехнологии и клинические исследования. Доступ здесь.
- Источник: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- :имеет
- :является
- :нет
- :куда
- ][п
- $UP
- 000
- 20
- 70
- a
- способность
- в состоянии
- О нас
- отсутствующий
- обильный
- накопленный
- Действие (Act):
- действующий
- Действие
- активировать
- деятельность
- акты
- приспосабливать
- Добавить
- добавленный
- примыкающий
- плюс
- влиять на
- сродство
- снова
- решено
- соглашается с тем,
- алгоритм
- Все
- в одиночестве
- вдоль
- причислены
- Несмотря на то, что
- всегда
- среди
- усиливается
- an
- и
- животные
- Другой
- любой
- кроме
- МЫ
- спорить
- возникать
- около
- гайд
- AS
- внешний вид
- предполагается,
- At
- внимание
- автор
- Автоматизированный
- прочь
- назад
- Бактерии
- Использование темпера с изогнутым основанием
- BE
- , так как:
- становиться
- было
- начинать
- не являетесь
- польза
- Беркли
- ЛУЧШЕЕ
- Лучшая
- между
- большой
- Большая фотография
- больший
- Крупнейшая
- связывать
- переплет
- биология
- Заблокировать
- Блоки
- взрывать
- повышение
- изоферменты печени
- принес
- но
- by
- Калифорния
- под названием
- пришел
- CAN
- ячейка
- Клетки
- определенный
- проблемы
- изменения
- изменения
- выбор
- Хромосомы
- обстоятельства
- классический
- классифицировать
- Очистить
- явно
- Закрыть
- тесно
- Кластер
- сотрудничать
- коллеги
- коллективно
- сочетание
- комбинации
- как
- комитет
- Общий
- сравнение
- комплекс
- сложный
- компонент
- скомпрометированы
- Условия
- Коммутация
- соображения
- содержание
- Континуум
- контроль
- контрольная
- Беседы
- кооперативный
- может
- Пара
- толпа
- десятилетия
- решать
- решение
- решения
- Декодирование
- снижение
- в зависимости
- зависит
- глубина
- подробность
- подробный
- Развитие
- отличаться
- Различия
- различный
- трудный
- Вещание
- Интернет
- цифровая мода
- непосредственно
- открытие
- Болезнь
- отчетливый
- Разное
- Г-жа
- do
- Dock
- не
- доменов
- Господствовать
- Dont
- управляемый
- дублированный
- в течение
- динамический
- Рано
- легко
- легко
- эффект
- Эффективный
- эффекты
- или
- элементы
- слон
- в другом месте
- появляться
- повышать
- окружающий
- ошибки
- Даже
- Каждая
- , поскольку большинство сенаторов
- эволюция
- развивается
- развивается
- точно,
- пример
- Примеры
- существовать
- существующий
- ожидать
- Эксперименты
- объяснены
- Эксплуатировать
- выражение
- чрезвычайно
- факт
- фактор
- факторы
- Фэшн
- Федеральный
- несколько
- поле
- фигурный
- Найдите
- First
- соответствовать
- фиксированной
- Что касается
- принудительный
- форма
- найденный
- Французский
- от
- функциональная
- функционирование
- Функции
- шайка
- собирать
- сбор
- сходы
- в общем
- геномика
- получить
- получает
- данный
- цель
- будет
- хорошо
- руководящий
- большой
- группы
- догадываться
- Половина
- обрабатывать
- произошло
- Жесткий
- Сильнее
- Есть
- имеющий
- he
- помогает
- ее
- очень
- его
- держать
- Как
- Однако
- HTML
- HTTPS
- огромный
- человек
- Людей
- идея
- идентифицированный
- if
- Влияние
- in
- В том числе
- действительно
- individual
- повлиять
- влияющий
- информация
- незначительный
- вместо
- Институт
- взаимодействовать
- взаимодействующий
- взаимодействие
- в
- введение
- неизменно
- включать в себя
- вовлеченный
- включает в себя
- IT
- ЕГО
- саму трезвость
- Джекоб
- Головоломка
- работа
- всего
- Сохранить
- хранение
- Вид
- виды
- знание
- лаборатория
- большой
- Фамилия
- ведущий
- изучение
- привело
- Меньше
- ЖИЗНЬЮ
- такое как
- Вероятно
- связанный
- мало
- жизнью
- логика
- Длинное
- дольше
- смотрел
- машина
- обучение с помощью машины
- сделанный
- журнал
- основной
- сделать
- ДЕЛАЕТ
- способ
- многих
- Массачусетс
- Массачусетский Технологический Институт
- Совпадение
- материала
- математически
- Вопрос
- Май..
- может быть
- значимым
- проводить измерение
- размеры
- механизм
- механизмы
- основным медицинским
- заседаниях
- Участники
- методы
- может быть
- MIT
- режим
- Модули
- молекулярный
- молекула
- БОЛЕЕ
- двигаться
- перемещение
- много
- с разными
- должен
- мириады
- таинственный
- Тайна
- природа
- Возле
- почти
- обязательно
- Необходимость
- необходимый
- сеть
- нет
- Заметки
- сейчас
- нюансы
- многочисленный
- происходить
- произошло
- происходящий
- of
- от
- предлагают
- .
- on
- консолидировать
- ONE
- только
- or
- Другое
- наши
- внешний
- Результат
- за
- общий
- собственный
- упакованный
- спариваний
- пар
- бумага & картон
- параметры
- часть
- новыми участниками
- особый
- мимо
- Патчи
- ОПЛАТИТЬ
- возможно
- убежденный
- физический
- картина
- штук
- растения
- Платон
- Платон Интеллектуальные данные
- ПлатонДанные
- Играть
- игроки
- Точка
- должность
- возможное
- мощностью
- мощный
- необходимость
- предсказывать
- предсказанный
- присутствие
- представить
- гордость
- вероятно
- процедуры
- процесс
- производит
- Продукт
- глубокий
- Программы
- Проект
- Белкове продукты
- Белки
- обеспечивать
- приводит
- доказывания
- опубликованный
- положил
- головоломка
- вопрос
- быстро
- вполне
- случайный
- Обменный курс
- скорее
- Сырье
- легко
- готовый
- реальные
- область
- районы
- регламентировать
- "Регулирование"
- регуляторы
- относительно
- выпустил
- полагаться
- опираясь
- оставаться
- заметил
- удаление
- ремонт
- повторяющийся
- копирование
- исследователи
- те
- Реагируйте
- результат
- в результате
- Итоги
- Рис
- Богатые
- правую
- жесткий
- Снижение
- Прибрежный
- РНК
- Роли
- Комната
- обычно
- Run
- Сказал
- то же
- сообщили
- Ученые
- безопасно
- посмотреть
- казаться
- по-видимому
- кажется
- видел
- сегмент
- сегментами
- Воспользоваться
- селективный
- чувствительный
- предложение
- сентябрь
- Последовательность
- служить
- служил
- несколько
- Сексуальный
- мелкий
- Форма
- Короткое
- должен
- показывать
- значение
- просто
- с
- одинарной
- сайте
- Сайтов
- ШЕСТЬ
- Размер
- слабина
- меньше
- So
- Решение
- некоторые
- как-то
- удалось
- иногда
- Источники
- Space
- конкретный
- специфичность
- стабильный
- Стэнфорд
- Стэнфордский университет
- Области
- статистический
- оставаться
- Шаг
- прилипание
- липкий
- Stop
- История
- простой
- прочность
- сильный
- сильно
- структурный
- Структура
- "Студент"
- Кабинет
- впоследствии
- по существу
- такие
- сахар
- Предлагает
- suite
- поддержка
- Убедитесь
- окружающих
- швейцарский
- Коммутатор
- Швейцария
- система
- системы
- взять
- Тандем
- цель
- направлена против
- Сложность задачи
- команда
- Технологии
- говорит
- Тенденцию
- десятки
- чем
- который
- Ассоциация
- их
- Их
- сами
- тогда
- Там.
- следовательно
- Эти
- они
- вещи
- think
- Думает
- этой
- те
- хоть?
- мысль
- тысячи
- Через
- по всему
- Таким образом
- плотно
- время
- раз
- в
- вместе
- слишком
- Переводы
- прозрачный
- улавливать
- правда
- Поворот
- два
- напишите
- Типы
- типично
- укр
- В конечном счете
- непокрытый
- понимать
- Университет
- Университет Калифорнии
- неизвестный
- на
- использование
- используемый
- полезный
- обычно
- Наши ценности
- различный
- Огромная
- очень
- жизнеспособный
- вирус
- Режимы
- Ожидание
- законопроект
- Смотреть
- Вода
- Путь..
- способы
- we
- слабый
- WebP
- ЧТО Ж
- вполне определенный
- Скважины
- были
- Что
- когда
- будь то
- который
- в то время как
- КТО
- все
- полностью
- зачем
- будете
- в
- без
- Работа
- работавший
- мире
- червь
- бы
- письменный
- писал
- еще
- Ты
- молодой
- зефирнет