Вступ
Відкриття під час проекту «Геном людини» на початку 2000-х років того, що ми, люди, маємо лише близько 20,000 XNUMX генів, що кодують білок — приблизно стільки ж, скільки у крихітного черв’яка-нематоди, що живе в ґрунті, і менше ніж вдвічі менше, ніж у рослини рису, — стало шоком. . Однак цей удар по нашій гордості був пом’якшений ідеєю, що людський геном багатий регуляторними зв’язками. Наші гени взаємодіють у щільній мережі, у якій фрагменти ДНК і молекули, які вони кодують (РНК і білки), контролюють «експресію» інших генів, впливаючи на те, чи утворюють вони відповідні РНК і білки. Щоб зрозуміти геном людини, нам потрібно було зрозуміти процес регуляції генів.
Однак це завдання виявляється набагато складнішим, ніж розшифровка послідовності геному.
Спочатку підозрювали, що регуляція генів є простою справою, коли один генний продукт діє як перемикач для іншого гена, цифровим способом. У 1960-х роках французькі біологи Франсуа Жакоб і Жак Моно вперше з'ясували процес генної регуляції в механістичних подробицях: В Кишкова паличка бактерій, коли білок-репресор зв’язується з певним сегментом ДНК, він блокує транскрипцію та трансляцію суміжного набору генів, які кодують ферменти для перетравлення цукру лактози. Цей регуляторний контур, який Моно та Якоб охрестили озеро operon, має акуратну, прозору логіку.
Але регуляція генів у складних багатоплідних — тварин, подібних до людини, зі складними еукаріотичними клітинами — зазвичай не працює таким чином. Натомість це включає в себе групу молекул, включаючи білки, РНК і фрагменти ДНК з усієї хромосоми, які якимось чином співпрацюють, щоб контролювати експресію гена.
Справа не тільки в тому, що цей регуляторний процес в еукаріотах має більше гравців, ніж це зазвичай спостерігається в бактеріях та інших простих прокаріотичних клітинах; це, здається, категорично інший процес, і туманніший.
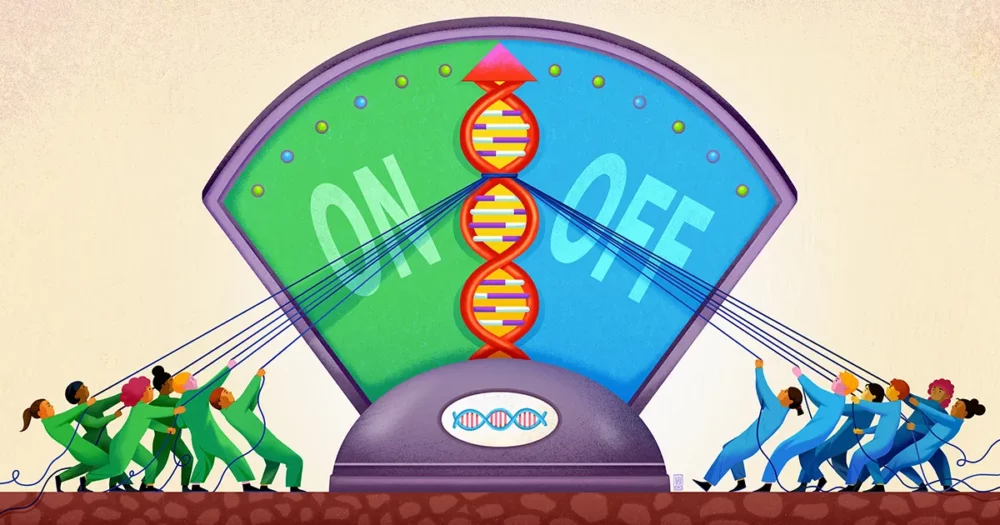
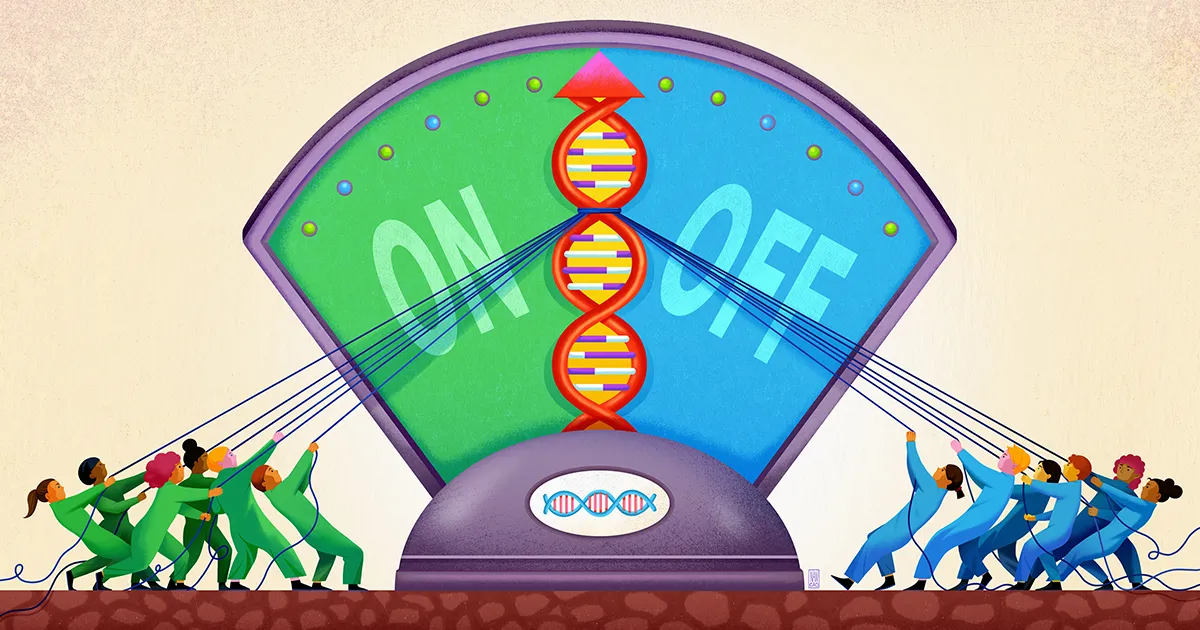
Команда Стенфордського університету під керівництвом біофізика та біоінженера Поллі Фордайс, тепер, здається, виявив компонент цього нечіткого способу регуляції генів. Їхня робота, опублікований у вересні минулого року в наука, припускає, що ДНК поблизу гена діє як своєрідний неглибокий колодязь для захоплення різноманітних регуляторних молекул, підтримуючи їх готовими до дії, щоб у разі потреби вони могли додати свій голос до рішення про те, чи активувати ген.
Вступ
Ці регуляторні лунки зроблені з явно дивних ділянок ДНК. Вони складаються з послідовностей, у яких коротка ділянка ДНК довжиною від однієї до шести пар основ повторюється багато разів. Десятки копій цих «коротких тандемних повторень» (STR) можна нанизувати разом у ці послідовності, як те саме маленьке «слово», написане знову і знову.
STR у великій кількості присутні в геномі людини: вони складають близько 5% усієї нашої ДНК. Колись їх вважали класичними прикладами «сміттєвої» ДНК, тому що повторюваний «текст» ДНК, що складається лише з STR, не може містити майже стільки значущої інформації, скільки, скажімо, неправильна послідовність літер, що складають речення в цьому стаття.
І все ж STR явно не є незначним: вони були пов’язані з такими захворюваннями, як хвороба Гентінгтона, спінобульбарна м’язова атрофія, хвороба Крона та деякі види раку. За останні кілька десятиліть накопичено докази того, що вони можуть якимось чином посилювати або гальмувати регуляцію генів. Загадка полягала в тому, як вони могли бути настільки потужними, маючи так мало інформації.
Складні елементи керування для складних комірок
Щоб зрозуміти, як STR вписуються в загальну картину регуляції генів, давайте зробимо крок назад. Гени зазвичай фланковані фрагментами ДНК, які не кодують РНК або білок, але виконують регуляторні функції. Бактеріальні гени мають «промоторні» ділянки, де ферменти полімерази можуть зв’язуватися, щоб розпочати транскрипцію суміжної ДНК у РНК. Вони також зазвичай мають «операторські» ділянки, де білки-репресори можуть зв’язуватися, блокуючи транскрипцію, вимикаючи ген, як у озеро оперон.
У людей та інших еукаріотів регуляторні послідовності можуть бути численнішими, різноманітнішими — і викликати здивування. Області, які називаються енхансерами, наприклад, впливають на ймовірність того, що ген буде транскрибований. Енхансери часто є мішенями для білків, які називаються факторами транскрипції, які можуть зв’язуватися, посилюючи або пригнічуючи експресію генів. Дивно, але деякі енхансери знаходяться на відстані десятків тисяч пар основ від генів, які вони регулюють, і наближаються до них лише через фізичну перебудову петель ДНК у упакованій хромосомі.
Регуляція еукаріотичних генів зазвичай включає в себе ці багато різноманітних регуляторних блоків ДНК разом з одним або декількома факторами транскрипції та іншими молекулами, які збираються навколо гена, як комітет, скликаний для вирішення того, що він повинен робити. Вони збираються в пухке щільне скупчення.
Часто молекулярні учасники, здається, також не взаємодіють через дуже вибіркові пари «замок і ключ», поширені в молекулярній біології. Натомість вони набагато менш вибагливі, взаємодіють досить слабко й невибірково, ніби блукаючи й затіваючи короткі розмови один з одним.
Насправді те, як фактори транскрипції зв’язуються з ДНК еукаріотів, було чимось на зразок загадки. Довгий час вважалося, що якась частина транскрипційного фактора повинна точно відповідати зв’язувальній послідовності «мотиву» в ДНК, як шматочки головоломки. Але хоча деякі такі мотиви були ідентифіковані, їх присутність не завжди добре корелює з тим, де вчені знаходять фактори транскрипції, що прилипають до ДНК у клітинах. Іноді транскрипційні фактори затримуються в областях без будь-яких мотивів, тоді як деякі мотиви, які, здається, повинні міцно зв’язувати транскрипційні фактори, залишаються порожніми.
«Традиційно в геноміці мета полягала в тому, щоб класифікувати геномні сайти [бінарним] способом як «зв’язані» або «незв’язані»» за факторами транскрипції, сказав Фордайс. «Але картина набагато складніша». Окремі члени цих генно-регуляторних «комітетів», здається, не завжди присутні на своїх засіданнях або відсутні на них, а мають різну ймовірність бути там чи ні.
Тенденція регуляції генів в еукаріотах покладатися на таку кількість різноманітних слабких взаємодій між великими молекулярними комплексами «є однією з речей, через яку, як відомо, важко впоратися теоретично», — сказав біофізик. Томас Кульман Каліфорнійського університету в Ріверсайді, який написав коментар на папері лабораторії Fordyce для наука. Це глибока загадка, як із цього, здавалося б, хаотичного процесу виникають точні рішення про ввімкнення та вимкнення генів.
Окрім таємничої нечіткої логіки цього процесу прийняття рішень, є також питання про те, як усі члени комітету взагалі знаходять дорогу до потрібної кімнати — і потім там залишаються. Молекули, як правило, рухаються навколо клітини шляхом дифузії, під впливом інших навколишніх молекул, таких як вода, і блукаючи у випадкових напрямках. Ми можемо очікувати, що ці вільні комітети надто швидко розпадуться, щоб виконувати свою регуляторну роботу.
Саме тут, на думку Фордайс та її колег, з’являються STR. STR вражаюче поширені в енхансерних сайтах ДНК. У своїй статті дослідники стверджують, що STR діють як липкі плями, які збирають транскрипційні фактори та запобігають їх відхиленню.
Точне налаштування липкості
Група Фордайса систематично досліджувала, як відмінності в послідовності STR впливають на прилипання факторів транскрипції до мотиву зв'язування. Вони розглядали два фактори — один від дріжджів, інший від людей — які прикріплюються до конкретного шестиосновного мотиву. Дослідники виміряли як силу (або спорідненість) цього зв’язування, так і швидкість, з якою фактори транскрипції застрягають і відклеюються (кінетика), коли мотив фланкований STR замість випадкової послідовності. Для порівняння вони перевірили, наскільки легко фактори зв’язуються лише з STR і з цілком випадковою послідовністю ДНК.
«Одна з найбільших проблем у цій галузі полягає в тому, щоб роз’єднати безліч змінних, які впливають на зв’язування [транскрипційного фактора] у певній позиції геному», — сказав Девід Сутер, молекулярний біолог Швейцарського федерального технологічного інституту в Лозанні, Швейцарія. Форма ДНК, близькість до інших сегментів ДНК і фізичне напруження в молекулах ДНК можуть відігравати важливу роль у зв’язуванні фактора транскрипції. Значення цих параметрів, ймовірно, відрізняються в кожній позиції в геномі, і, можливо, також між типами клітин і в одній клітині протягом певного часу в даній позиції. «Це величезний простір невідомих змінних, які дуже важко кількісно визначити», — сказав Сутер.
Вступ
Ось чому добре контрольовані експерименти, такі як експерименти Стенфордської команди, такі корисні, додав Кулман. Зазвичай, коли дослідникам потрібно виміряти подібні слабкі взаємодії, у них є два варіанти: вони можуть зробити кілька дуже детальних, надзвичайно точних вимірювань і узагальнити їх, або вони можуть провести велику кількість швидких і брудних вимірювань і використовувати математично складні статистичні методи для виведення результатів. Але Фордайс та її колеги, за словами Кульмана, використовували автоматизовану процедуру на основі мікрофлюїдного чіпа для проведення точних вимірювань під час високопродуктивних експериментів, «щоб отримати найкраще з обох світів».
Стенфордська команда виявила, що різні послідовності STR можуть змінювати афінність зв’язування факторів транскрипції з ДНК у 70 разів; вони іноді мають сильніший вплив на зв'язування фактора транскрипції, ніж зміна послідовності самого мотиву зв'язування. І ефекти були різними для двох різних факторів транскрипції, які вони розглядали.
Таким чином, STR, здається, здатні точно налаштовувати здатність факторів транскрипції приєднуватися до ділянки ДНК і таким чином регулювати ген. Але як саме?
Зала очікування біля ген
Дослідники прийшли до висновку, що частина фактора транскрипції, яка зв’язує ДНК, може слабко взаємодіяти з STR, причому точна сила цієї спорідненості залежить від послідовності STR. Оскільки таке зв’язування є слабким, воно не матиме великої специфічності. Але якщо фактор транскрипції слабко захоплюється і вивільняється STR знову і знову, кумулятивний ефект полягає в утриманні фактора транскрипції в околицях гена, щоб він з більшою ймовірністю надійно зв’язувався з ділянкою мотиву, якщо це необхідно.
Фордайс та її колеги передбачили, що STR, таким чином, діють як «вестибюль» або колодязь, де транскрипційні фактори можуть збиратися, хоча й короткочасно, поблизу регуляторного сайту зв’язування. «Повторюваний характер STR підсилює слабкий ефект будь-якого окремого сайту зв’язування, з якого він складається», — сказав Коннор Хортон, перший автор дослідження, який зараз є докторантом Каліфорнійського університету в Берклі.
І навпаки, додав він, деякі STR також можуть діяти, щоб відтягнути транскрипційні фактори від регуляторних послідовностей, вбираючи транскрипційні фактори в інших місцях, як губка. Таким чином вони можуть пригнічувати експресію генів.
За словами Сатера, робота переконливо показує, що STR безпосередньо впливають на зв’язування факторів транскрипції in vitro. Більше того, Стенфордська команда використала алгоритм машинного навчання, щоб показати, що ефекти, які спостерігаються в їхніх експериментах in vitro, також, здається, відбуваються в живих клітинах (тобто in vivo).
але Роберт Тзянь, біохімік з Берклі та дослідник Медичного інституту Говарда Хьюза, вважає, що, можливо, ще занадто рано бути впевненим у тому, який вплив дана комбінація STR-фактора транскрипції має на експресію генів у реальних клітинах.
Тянь, Ксав'є Дарзак і їхні колеги в лабораторії, якою вони керують разом у Берклі, погоджуються, що STRs, здається, пропонують спосіб концентрації транскрипційних факторів поблизу сайтів регуляції генів. Однак, не знаючи, наскільки близькими повинні бути фактори, щоб активувати транскрипцію, важко зрозуміти функціональне значення цього результату. Тзянь сказав, що він хотів би побачити, чи введення STR у живу клітину передбачувано впливає на експресію цільового гена. Наразі, за його словами, він «не переконаний, що STR обов’язково будуть основним аспектом [регуляторних] механізмів in vivo».
Комбінаторна граматика
Одна з головоломок полягає в тому, як такий механізм надійно забезпечує тип точної регуляції генів, який необхідний клітинам, оскільки як сила, так і вибірковість зв’язування транскрипційного фактора в лунках STR слабкі. Фордайс вважає, що така специфічність впливу може походити з багатьох джерел — не тільки через відмінності в послідовностях STR, але також через кооперативну взаємодію між факторами транскрипції та іншими білками, залученими до регуляції.
Враховуючи все це, Хортон сказав, що не ясно, чи буде просто передбачити вплив даної комбінації STR-фактора транскрипції на експресію гена. Логіка процесу справді нечітка. І «граматика» впливу, ймовірно, комбінаторна, додав Хортон: результат залежить від різних комбінацій факторів транскрипції та інших молекул.
Стенфордська команда вважає, що, можливо, 90% факторів транскрипції чутливі до STR, але типів факторів транскрипції в геномі людини набагато більше, ніж типів STR. «Мутація послідовності STR може вплинути на зв’язування 20 різних факторів транскрипції в цьому типі клітин, що призведе до загального зниження транскрипції цього сусіднього гена без залучення будь-якого конкретного фактора транскрипції», – сказав Хортон.
Таким чином, по суті, Стенфордська команда погоджується з Тіаном, що регуляція генів у живих клітинах не буде керуватися єдиним простим механізмом. Швидше, фактори транскрипції, їхні сайти зв’язування з ДНК та інші регуляторні молекули можуть збиратися у щільні скупчення, які впливають колективно.
«Тепер є багато прикладів, які підтверджують ідею, що елементи ДНК можуть переповнювати фактори транскрипції до точки, коли вони утворюють конденсати з кофакторами», — сказав Річард Янг, клітинний біолог в Інституті Уайтхеда Массачусетського технологічного інституту. Енхансери зв'язують багато факторів транскрипції, щоб створити таке скупчення. STRs можуть бути інгредієнтом, який допомагає зібрати транскрипційні фактори для групування біля гена, але це не вся історія.
Навіщо регулювати гени таким складним чином, замість того, щоб покладатися на такі сильні та специфічні взаємодії між регуляторними білками та ділянками ДНК, які домінують у прокаріотів? Цілком можливо, що саме така нечіткість взагалі зробила можливими великі складні метазоани.
Щоб бути життєздатними видами, організми повинні мати можливість розвиватися та пристосовуватися до мінливих обставин. Якби наші клітини покладалися на якусь величезну, але суворо встановлену мережу регуляційних взаємодій генів, було б важко внести в неї будь-які зміни, не порушивши всієї техніки, так само як швейцарський годинник заклинить, якщо ми видалимо (або навіть трохи зрушимо) будь-яку його безлічі зубчастих коліс. Однак, якщо регулятивні молекулярні взаємодії нещільні та досить неспецифічні, у системі є корисна слабина — так само, як комітет зазвичай може прийняти правильне рішення, навіть якщо один із його членів хворіє.
Фордайс зазначає, що в таких прокаріотах, як бактерії, факторам транскрипції може бути відносно легко знайти свої сайти зв’язування, оскільки геном, який потрібно шукати, менший. Але це стає складніше, оскільки геном стає більшим. У великих геномах еукаріотів «ви більше не можете терпіти ризик того, що ви тимчасово застрягнете в «неправильному» місці зв’язування», – сказав Фордайс, оскільки це поставило б під загрозу здатність швидко реагувати на зміну умов навколишнього середовища.
Крім того, самі STR є дуже еволюційними. Подовження або вкорочення їхньої послідовності або зміна розміру та глибини «колодки транскрипційного фактора» може легко статися через збої в реплікації чи репарації ДНК або через статеву рекомбінацію хромосом. Для Фордайса це припускає, що STR «можуть служити сировиною для розробки нових регуляторних елементів і тонкого налаштування існуючих регуляторних модулів для чутливих програм транскрипції», таких як ті, що керують розвитком тварин і рослин.
Сила слабких взаємодій
Такі міркування змушують молекулярних біологів приділяти набагато більше уваги слабким і відносно невибірковим взаємодіям у геномі. Багато з них включають білки, які замість того, щоб мати фіксовану і точну структуру, є пухкими та гнучкими — «внутрішньо невпорядкованими», як кажуть біохіміки. Якби білки працювали лише через жорсткі структурні домени, пояснив Янг, це обмежувало б не лише те, наскільки добре могли б розвиватися регуляторні системи, але й види динамічного регулювання, які спостерігаються в житті. «Ви не знайдете живий організм — чи навіть вірус — який би функціонував лише зі стабільними структурними елементами, як у швейцарських годинниках», — сказав Янг.
Можливо, еволюція просто наштовхнулася на STR як компонент такого складного, але в кінцевому підсумку більш ефективного рішення для регуляції генів в еукаріотах. Самі STR можуть виникати декількома способами — наприклад, через помилки в реплікації ДНК або через активність сегментів ДНК, які називаються переносними елементами, які створюють свої копії по всьому геному.
«Так сталося, що слабка взаємодія між білками та повторюваними послідовностями, що виникла в результаті, могла … забезпечити селективну перевагу клітинам, де це сталося», — сказав Кульман. Його припущення полягає в тому, що ця нечіткість, ймовірно, була нав’язана еукаріотам, але «згодом вони змогли використати [це] для власної вигоди». Бактерії та інші прокаріоти можуть покладатися на чітко визначену «цифрову» регулятивну логіку, оскільки їхні клітини, як правило, існують лише в кількох простих, чітких станах, таких як пересування та реплікація.
Але різні стани клітин багатоплідних є «набагато складнішими та іноді близькими до континууму», сказав Сатер, тому вони краще обслуговуються більш нечітким «аналоговим» регулюванням.
«Системи регуляції генів у бактерій і еукаріот, здається, досить суттєво розійшлися», — погодився Тзянь. Хоча кажуть, що Моно одного разу зауважив, що «те, що вірно для E. палички це вірно для слона», здається, що це не завжди так.
- Розповсюдження контенту та PR на основі SEO. Отримайте посилення сьогодні.
- PlatoData.Network Vertical Generative Ai. Додайте собі сили. Доступ тут.
- PlatoAiStream. Web3 Intelligence. Розширення знань. Доступ тут.
- ПлатонЕСГ. вуглець, CleanTech, Енергія, Навколишнє середовище, Сонячна, Поводження з відходами. Доступ тут.
- PlatoHealth. Розвідка про біотехнології та клінічні випробування. Доступ тут.
- джерело: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- : має
- :є
- : ні
- :де
- ][стор
- $UP
- 000
- 20
- 70
- a
- здатність
- Здатний
- МЕНЮ
- відсутнім
- рясний
- Накопичений
- Діяти
- діючий
- дію
- активоване
- діяльність
- акти
- пристосовувати
- додавати
- доданий
- сусідній
- Перевага
- впливати
- спорідненість
- знову
- вирішено
- погоджується
- алгоритм
- ВСІ
- тільки
- по
- Також
- хоча
- завжди
- серед
- посилюється
- an
- та
- тварини
- Інший
- будь-який
- крім
- ЕСТЬ
- сперечатися
- виникати
- навколо
- стаття
- AS
- зовнішній вигляд
- передбачається
- At
- увагу
- автор
- Автоматизований
- геть
- назад
- Бактерії
- база
- BE
- оскільки
- ставати
- було
- починати
- буття
- користь
- Берклі
- КРАЩЕ
- Краще
- між
- Великий
- Велика картина
- більший
- найбільший
- пов'язувати
- обов'язковий
- біологія
- Блокувати
- блоки
- підривати
- підвищення
- обидва
- приніс
- але
- by
- Каліфорнія
- званий
- прийшов
- CAN
- осередок
- Клітини
- певний
- проблеми
- Зміни
- заміна
- вибір
- Хромосоми
- обставин
- classic
- Класифікувати
- ясно
- очевидно
- близько
- тісно
- кластер
- співпрацювати
- колеги
- колективно
- поєднання
- комбінації
- Приходити
- комітет
- загальний
- порівняння
- комплекс
- складний
- компонент
- компроміс
- Умови
- Зв'язки
- міркування
- зміст
- Континуум
- контроль
- управління
- розмови
- кооперативний
- може
- Пара
- натовп
- десятиліття
- вирішувати
- рішення
- рішення
- Декодування
- зменшити
- Залежно
- залежить
- глибина
- деталь
- докладно
- розробка
- відрізняються
- Відмінності
- різний
- важкий
- радіомовлення
- цифровий
- цифрова мода
- безпосередньо
- відкриття
- Захворювання
- чіткий
- Різне
- ДНК
- do
- Док
- Ні
- домени
- Домінувати
- Не знаю
- керований
- охрестили
- під час
- динамічний
- Рано
- легко
- легко
- ефект
- Ефективний
- ефекти
- або
- елементи
- слон
- в іншому місці
- з'являтися
- підвищувати
- навколишній
- помилки
- Навіть
- Кожен
- докази
- еволюція
- еволюціонувати
- еволюціонує
- точно
- приклад
- Приклади
- існувати
- існуючий
- очікувати
- Експерименти
- пояснені
- Експлуатувати
- вираз
- надзвичайно
- факт
- фактор
- фактори
- мода
- Федеральний
- кілька
- поле
- розібрався
- знайти
- Перший
- відповідати
- фіксованою
- для
- примусовий
- форма
- знайдений
- французька
- від
- функціональний
- функціонування
- Функції
- Банда
- збирати
- збір
- зібрання
- в цілому
- геномика
- отримати
- отримує
- даний
- мета
- буде
- добре
- управління
- великий
- Group
- здогадуватися
- Половина
- обробляти
- сталося
- Жорсткий
- важче
- Мати
- має
- he
- допомагає
- її
- дуже
- його
- тримати
- Як
- Однак
- HTML
- HTTPS
- величезний
- людина
- Людей
- ідея
- ідентифікований
- if
- Impact
- in
- У тому числі
- дійсно
- індивідуальний
- вплив
- впливаючи
- інформація
- незначний
- замість
- Інститут
- взаємодіяти
- взаємодіючих
- Взаємодії
- в
- введення
- незмінно
- залучати
- залучений
- включає в себе
- IT
- ЙОГО
- сам
- Джекоб
- Головоломка
- робота
- просто
- тримати
- зберігання
- Дитина
- види
- Знання
- lab
- великий
- останній
- провідний
- вивчення
- Led
- менше
- життя
- як
- Ймовірно
- пов'язаний
- трохи
- життя
- логіка
- Довго
- довше
- подивився
- машина
- навчання за допомогою машини
- made
- журнал
- основний
- зробити
- РОБОТИ
- манера
- багато
- Массачусетс
- Массачусетський технологічний інститут
- матч
- матеріал
- математично
- Матерія
- Може..
- може бути
- значущим
- вимір
- вимірювання
- механізм
- механізми
- медичний
- зустрічі
- члени
- методика
- може бути
- MIT
- режим
- Модулі
- молекулярний
- молекули
- більше
- рухатися
- переміщення
- багато
- множинний
- повинен
- безліч
- таємничий
- Таємниця
- природа
- Близько
- майже
- обов'язково
- Необхідність
- необхідний
- мережу
- немає
- примітки
- зараз
- нюанс
- численний
- відбуваються
- сталося
- трапляються
- of
- від
- пропонувати
- часто
- on
- один раз
- ONE
- тільки
- or
- Інше
- наші
- з
- Результат
- над
- загальний
- власний
- упакований
- спаровування
- пар
- Папір
- параметри
- частина
- Учасники
- приватність
- Минуле
- Патчі
- Платити
- може бути
- переконаний
- фізичний
- картина
- частин
- Рослини
- plato
- Інформація про дані Платона
- PlatoData
- Play
- гравці
- точка
- положення
- це можливо
- влада
- потужний
- необхідність
- передбачати
- передвіщений
- наявність
- представити
- гордість
- ймовірно
- процедура
- процес
- виробляти
- Product
- глибокий
- програми
- проект
- Білок
- Білки
- забезпечувати
- забезпечує
- доведення
- опублікований
- put
- головоломка
- питання
- швидко
- досить
- випадковий
- ставка
- швидше
- Сировина
- легко
- готовий
- реальний
- регіон
- райони
- Регулювати
- Регулювання
- регуляторні
- щодо
- випущений
- покладатися
- покладаючись
- залишатися
- зауважив
- видаляти
- ремонт
- повторювані
- копіювання
- Дослідники
- ті
- Реагувати
- результат
- в результаті
- результати
- рис
- Багаті
- право
- жорсткий
- Risk
- прибережна смуга
- РНК
- Роль
- Кімната
- звичайно
- прогін
- Зазначений
- то ж
- say
- Вчені
- безпечно
- побачити
- здається
- мабуть
- Здається,
- бачив
- сегмент
- сегменти
- Захопити
- селективний
- чутливий
- пропозиція
- Вересень
- Послідовність
- служити
- служив
- кілька
- Сексуальний
- дрібний
- Форма
- Короткий
- Повинен
- Показувати
- значення
- простий
- з
- один
- сайт
- сайти
- SIX
- Розмір
- слабкий
- менше
- So
- рішення
- деякі
- якось
- що в сім'ї щось
- іноді
- Джерела
- Простір
- конкретний
- специфічність
- стабільний
- Станфорд
- Стенфордський університет
- Штати
- статистичний
- залишатися
- Крок
- прилипання
- липкий
- Стоп
- Історія
- просто
- сила
- сильний
- сильно
- структурний
- структура
- студент
- Вивчення
- Згодом
- по суті
- такі
- цукор
- Запропонує
- набір
- підтримка
- Переконайтеся
- Навколо
- Швейцарський
- перемикач
- Швейцарія
- система
- Systems
- Приймати
- Тандем
- Мета
- цілі
- Завдання
- команда
- Технологія
- розповідає
- як правило,
- тензор
- ніж
- Що
- Команда
- їх
- Їх
- самі
- потім
- Там.
- отже
- Ці
- вони
- речі
- думати
- Думає
- це
- ті
- хоча?
- думка
- тисячі
- через
- по всьому
- Таким чином
- щільно
- час
- times
- до
- разом
- занадто
- Переклад
- прозорий
- захоплення
- правда
- Поворот
- два
- тип
- Типи
- типово
- укр
- Зрештою
- непокритий
- розуміти
- університет
- Університет Каліфорнії
- невідомий
- на
- використання
- використовуваний
- корисний
- зазвичай
- Цінності
- різний
- величезний
- дуже
- viable
- вірус
- Голос
- Очікування
- було
- годинник
- вода
- шлях..
- способи
- we
- слабкий
- webp
- ДОБРЕ
- добре визначений
- Wells
- були
- Що
- коли
- Чи
- який
- в той час як
- ВООЗ
- всі
- повністю
- чому
- волі
- з
- в
- без
- Work
- працював
- світі
- черв'як
- б
- письмовий
- пише
- ще
- Ти
- молодий
- зефірнет