প্রোটিন অনেক জৈবিক প্রক্রিয়া চালায়, যেমন এনজাইম কার্যকলাপ, আণবিক পরিবহন, এবং সেলুলার সমর্থন। একটি প্রোটিনের ত্রিমাত্রিক গঠন তার কার্যকারিতার অন্তর্দৃষ্টি প্রদান করে এবং কীভাবে এটি অন্যান্য জৈব অণুর সাথে মিথস্ক্রিয়া করে। প্রোটিন গঠন নির্ধারণের পরীক্ষামূলক পদ্ধতি, যেমন এক্স-রে ক্রিস্টালোগ্রাফি এবং NMR স্পেকট্রোস্কোপি, ব্যয়বহুল এবং সময়সাপেক্ষ।
বিপরীতে, সম্প্রতি-বিকশিত গণনা পদ্ধতিগুলি দ্রুত এবং সঠিকভাবে একটি প্রোটিনের গঠনটি তার অ্যামিনো অ্যাসিড ক্রম থেকে ভবিষ্যদ্বাণী করতে পারে। এই পদ্ধতিগুলি প্রোটিনের জন্য গুরুত্বপূর্ণ যা পরীক্ষামূলকভাবে অধ্যয়ন করা কঠিন, যেমন ঝিল্লি প্রোটিন, অনেক ওষুধের লক্ষ্য। এর একটি সুপরিচিত উদাহরণ আলফাফোল্ড, একটি গভীর শিক্ষা-ভিত্তিক অ্যালগরিদম তার সঠিক ভবিষ্যদ্বাণীর জন্য উদযাপন করা হয়।
ESMFold অ্যামিনো অ্যাসিড ক্রম থেকে প্রোটিন গঠনের পূর্বাভাস দেওয়ার জন্য তৈরি করা আরেকটি অত্যন্ত সঠিক, গভীর শিক্ষা-ভিত্তিক পদ্ধতি। ESMFold একটি বৃহৎ প্রোটিন ভাষা মডেল (pLM) ব্যাকবোন হিসেবে ব্যবহার করে এবং শেষ থেকে শেষ পর্যন্ত কাজ করে। AlphaFold2 এর বিপরীতে, এটির একটি অনুসন্ধান বা প্রয়োজন নেই একাধিক ক্রম প্রান্তিককরণ (MSA) পদক্ষেপ, বা এটি পূর্বাভাস তৈরি করতে বহিরাগত ডাটাবেসের উপর নির্ভর করে না। পরিবর্তে, উন্নয়ন দল ইউনিরেফ থেকে লক্ষ লক্ষ প্রোটিন সিকোয়েন্সে মডেলটিকে প্রশিক্ষণ দিয়েছে। প্রশিক্ষণের সময়, মডেলটি মনোযোগের নিদর্শন তৈরি করেছে যা ক্রমানুসারে অ্যামিনো অ্যাসিডের মধ্যে বিবর্তনীয় মিথস্ক্রিয়াকে সুন্দরভাবে উপস্থাপন করে। একটি MSA এর পরিবর্তে একটি pLM এর ব্যবহার অন্যান্য অত্যাধুনিক মডেলের তুলনায় 60 গুণ দ্রুত ভবিষ্যদ্বাণীর সময় সক্ষম করে।
এই পোস্টে, আমরা Hugging Face with থেকে প্রাক-প্রশিক্ষিত ESMFold মডেল ব্যবহার করি আমাজন সেজমেকার এর ভারী চেইন কাঠামোর পূর্বাভাস দিতে ট্রাস্টুজুমাব, একটি একরঙা অ্যান্টিবডি প্রথম দ্বারা উন্নত Genentech চিকিত্সার জন্য এইচআর 2 পজিটিভ স্তন ক্যান্সার. গবেষকরা ক্রম পরিবর্তনের প্রভাব পরীক্ষা করতে চাইলে এই প্রোটিনের গঠনের দ্রুত ভবিষ্যদ্বাণী করা কার্যকর হতে পারে। এটি সম্ভাব্যভাবে উন্নত রোগীর বেঁচে থাকার বা কম পার্শ্ব প্রতিক্রিয়া হতে পারে।
এই পোস্টটি নিম্নলিখিত জুপিটার নোটবুক এবং সম্পর্কিত স্ক্রিপ্টগুলির একটি উদাহরণ প্রদান করে৷ GitHub সংগ্রহস্থল.
পূর্বশর্ত
আমরা একটি এই উদাহরণ চালানোর সুপারিশ অ্যামাজন সেজমেকার স্টুডিও নোটবই PyTorch 1.13 Python 3.9 CPU-অপ্টিমাইজ করা ইমেজ একটি ml.r5.xlarge ইনস্ট্যান্স টাইপে চলছে।
ট্রাস্টুজুমাবের পরীক্ষামূলক কাঠামোটি কল্পনা করুন
শুরু করতে, আমরা ব্যবহার করি biopython লাইব্রেরি এবং একটি সহায়ক স্ক্রিপ্ট থেকে ট্রাস্টুজুমাব কাঠামো ডাউনলোড করতে RCSB প্রোটিন ডেটা ব্যাংক:
পরবর্তী, আমরা ব্যবহার py3Dmol একটি ইন্টারেক্টিভ 3D ভিজ্যুয়ালাইজেশন হিসাবে কাঠামোটি কল্পনা করার জন্য লাইব্রেরি:
নিম্নলিখিত চিত্রটি প্রোটিন ডেটা ব্যাংক (PDB) থেকে 3D প্রোটিন গঠন 1N8Z প্রতিনিধিত্ব করে। এই ছবিতে, ট্রাস্টুজুমাব হালকা চেইনটি কমলা রঙে প্রদর্শিত হয়েছে, ভারী চেইনটি নীল (হালকা নীলে পরিবর্তনশীল অঞ্চলের সাথে), এবং HER2 অ্যান্টিজেনটি সবুজ।
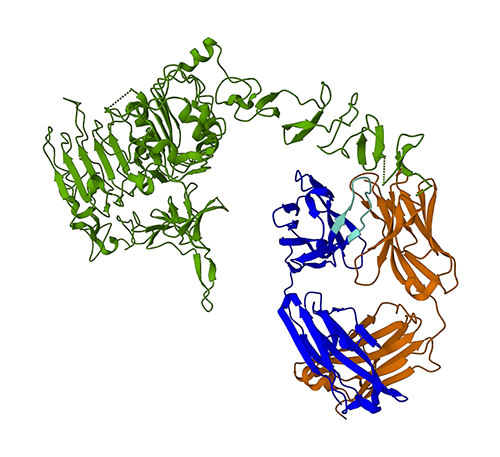
আমরা প্রথমে ইএসএমএফোল্ড ব্যবহার করব ভারী চেইন (চেইন বি) এর অ্যামিনো অ্যাসিড ক্রম থেকে কাঠামোর পূর্বাভাস দিতে। তারপরে, আমরা উপরে দেখানো পরীক্ষামূলকভাবে নির্ধারিত কাঠামোর সাথে ভবিষ্যদ্বাণীটির তুলনা করব।
ESMFold ব্যবহার করে এর ক্রম থেকে ট্রাস্টুজুমাব ভারী চেইন কাঠামোর পূর্বাভাস দিন
হেভি চেইনের গঠন ভবিষ্যদ্বাণী করতে এবং পরীক্ষামূলক ফলাফলের সাথে তুলনা করতে ESMFold মডেলটি ব্যবহার করা যাক। শুরু করতে, আমরা স্টুডিওতে একটি পূর্ব-নির্মিত নোটবুক পরিবেশ ব্যবহার করব যা বেশ কয়েকটি গুরুত্বপূর্ণ লাইব্রেরির সাথে আসে, যেমন পাইটর্চ, আগে থেকে ইনস্টল করা. যদিও আমরা আমাদের নোটবুক বিশ্লেষণের কর্মক্ষমতা উন্নত করতে একটি ত্বরান্বিত উদাহরণ টাইপ ব্যবহার করতে পারি, আমরা পরিবর্তে একটি নন-এক্সিলারেটেড উদাহরণ ব্যবহার করব এবং একটি CPU-তে ESMFold পূর্বাভাস চালাব।
প্রথমত, আমরা প্রাক-প্রশিক্ষিত ESMFold মডেল এবং টোকেনাইজার থেকে লোড করি আলিঙ্গন ফেস হাব:
এর পরে, আমরা আমাদের ডিভাইসে মডেলটি অনুলিপি করি (এই ক্ষেত্রে CPU) এবং কিছু মডেল পরামিতি সেট করি:
বিশ্লেষণের জন্য প্রোটিন ক্রম প্রস্তুত করতে, আমাদের এটিকে টোকেনাইজ করতে হবে। এটি অ্যামিনো অ্যাসিড চিহ্নগুলিকে (EVQLV…) একটি সংখ্যাসূচক বিন্যাসে অনুবাদ করে যা ESMFold মডেল বুঝতে পারে (6,19,5,10,19,…):
এর পরে, আমরা টোকেনাইজড ইনপুটটি মোডে অনুলিপি করি, একটি ভবিষ্যদ্বাণী করি এবং ফলাফলটি একটি ফাইলে সংরক্ষণ করি:
এটি একটি r3 এর মতো একটি নন-এক্সিলারেটেড ইনস্ট্যান্স টাইপে প্রায় 5 মিনিট সময় নেয়।
আমরা পরীক্ষামূলক কাঠামোর সাথে তুলনা করে ESMFold ভবিষ্যদ্বাণীর যথার্থতা পরীক্ষা করতে পারি। আমরা ব্যবহার করে এই কাজ US-সারিবদ্ধ মিশিগান বিশ্ববিদ্যালয়ের ঝাং ল্যাব দ্বারা বিকশিত টুল:
| PDBchain1 | PDBchain2 | টিএম-স্কোর |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
সার্জারির টেমপ্লেট মডেলিং স্কোর (TM-স্কোর) প্রোটিন গঠনের সাদৃশ্য মূল্যায়নের জন্য একটি মেট্রিক। 1.0 স্কোর একটি নিখুঁত ম্যাচ নির্দেশ করে। 0.7 এর উপরে স্কোরগুলি নির্দেশ করে যে প্রোটিনগুলি একই মেরুদণ্ডের গঠন ভাগ করে। 0.9 এর উপরে স্কোরগুলি নির্দেশ করে যে প্রোটিনগুলি কার্যকরীভাবে বিনিময়যোগ্য ডাউনস্ট্রিম ব্যবহারের জন্য। আমাদের TM-স্কোর 0.802 অর্জনের ক্ষেত্রে, ESMFold ভবিষ্যদ্বাণী সম্ভবত স্ট্রাকচার স্কোরিং বা লিগ্যান্ড বাইন্ডিং পরীক্ষা-নিরীক্ষার মতো অ্যাপ্লিকেশনের জন্য উপযুক্ত হবে, কিন্তু ব্যবহারের ক্ষেত্রে উপযুক্ত নাও হতে পারে আণবিক প্রতিস্থাপন যে অত্যন্ত উচ্চ নির্ভুলতা প্রয়োজন.
আমরা সারিবদ্ধ কাঠামোগুলি কল্পনা করে এই ফলাফলটি যাচাই করতে পারি। দুটি কাঠামো একটি উচ্চ, কিন্তু নিখুঁত নয়, ওভারল্যাপের ডিগ্রি দেখায়। প্রোটিন গঠন ভবিষ্যদ্বাণী একটি দ্রুত-বিকশিত ক্ষেত্র এবং অনেক গবেষণা দল আরও সঠিক অ্যালগরিদম বিকাশ করছে!
একটি সেজমেকার ইনফারেন্স এন্ডপয়েন্ট হিসাবে ESMFold স্থাপন করুন
একটি নোটবুকে মডেল ইনফারেন্স চালানো পরীক্ষা-নিরীক্ষার জন্য জরিমানা, কিন্তু আপনি যদি একটি অ্যাপ্লিকেশনের সাথে আপনার মডেলকে সংহত করতে চান? অথবা একটি MLOps পাইপলাইন? এই ক্ষেত্রে, একটি ভাল বিকল্প হল আপনার মডেলটিকে একটি অনুমান শেষ পয়েন্ট হিসাবে স্থাপন করা। নিম্নলিখিত উদাহরণে, আমরা একটি ত্বরিত উদাহরণে একটি SageMaker রিয়েল-টাইম ইনফারেন্স এন্ডপয়েন্ট হিসাবে ESMFold স্থাপন করব। সেজমেকার রিয়েল-টাইম এন্ডপয়েন্ট মেশিন লার্নিং (এমএল) মডেল স্থাপন এবং হোস্ট করার জন্য একটি মাপযোগ্য, সাশ্রয়ী এবং নিরাপদ উপায় প্রদান করে। স্বয়ংক্রিয় স্কেলিং সহ, আপনি আপনার অ্যাপ্লিকেশনের চাহিদা মেটাতে, খরচ অপ্টিমাইজ করতে এবং উচ্চ প্রাপ্যতা নিশ্চিত করতে শেষ পয়েন্টে চলমান দৃষ্টান্তের সংখ্যা সামঞ্জস্য করতে পারেন।
প্রাক বিল্ট মুখ আলিঙ্গন জন্য SageMaker পাত্রে সাধারণ কাজের জন্য গভীর শিক্ষার মডেল স্থাপন করা সহজ করে তোলে। যাইহোক, প্রোটিন গঠন ভবিষ্যদ্বাণী মত উপন্যাস ব্যবহারের ক্ষেত্রে, আমাদের একটি কাস্টম সংজ্ঞায়িত করতে হবে inference.py মডেল লোড করতে স্ক্রিপ্ট, ভবিষ্যদ্বাণী চালান এবং আউটপুট ফর্ম্যাট করুন। এই স্ক্রিপ্টে আমরা আমাদের নোটবুকে ব্যবহার করা একই কোডের অনেকটাই অন্তর্ভুক্ত করে। আমরা একটি তৈরি করি requirements.txt আমাদের শেষ পয়েন্ট ব্যবহারের জন্য কিছু পাইথন নির্ভরতা সংজ্ঞায়িত করার জন্য ফাইল। আপনি আমাদের তৈরি ফাইল দেখতে পারেন GitHub সংগ্রহস্থল.
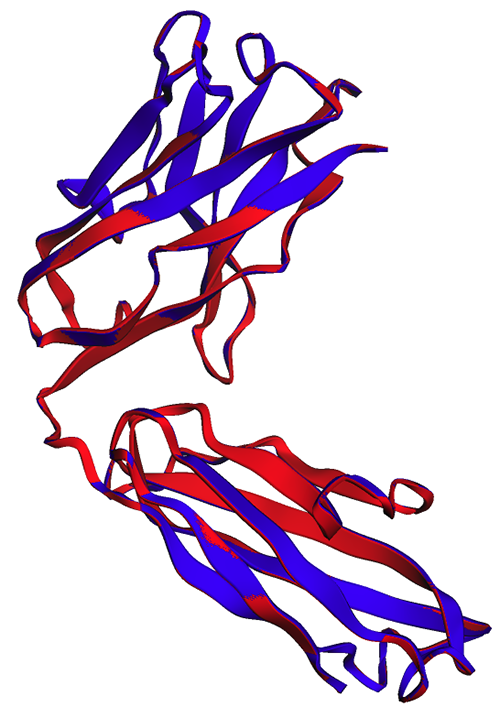
নিম্নলিখিত চিত্রে, ট্রাস্টুজুমাব ভারী চেইনের পরীক্ষামূলক (নীল) এবং পূর্বাভাসিত (লাল) কাঠামোগুলি খুব মিল, কিন্তু অভিন্ন নয়।

আমরা প্রয়োজনীয় ফাইল তৈরি করার পরে code ডিরেক্টরি, আমরা সেজমেকার ব্যবহার করে আমাদের মডেল স্থাপন করি HuggingFaceModel ক্লাস এটি সেজমেকারে হাগিং ফেস মডেলগুলি স্থাপনের প্রক্রিয়াটিকে সহজ করার জন্য একটি পূর্ব-নির্মিত ধারক ব্যবহার করে। মনে রাখবেন যে শেষবিন্দু তৈরি করতে 10 মিনিট বা তার বেশি সময় লাগতে পারে, এর প্রাপ্যতার উপর নির্ভর করে ml.g4dn আমাদের অঞ্চলে উদাহরণ প্রকার।
এন্ডপয়েন্ট ডিপ্লয়মেন্ট সম্পূর্ণ হলে, আমরা প্রোটিন সিকোয়েন্স আবার জমা দিতে পারি এবং ভবিষ্যদ্বাণীর প্রথম কয়েকটি সারি প্রদর্শন করতে পারি:
যেহেতু আমরা একটি ত্বরিত উদাহরণে আমাদের শেষ পয়েন্ট স্থাপন করেছি, ভবিষ্যদ্বাণীটি কেবল কয়েক সেকেন্ড সময় নেওয়া উচিত। ফলাফলের প্রতিটি সারি একটি একক পরমাণুর সাথে মিলে যায় এবং এতে অ্যামিনো অ্যাসিড পরিচয়, তিনটি স্থানিক স্থানাঙ্ক এবং একটি pLDDT স্কোর যে অবস্থানে ভবিষ্যদ্বাণী আস্থা প্রতিনিধিত্ব.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | দখল | PLDDT | ATOM_ID |
| ATOM | 1 | N | জিএলইউ | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | জিএলইউ | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | জিএলইউ | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | জিএলইউ | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | জিএলইউ | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | জিএলইউ | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | জিএলইউ | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | জিএলইউ | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | জিএলইউ | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | Val | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | Val | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
আগের মতো একই পদ্ধতি ব্যবহার করে, আমরা দেখতে পাচ্ছি যে নোটবুক এবং এন্ডপয়েন্ট ভবিষ্যদ্বাণীগুলি অভিন্ন।
| PDBchain1 | PDBchain2 | টিএম-স্কোর |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
নিচের চিত্রে যেমন দেখা গেছে, ESMFold ভবিষ্যদ্বাণীগুলি ইন-নোটবুক (লাল) এবং শেষবিন্দু (নীল) দ্বারা নিখুঁত প্রান্তিককরণ দেখায়।
পরিষ্কার কর
আরও চার্জ এড়াতে, আমরা আমাদের অনুমান শেষ পয়েন্ট এবং পরীক্ষার ডেটা মুছে দিই:
সারাংশ
কম্পিউটেশনাল প্রোটিন গঠন ভবিষ্যদ্বাণী প্রোটিনের কাজ বোঝার জন্য একটি গুরুত্বপূর্ণ হাতিয়ার। মৌলিক গবেষণার পাশাপাশি, AlphaFold এবং ESMFold-এর মতো অ্যালগরিদমগুলির ওষুধ এবং জৈবপ্রযুক্তিতে অনেকগুলি অ্যাপ্লিকেশন রয়েছে। এই মডেলগুলি দ্বারা উত্পন্ন কাঠামোগত অন্তর্দৃষ্টিগুলি আমাদের আরও ভালভাবে বুঝতে সাহায্য করে যে কীভাবে জৈব অণুগুলি ইন্টারঅ্যাক্ট করে। এটি তখন রোগীদের জন্য আরও ভাল ডায়াগনস্টিক সরঞ্জাম এবং থেরাপির দিকে নিয়ে যেতে পারে।
এই পোস্টে, আমরা SageMaker ব্যবহার করে একটি পরিমাপযোগ্য ইনফারেন্স এন্ডপয়েন্ট হিসাবে Hugging Face Hub থেকে ESMFold প্রোটিন ভাষা মডেলকে কীভাবে স্থাপন করতে হয় তা দেখাই। SageMaker-এ Hugging Face মডেল স্থাপন সম্পর্কে আরও তথ্যের জন্য, পড়ুন Amazon SageMaker এর সাথে Hugging Face ব্যবহার করুন. এছাড়াও আপনি আরও প্রোটিন বিজ্ঞান উদাহরণ পেতে পারেন AWS-এ দুর্দান্ত প্রোটিন বিশ্লেষণ GitHub রেপো। আপনি দেখতে চান অন্য কোন উদাহরণ আছে যদি আমাদের একটি মন্তব্য করুন!
লেখক সম্পর্কে
 ব্রায়ান অনুগত অ্যামাজন ওয়েব সার্ভিসে গ্লোবাল হেলথকেয়ার এবং লাইফ সায়েন্সেস টিমের একজন সিনিয়র এআই/এমএল সলিউশন আর্কিটেক্ট। তার বায়োটেকনোলজি এবং মেশিন লার্নিং-এ 17 বছরেরও বেশি অভিজ্ঞতা রয়েছে এবং গ্রাহকদের জিনোমিক এবং প্রোটিওমিক চ্যালেঞ্জগুলি সমাধান করতে সাহায্য করার বিষয়ে উত্সাহী৷ তার অবসর সময়ে, তিনি তার বন্ধু এবং পরিবারের সাথে রান্না এবং খাওয়া উপভোগ করেন।
ব্রায়ান অনুগত অ্যামাজন ওয়েব সার্ভিসে গ্লোবাল হেলথকেয়ার এবং লাইফ সায়েন্সেস টিমের একজন সিনিয়র এআই/এমএল সলিউশন আর্কিটেক্ট। তার বায়োটেকনোলজি এবং মেশিন লার্নিং-এ 17 বছরেরও বেশি অভিজ্ঞতা রয়েছে এবং গ্রাহকদের জিনোমিক এবং প্রোটিওমিক চ্যালেঞ্জগুলি সমাধান করতে সাহায্য করার বিষয়ে উত্সাহী৷ তার অবসর সময়ে, তিনি তার বন্ধু এবং পরিবারের সাথে রান্না এবং খাওয়া উপভোগ করেন।
 শমিকা আরিয়াওয়ানসা অ্যামাজন ওয়েব সার্ভিসেসের গ্লোবাল হেলথকেয়ার এবং লাইফ সায়েন্সেস টিমের একজন এআই/এমএল স্পেশালিস্ট সলিউশন আর্কিটেক্ট। তিনি প্রযুক্তিগত দিকনির্দেশনা প্রদান করে এবং AWS-এ নিরাপদ ক্লাউড সমাধানগুলি উদ্ভাবন ও তৈরি করতে সহায়তা করে গ্রাহকদের তাদের AI এবং ML গ্রহণকে ত্বরান্বিত করতে তাদের সাথে কাজ করেন। কাজের বাইরে, তিনি স্কিইং এবং অফ-রোডিং পছন্দ করেন।
শমিকা আরিয়াওয়ানসা অ্যামাজন ওয়েব সার্ভিসেসের গ্লোবাল হেলথকেয়ার এবং লাইফ সায়েন্সেস টিমের একজন এআই/এমএল স্পেশালিস্ট সলিউশন আর্কিটেক্ট। তিনি প্রযুক্তিগত দিকনির্দেশনা প্রদান করে এবং AWS-এ নিরাপদ ক্লাউড সমাধানগুলি উদ্ভাবন ও তৈরি করতে সহায়তা করে গ্রাহকদের তাদের AI এবং ML গ্রহণকে ত্বরান্বিত করতে তাদের সাথে কাজ করেন। কাজের বাইরে, তিনি স্কিইং এবং অফ-রোডিং পছন্দ করেন।
 ইয়ানজুন কুই AWS মেশিন লার্নিং সলিউশন ল্যাবের একজন সিনিয়র ফলিত বিজ্ঞান ব্যবস্থাপক। তিনি AWS গ্রাহকদের তাদের AI এবং ক্লাউড গ্রহণের গতি বাড়াতে সাহায্য করার জন্য মেশিন লার্নিং উদ্ভাবন এবং প্রয়োগ করেন।
ইয়ানজুন কুই AWS মেশিন লার্নিং সলিউশন ল্যাবের একজন সিনিয়র ফলিত বিজ্ঞান ব্যবস্থাপক। তিনি AWS গ্রাহকদের তাদের AI এবং ক্লাউড গ্রহণের গতি বাড়াতে সাহায্য করার জন্য মেশিন লার্নিং উদ্ভাবন এবং প্রয়োগ করেন।
- এসইও চালিত বিষয়বস্তু এবং পিআর বিতরণ। আজই পরিবর্ধিত পান।
- প্লেটোএআইস্ট্রিম। Web3 ডেটা ইন্টেলিজেন্স। জ্ঞান প্রসারিত. এখানে প্রবেশ করুন.
- অ্যাড্রিয়েন অ্যাশলির সাথে ভবিষ্যত মিন্টিং। এখানে প্রবেশ করুন.
- PREIPO® এর সাথে PRE-IPO কোম্পানিতে শেয়ার কিনুন এবং বিক্রি করুন। এখানে প্রবেশ করুন.
- উত্স: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- : আছে
- : হয়
- :না
- $ ইউপি
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- সম্পর্কে
- উপরে
- AC
- দ্রুততর করা
- দ্রুততর
- সঠিকতা
- সঠিক
- সঠিক
- অর্জনের
- কার্যকলাপ
- যোগ
- গ্রহণ
- AI
- এআই / এমএল
- অ্যালগরিদম
- আলগোরিদিম
- প্রান্তিককৃত
- শ্রেণীবিন্যাস
- এছাড়াও
- যদিও
- মর্দানী স্ত্রীলোক
- আমাজন সেজমেকার
- অ্যামাজন ওয়েব সার্ভিসেস
- an
- বিশ্লেষণ
- এবং
- অন্য
- কোন
- আবেদন
- অ্যাপ্লিকেশন
- ফলিত
- যথাযথ
- রয়েছি
- AS
- পরিমাপন
- At
- পরমাণু
- মনোযোগ
- স্বয়ংক্রিয়
- উপস্থিতি
- এড়াতে
- ডেস্কটপ AWS
- এডাব্লুএস মেশিন লার্নিং
- দাঁড়া
- ব্যাংক
- মৌলিক
- BE
- আগে
- শুরু করা
- উত্তম
- মধ্যে
- বাঁধাই
- জৈবপ্রযুক্তি
- নীল
- নির্মাণ করা
- কিন্তু
- by
- CAN
- কর্কটরাশি
- কার্টুন
- কেস
- মামলা
- সুপ্রসিদ্ধ
- চেন
- চ্যালেঞ্জ
- চার্জ
- চেক
- শ্রেণী
- মেঘ
- মেঘ গ্রহণ
- কোড
- রঙ
- আসে
- মন্তব্য
- সাধারণ
- তুলনা করা
- তুলনা
- সম্পূর্ণ
- বিশ্বাস
- আধার
- বিপরীত হত্তয়া
- অনুরূপ
- সাশ্রয়ের
- খরচ
- পারা
- সৃষ্টি
- নির্মিত
- সংকটপূর্ণ
- গ্রাহকদের
- উপাত্ত
- ডাটাবেস
- DATETIME
- গভীর
- গভীর জ্ঞানার্জন
- DeepMind
- ডিগ্রী
- দাবি
- নির্ভর করে
- স্থাপন
- মোতায়েন
- মোতায়েন
- বিস্তৃতি
- নির্ধারণ
- নির্ধারিত
- উন্নত
- উন্নয়নশীল
- উন্নয়ন
- যন্ত্র
- কঠিন
- প্রদর্শন
- do
- না
- না
- ডাউনলোড
- ড্রাইভ
- ওষুধের
- সময়
- প্রতি
- সহজ
- প্রভাব
- প্রভাব
- সম্ভব
- শেষ
- শেষপ্রান্ত
- নিশ্চিত
- পরিবেশ
- উদাহরণ
- উদাহরণ
- ব্যয়বহুল
- অভিজ্ঞতা
- পরীক্ষা-নিরীক্ষা
- বহিরাগত
- অত্যন্ত
- মুখ
- পরিবার
- দ্রুত
- কয়েক
- কম
- ক্ষেত্র
- ব্যক্তিত্ব
- ফাইল
- নথি পত্র
- আবিষ্কার
- জরিমানা
- প্রথম
- অনুসরণ
- জন্য
- বিন্যাস
- বন্ধুদের
- থেকে
- ক্রিয়া
- অধিকতর
- উত্পাদন করা
- উত্পন্ন
- GitHub
- বিশ্বব্যাপী
- Green
- পথপ্রদর্শন
- আছে
- he
- স্বাস্থ্যসেবা
- ভারী
- সাহায্য
- সাহায্য
- উচ্চ
- তার
- নিমন্ত্রণকর্তা
- কিভাবে
- কিভাবে
- যাহোক
- এইচটিএমএল
- HTTPS দ্বারা
- নাভি
- জড়িয়ে আছে
- অভিন্ন
- পরিচয়
- if
- ভাবমূর্তি
- আমদানি
- গুরুত্বপূর্ণ
- উন্নত করা
- উন্নত
- in
- অন্তর্ভুক্ত
- ইঙ্গিত
- ইঙ্গিত
- তথ্য
- পরিবর্তন করা
- ইনপুট
- সূক্ষ্মদৃষ্টি
- অর্ন্তদৃষ্টি
- উদাহরণ
- পরিবর্তে
- সম্পূর্ণ
- গর্ভনাটিকা
- পারস্পরিক ক্রিয়ার
- ইন্টারেক্টিভ
- ইন্টারেক্টিভ
- মধ্যে
- IT
- এর
- গবেষণাগার
- ভাষা
- বড়
- নেতৃত্ব
- শিক্ষা
- ত্যাগ
- লাইব্রেরি
- লাইব্রেরি
- জীবন
- জীবন বিজ্ঞান
- আলো
- মত
- সম্ভবত
- বোঝা
- অবস্থান
- খুঁজে দেখো
- মেশিন
- মেশিন লার্নিং
- করা
- তৈরি করে
- পরিচালক
- অনেক
- ম্যাচ
- মে..
- ঔষধ
- সম্মেলন
- পদ্ধতি
- পদ্ধতি
- ছন্দোময়
- মিশিগান
- লক্ষ লক্ষ
- মিনিট
- ML
- এমএলওএস
- মোড
- মডেল
- মডেল
- পরিবর্তন
- আণবিক
- অধিক
- অনেক
- নাম
- প্রয়োজনীয়
- প্রয়োজন
- NIH এ
- নোটবই
- উপন্যাস
- সংখ্যা
- বস্তু
- অপ্রচলিত
- of
- on
- ONE
- কেবল
- পরিচালনা
- সর্বোচ্চকরন
- পছন্দ
- or
- কমলা
- OS
- অন্যান্য
- আমাদের
- আউটপুট
- বাহিরে
- পরামিতি
- কামুক
- রোগী
- রোগীদের
- নিদর্শন
- নির্ভুল
- কর্মক্ষমতা
- পাইপলাইন
- Plato
- প্লেটো ডেটা ইন্টেলিজেন্স
- প্লেটোডাটা
- দয়া করে
- পোস্ট
- সম্ভাব্য
- ভবিষ্যদ্বাণী করা
- পূর্বাভাস
- পূর্বাভাসের
- ভবিষ্যদ্বাণী
- ভবিষ্যতবাণী
- প্রস্তুত করা
- পূর্বে
- প্রক্রিয়া
- প্রসেস
- প্রোটিন
- প্রোটিন
- প্রদান
- উপলব্ধ
- প্রদানের
- পাইথন
- পাইটার্চ
- Qi
- দ্রুত
- দ্রুত
- দ্রুত বিকশিত ক্ষেত্র
- প্রকৃত সময়
- সুপারিশ করা
- লাল
- এলাকা
- সংশ্লিষ্ট
- নির্ভর করা
- চিত্রিত করা
- প্রতিনিধিত্বমূলক
- প্রতিনিধিত্ব করে
- প্রয়োজন
- গবেষণা
- গবেষকরা
- ফল
- সারিটি
- চালান
- দৌড়
- ঋষি নির্মাতা
- সেজমেকার ইনফারেন্স
- একই
- সংরক্ষণ করুন
- মাপযোগ্য
- আরোহী
- বিজ্ঞান
- বিজ্ঞান
- স্কোর
- স্কোরিং
- স্ক্রিপ্ট
- সেকেন্ড
- নিরাপদ
- দেখ
- জ্যেষ্ঠ
- ক্রম
- সেবা
- সেট
- বিভিন্ন
- শেয়ার
- সে
- উচিত
- প্রদর্শনী
- প্রদর্শিত
- পাশ
- অনুরূপ
- সহজতর করা
- একক
- সমাধান
- সলিউশন
- সমাধান
- কিছু
- স্থান-সংক্রান্ত
- বিশেষজ্ঞ
- বর্ণালী
- স্পীড
- শুরু
- রাষ্ট্র-এর-শিল্প
- ধাপ
- কাঠামোগত
- গঠন
- চিত্রশালা
- অধ্যয়ন
- এমন
- উপযুক্ত
- সমর্থন
- উদ্বর্তন
- গ্রহণ করা
- লাগে
- লক্ষ্যমাত্রা
- কাজ
- টীম
- দল
- কারিগরী
- পরীক্ষা
- চেয়ে
- যে
- সার্জারির
- তাদের
- তাহাদিগকে
- তারপর
- সেখানে।
- এইগুলো
- এই
- তিন
- ত্রিমাত্রিক
- সময়
- সময় অপগিত হয় এমন
- বার
- থেকে
- টোকেনাইজ
- টোকেনাইজড
- টুল
- সরঞ্জাম
- মশাল
- প্রশিক্ষিত
- প্রশিক্ষণ
- ট্রান্সফরমার
- পরিবহন
- চিকিৎসা
- দুই
- আদর্শ
- ধরনের
- বোঝা
- বোধশক্তি
- বিশ্ববিদ্যালয়
- অসদৃশ
- us
- ব্যবহার
- ব্যবহৃত
- ব্যবহার
- যাচাই করুন
- খুব
- চেক
- কল্পনা
- W
- চেয়েছিলেন
- উপায়..
- we
- ওয়েব
- ওয়েব সার্ভিস
- সুপরিচিত
- কি
- উইকিপিডিয়া
- ইচ্ছা
- সঙ্গে
- ওয়ার্ডপ্রেস
- হয়া যাই ?
- কাজ
- would
- এক্সরে
- আপনি
- আপনার
- zephyrnet