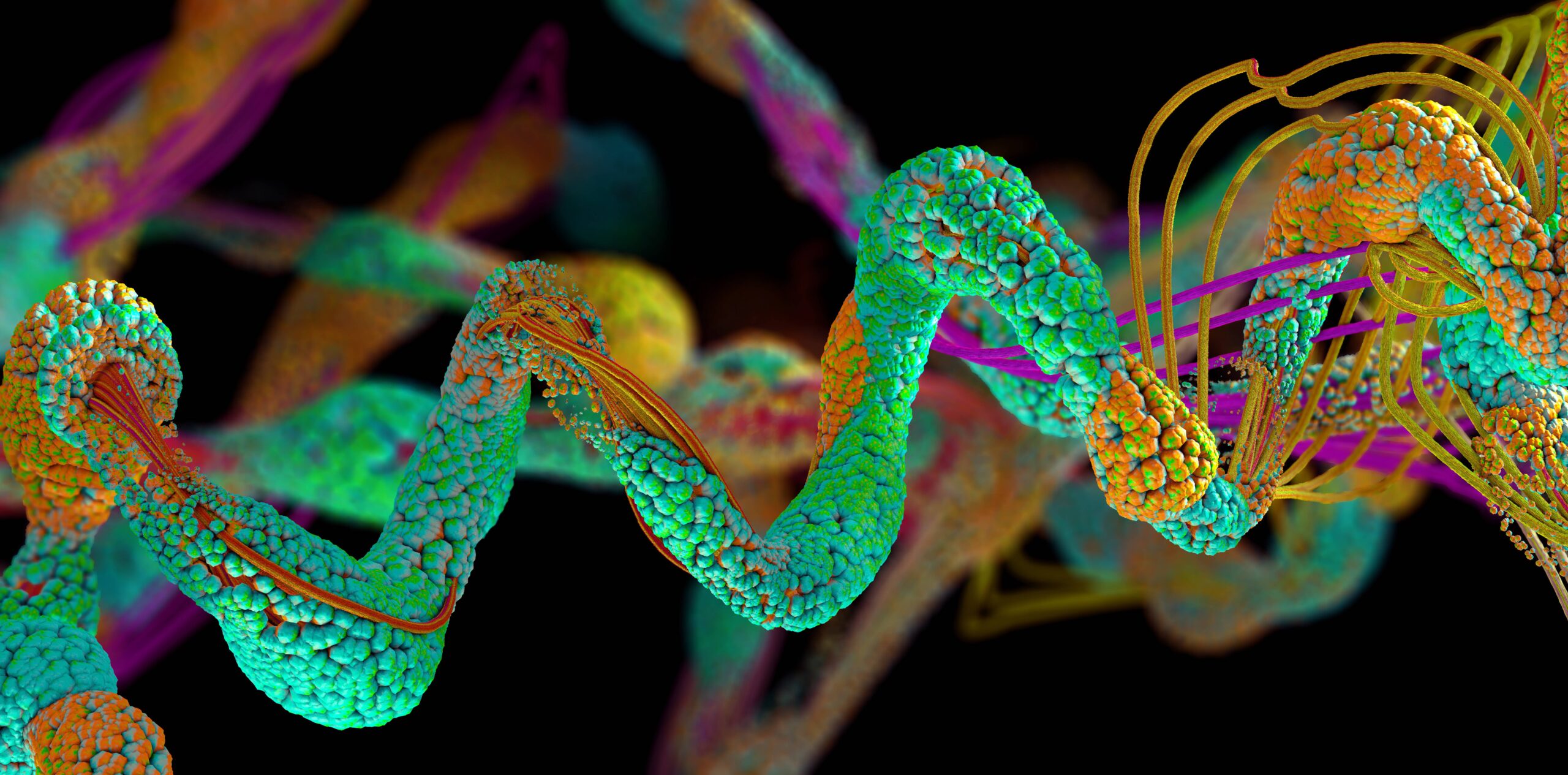
Den AI-drevne proteinfoldningsmodel AlphaFold har forudsagt mere end 200 millioner proteiner, næsten alle sådanne strukturer kendt af videnskaben, sagde DeepMind torsdag.
Proteiner er komplekse biologiske molekyler produceret i levende organismer fra instruktioner gemt i DNA. Lavet af så mange som 20 typer aminosyrer, udfører disse nanoskalakæder vitale cellulære opgaver for at udføre alle mulige kropsfunktioner. At kende den tredimensionelle form af proteiner er vigtigt, da dets fysiske struktur giver hints om, hvordan det opfører sig, og hvilket formål det tjener, hvilket hjælper os med at gøre ting som at udvikle lægemidler og skabe copycat-proteiner til dem, der mangler dem.
Nogle proteiner er nyttige, såsom dem, der er involveret i at fordøje mad, mens andre kan være skadelige, såsom dem, der er involveret i væksten af tumorer. Det er imidlertid svært at finde ud af deres komplicerede vridende former. Molekylærbiologer kan bruge år på at udføre eksperimenter for at dechifrere et proteins struktur, og AlphaFold kan gøre dette på få minutter, afhængigt af hvor stort molekylet er, ud fra aminosyresammensætningen.
AlphaFold blev trænet på hundredtusindvis af kendte proteinstrukturer og lærte forholdet mellem de indgående aminosyrer og de endelige overordnede former. Givet en vilkårlig input-aminosyresekvens kan modellen forudsige en 3D-proteinstruktur. Nu har modellen forudsagt næsten alle proteinstrukturer kendt af videnskaben.
I samarbejde med European Bioinformatics Institute har DeepMind udvidet sit AlphaFold Protein Structure Database at indeholde over 200 millioner 3D-former af proteiner fra dyr til planter, bakterier til vira – en stigning på mere end 200 gange fra næsten en million molekyler til mindst 200 millioner molekyler på bare et år.
"Vi håbede, at denne banebrydende ressource ville hjælpe med at accelerere videnskabelig forskning og opdagelse globalt, og at andre hold kunne lære af og bygge videre på de fremskridt, vi gjorde med AlphaFold for at skabe yderligere gennembrud," Demis Hassibis, DeepMinds medstifter og administrerende direktør, sagde i en erklæring torsdag.
”Det håb er blevet til virkelighed langt hurtigere, end vi havde turdet drømme om. Blot tolv måneder senere er AlphaFold blevet tilgået af mere end en halv million forskere og brugt til at accelerere fremskridt med vigtige problemer i den virkelige verden lige fra plastikforurening til antibiotikaresistens."
Registret har bedt DeepMind om yderligere kommentar.
AlphaFold har også vist et stort potentiale for at designe nye lægemidler. Strukturerne hjælper videnskabsmænd med at finde ud af kemiske forbindelser, der kan binde sig til målproteiner for at behandle eller forhindre dem i at udføre patologiske funktioner. Virksomheder, herunder Insilco Medicine har eksperimenteret med modellen til at opdage nye lægemidler; Det fortalte administrerende direktør Alex Zhavoronkov Registret at processen er meget mere kompliceret, end du skulle tro, og involverer flere trin.
Det er ikke klart, hvor fuldstændig nøjagtige AlphaFolds forudsigelser er. Et proteins båndlignende struktur ændrer ofte form, når det interagerer med et lægemiddel, noget AlphaFold ikke kan hjælpe videnskabsmænd med, da det ikke er trænet i det. Zhavoronkov sagde, at modellen er et "temmelig bemærkelsesværdigt gennembrud", men var på vagt over for al hypen.
"Indtil vi ser en struktur for et nyt mål i en stor sygdom opnået via AlphaFold uden yderligere eksperimenter, et molekyle designet ved hjælp af AI - eller andre metoder - ved hjælp af denne forudsagte struktur, syntetiseret og testet hele vejen og derefter publiceret i et højt tidsskrift – [det kan vi] så fejre.
Big pharma ønsker at se molekyler designet ved hjælp af AI-værktøjer som AlphaFold faktisk testet i mus og mennesker. "Rene algoritmiske præstationer er ikke værdifulde for farmaselskaberne og især for patienterne," tilføjede Zhavoronkov.
Fabio Urbina, seniorforsker hos Collaboration Pharmaceuticals, en startup, der bruger maskinlæringsalgoritmer til at udvikle lægemidler til sjældne genetiske sygdomme, sagde, at AlphaFold endnu ikke har vist sig at være nyttig i hans forskning. Urbina bruger en anden teknik og fokuserer mere på strukturen af et potentielt nyt lægemiddel frem for et målprotein.
Det skal endnu ses, om proteinstrukturerne vil være nyttige nok … til at hjælpe os med at opdage nye potentielle lægemidler til sjældne sygdomme
“Dette er af et par grunde; proteinstrukturerne for en masse lægemiddelmål var ofte ikke let tilgængelige for forskere at bruge, og proteininformation så ikke ud til at hjælpe de tidlige maskinlæringsmodeller med at forbedre deres forudsigelsesevne med en betydelig margin,” fortalte han. Registret.
"Jeg er forsigtigt optimistisk over, at AlphaFold i det væsentlige har 'løst' det første problem, men det er endnu uvist, om proteinstrukturerne vil være nyttige nok til vores downstream-anvendelse af forbedring af maskinlæringsforudsigelsesevne for at hjælpe os med at opdage nye potentielle lægemidler for sjældne sygdomme. Men vi har i stigende grad set proteinstrukturel information taget i betragtning som en del af nyere maskinlæringsmetoder, og vi har overvejet at gøre det samme."
At lave en database med næsten alle kendte proteinstrukturer tilgængelig, som DeepMind har lovet, betyder, at flere forskere vil have ressourcerne til at eksperimentere og bygge mere kraftfulde AI-modeller, sagde Urbina. "Jeg er forsigtigt optimistisk, men med hele biblioteket af proteinstrukturer til rådighed, vil jeg sige, at der er en god chance for, at AlphaFold-strukturer vil blive inkorporeret i nogle af vores maskinlæringsmodeller og i sidste ende kan hjælpe os med at opdage nye terapeutiske midler. ” ®
- AI
- ai kunst
- ai kunst generator
- en robot
- kunstig intelligens
- certificering af kunstig intelligens
- kunstig intelligens i banksektoren
- kunstig intelligens robot
- kunstig intelligens robotter
- software til kunstig intelligens
- blockchain
- blockchain konference ai
- coingenius
- samtale kunstig intelligens
- kryptokonference ai
- dalls
- dyb læring
- du har google
- machine learning
- plato
- platon ai
- Platon Data Intelligence
- Platon spil
- PlatoData
- platogaming
- skala ai
- syntaks
- Registret
- zephyrnet