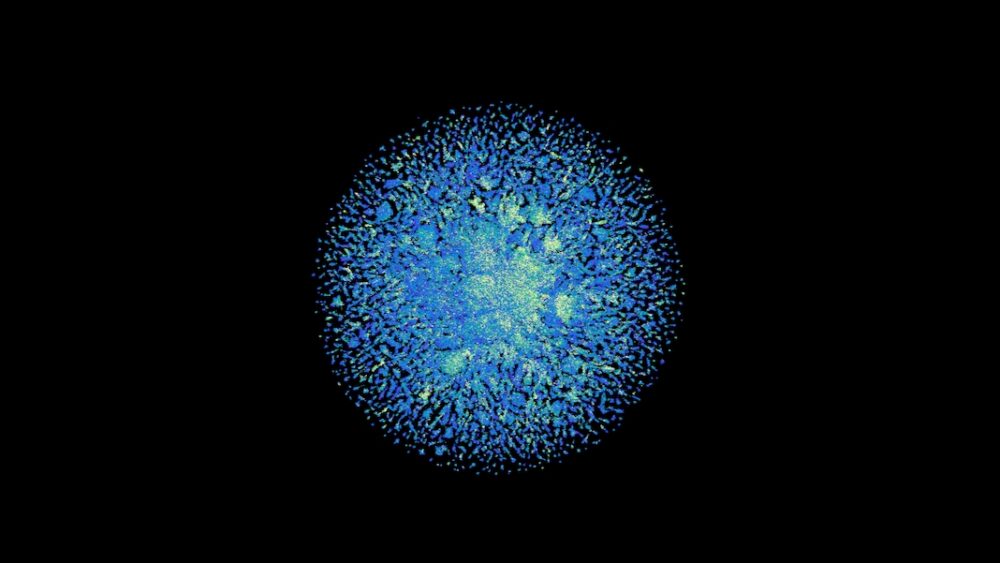
Das Rennen um die Lösung aller Proteinstrukturen hat gerade einen weiteren Technologiegiganten begrüßt: Meta AI.
Als Forschungsableger von Meta, bekannt für Facebook und Instagram, trat das Team mit einem ehrgeizigen Ziel in die Szene der Proteinformvorhersage ein: die „dunkle Materie“ des Proteinuniversums zu entschlüsseln. Diese Proteine, die oft in Bakterien, Viren und anderen Mikroorganismen vorkommen, sind in unserer alltäglichen Umgebung zu finden, sind aber für die Wissenschaft völlige Rätsel.
„Das sind die Strukturen, über die wir am wenigsten wissen. Dies sind unglaublich mysteriöse Proteine. Ich denke, sie bieten das Potenzial für großartige Einblicke in die Biologie“, sagte Seniorautor Dr. Alexander Rives zu Natur.
Mit anderen Worten, sie sind eine Fundgrube der Inspiration für die Biotechnologie. In ihren geheimnisvollen Formen sind Schlüssel zum Entwerfen verborgen effiziente Biokraftstoffe, Antibiotika, Enzyme, oder auch ganz neue Organismen. Die Daten aus Proteinvorhersagen wiederum könnten KI-Modelle weiter trainieren.
Das Herzstück von Metas neuer KI namens ESMFold ist ein großes Sprachmodell. Es mag vertraut klingen. Diese maschinellen Lernalgorithmen haben mit dem Rockstar-Chatbot ChatGPT die Welt im Sturm erobert. Bekannt für seine Fähigkeit, schöne Essays, Gedichte und Texte mit einfachen Eingabeaufforderungen zu erstellen, ist ChatGPT – und das kürzlich eingeführte GPT-4– werden mit Millionen von öffentlich zugänglichen Texten trainiert. Schließlich lernt die KI, Buchstaben und Wörter vorherzusagen und sogar ganze Absätze zu schreiben und, im Fall von Bings ähnlichem Chatbot, zu halten Gespräche das wird manchmal etwas nervig.
Die neue Studie, veröffentlicht Wissenschaft, verbindet das KI-Modell mit der Biologie. Proteine bestehen aus 20 „Buchstaben“. Dank der Evolution hilft die Buchstabenfolge dabei, ihre endgültigen Formen zu erzeugen. Wenn große Sprachmodelle die 26 Buchstaben des englischen Alphabets leicht in zusammenhängende Botschaften umwandeln können, warum funktionieren sie dann nicht auch für Proteine?
Spoiler: Sie tun es. ESM-2 hat in nur zwei Wochen mit 600 Grafikprozessoren (GPUs) rund 2,000 Millionen Proteinstrukturvorhersagen durchgesprengt. Im Vergleich zu früheren Versuchen hat die KI den Prozess bis zu 60-mal schneller gemacht. Die Autoren haben jede Struktur in den ESM Metagenomic Atlas aufgenommen, den Sie erkunden können hier.
Für Dr. Alfonso Valencia vom Barcelona National Supercomputing Centre (BCS), der nicht an der Arbeit beteiligt war, ist die Schönheit der Verwendung großer Sprachsysteme ein „konzeptionelle Einfachheit.“ Mit der Weiterentwicklung kann die KI „die Struktur nicht natürlicher Proteine vorhersagen und das bekannte Universum über das hinaus erweitern, was evolutionäre Prozesse erforscht haben“.
Reden wir über Evolution
ESMFold folgt einer einfachen Richtlinie: Die Reihenfolge sagt die Struktur voraus.
Gehen wir zurück. Proteine bestehen aus 20 Aminosäuren – jede ein „Buchstabe“ – und sind wie stachelige Perlen an einer Schnur aufgereiht. Unsere Zellen formen sie dann zu zarten Merkmalen: Einige sehen aus wie zerknitterte Bettlaken, andere wie eine wirbelnde Zuckerstange oder lose Bänder. Die Proteine können sich dann aneinander klammern, um ein Multiplex zu bilden – zum Beispiel einen Tunnel, der die Gehirnzellmembran durchquert, der ihre Aktionen steuert und wiederum steuert, wie wir denken und uns erinnern.
Wissenschaftler wissen seit langem, dass Aminosäurebuchstaben die endgültige Struktur eines Proteins formen. Ähnlich wie Buchstaben oder Zeichen in einer Sprache ergeben nur bestimmte aneinandergereiht einen Sinn. Im Fall von Proteinen machen diese Sequenzen sie funktionsfähig.
„Die biologischen Eigenschaften eines Proteins beschränken die Mutationen auf seine Sequenz, die durch die Evolution ausgewählt wird“, sagten die Autoren.
Ähnlich wie verschiedene Buchstaben im Alphabet zusammenlaufen, um Wörter, Sätze und Absätze zu bilden, ohne wie komplettes Kauderwelsch zu klingen, tun die Proteinbuchstaben dasselbe. Es gibt eine Art „evolutionäres Wörterbuch“, das dabei hilft, Aminosäuren zu Strukturen anzuordnen, die der Körper begreifen kann.
„Die Logik der Abfolge von Aminosäuren in bekannten Proteinen ist das Ergebnis eines Evolutionsprozesses, der dazu geführt hat, dass sie die spezifische Struktur haben, mit der sie eine bestimmte Funktion erfüllen“, sagte Valencia.
Mr. AI, machen Sie mir ein Protein
Das relativ begrenzte Wörterbuch des Lebens ist tolle Neuigkeiten für große Sprachmodelle.
Diese KI-Modelle durchforsten leicht verfügbare Texte, um zu lernen und Vorhersagen über das nächste Wort zu erstellen. Das Endergebnis, wie in GPT-3 und ChatGPT zu sehen, sind auffallend natürliche Gespräche und fantastische künstlerische Bilder.
Meta AI verwendete das gleiche Konzept, schrieb aber das Spielbuch für Proteinstrukturvorhersagen um. Anstatt den Algorithmus mit Texten zu füttern, gaben sie den Programmsequenzen bekannter Proteine.
Das KI-Modell – genannt Transformer-Protein-Sprachmodell – erlernte die allgemeine Architektur von Proteinen unter Verwendung von bis zu 15 Milliarden „Einstellungen“. Es sah insgesamt rund 65 Millionen verschiedene Proteinsequenzen.
Im nächsten Schritt versteckte das Team bestimmte Buchstaben vor der KI und forderte sie auf, die Lücken zu füllen. In welchen Mengen zur automatischen Vervollständigung lernte das Programm schließlich, wie sich verschiedene Aminosäuren miteinander verbinden (oder abstoßen). Am Ende hat die KI ein intuitives Verständnis der evolutionären Proteinsequenzen entwickelt – und wie sie zusammenarbeiten, um funktionelle Proteine herzustellen.
In das Unbekannte
Als Machbarkeitsnachweis testete das Team ESMFold mit zwei bekannten Testsets. Einer, CAMEO, umfasste fast 200 Strukturen; das andere, CASP14, hat 51 öffentlich freigegebene Proteinformen.
Insgesamt „bietet die KI eine hochmoderne Strukturvorhersagegenauigkeit“, so das Team, „die die Leistung von AlphaFold2 bei mehr als der Hälfte der Proteine erreicht“. Es bekämpft auch zuverlässig große Proteinkomplexe – zum Beispiel die Kanäle auf Neuronen, die ihre Aktionen steuern.
Das Team ging dann mit seiner KI einen Schritt weiter und wagte sich in die Welt der Metagenomik.
Metagenome sind das, wonach sie klingen: ein Sammelsurium von DNA-Material. Normalerweise stammen diese aus Umweltquellen wie dem Schmutz unter Ihren Füßen, Meerwasser oder sogar normalerweise unwirtlichen Thermalquellen. Die meisten Mikroben können nicht künstlich in Labors gezüchtet werden, aber einige haben Superkräfte wie die Widerstandsfähigkeit gegen vulkanische Hitze, was sie zu einer biologischen dunklen Materie macht, die noch erforscht werden muss.
Zum Zeitpunkt der Veröffentlichung des Papiers hatte die KI über 600 Millionen dieser Proteine vorhergesagt. Mit der neuesten Version liegt die Zahl jetzt bei über 700 Millionen. Die Vorhersagen kamen schnell und wütend in ungefähr zwei Wochen. Im Gegensatz dazu dauerten frühere Modellierungsversuche für nur ein einzelnes Protein bis zu 10 Minuten.
Etwa ein Drittel der Proteinvorhersagen waren von hoher Zuverlässigkeit, mit genügend Details, um in die atomare Ebene hineinzuzoomen. Da die Proteinvorhersagen ausschließlich auf ihren Sequenzen basierten, tauchten Millionen von „Aliens“ auf – Strukturen, die mit nichts in etablierten oder zuvor getesteten Datenbanken zu vergleichen waren.
„Es ist interessant, dass mehr als 10 Prozent der Vorhersagen für Proteine gelten, die keine Ähnlichkeit mit anderen bekannten Proteinen aufweisen“, sagte Valencia. Dies könnte auf die Magie von Sprachmodellen zurückzuführen sein, die weitaus flexibler bei der Erforschung – und potenziellen Generierung – bisher unbekannter Sequenzen sind, aus denen funktionelle Proteine bestehen. „Dies ist ein neuer Raum für das Design von Proteinen mit neuen Sequenzen und biochemischen Eigenschaften mit Anwendungen in der Biotechnologie und Biomedizin“, sagte er.
Beispielsweise könnte ESMFold möglicherweise dabei helfen, die Folgen von Änderungen einzelner Buchstaben in einem Protein herauszufinden. Diese scheinbar gutartigen Änderungen, die als Punktmutationen bezeichnet werden, richten Chaos im Körper an und verursachen verheerende metabolische Syndrome, Sichelzellenanämie und Krebs. Eine schlanke, mittlere und relativ einfache KI bringt Ergebnisse in ein durchschnittliches biomedizinisches Forschungslabor, während die Vorhersagen der Proteinform dank der Geschwindigkeit der KI hochskaliert werden.
Abgesehen von der Biomedizin besteht eine weitere faszinierende Idee darin, dass Proteine helfen können, große Sprachmodelle auf eine Weise zu trainieren, wie es Texte nicht können. Wie Valencia erklärte: „Einerseits sind Proteinsequenzen häufiger als Texte, haben definiertere Größen und einen höheren Grad an Variabilität. Andererseits haben Proteine eine starke innere „Bedeutung“ – das heißt, eine starke Beziehung zwischen Sequenz und Struktur, eine Bedeutung oder Kohärenz, die in Texten viel diffuser ist“, wodurch die beiden Felder in einer virtuosen Rückkopplungsschleife überbrückt werden.
Bild-Kredit: Meta-KI
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- Platoblockkette. Web3-Metaverse-Intelligenz. Wissen verstärkt. Hier zugreifen.
- Quelle: https://singularityhub.com/2023/03/21/metas-new-ai-is-digging-into-the-most-mysterious-proteins-on-earth/
- :Ist
- $UP
- 000
- 10
- a
- Fähigkeit
- LiveBuzz
- Genauigkeit
- Aktionen
- AI
- Udo
- Algorithmus
- Algorithmen
- Alphabet
- ehrgeizig
- Beträge
- und
- Ein anderer
- Anwendungen
- Architektur
- SIND
- künstlerisch
- AS
- At
- Versuche
- Autor
- Autoren
- verfügbar
- durchschnittlich
- Bakterien
- Barcelona
- basierend
- BE
- Denken Sie
- schön
- Die Schönheit
- weil
- zwischen
- Beyond
- Milliarde
- Biologie
- biomedizinische
- Biotechnologie
- Körper
- Gehirn
- Brücken
- Überbrückung
- Brings
- bauen
- by
- namens
- CAN
- Krebs
- Häuser
- verursacht
- Die Zellen
- Center
- sicher
- Änderungen
- Kanäle
- Zeichen
- Chatbot
- ChatGPT
- KOHÄRENT
- wie die
- verglichen
- abschließen
- begreifen
- konzept
- Vertrauen
- Vernetz Dich
- Folgen
- Kontrast
- Smartgeräte App
- Steuerung
- konvergieren
- Gespräche
- könnte
- erstellen
- Kredit
- Dunkel
- Dunkle Materie
- technische Daten
- Datenbanken
- Entziffern
- definiert
- Grad
- Design
- Entwerfen
- Detail
- verheerend
- Entwicklung
- anders
- Frau
- synchronisiert
- jeder
- leicht
- Englisch
- genug
- Ganz
- Umwelt-
- Umgebungen
- etablierten
- Sogar
- schließlich
- Jedes
- jeden Tag
- Evolution
- Beispiel
- Ausbau
- erklärt
- ERKUNDEN
- Erkundet
- vertraut
- faszinierend
- FAST
- beschleunigt
- Eigenschaften
- Feedback
- Fütterung
- Feet
- Felder
- füllen
- Finale
- flexibel
- folgt
- Aussichten für
- unten stehende Formular
- gebildet
- gefunden
- für
- Funktion
- funktional
- weiter
- weitere Entwicklung
- Allgemeines
- erzeugen
- Riese
- Kundenziele
- GPUs
- greifen
- Grafik
- groß
- gewachsen
- Hälfte
- Pflege
- Haben
- Herz
- Hilfe
- hilft
- versteckt
- GUTE
- höher
- Ultraschall
- HTML
- HTTPS
- i
- Idee
- Bilder
- in
- unglaublich
- Einblick
- Inspiration
- interessant
- intern
- intuitiv
- beteiligt
- IT
- SEINE
- Tasten
- Wissen
- bekannt
- Labor
- Labs
- Sprache
- grosse
- neueste
- neueste Erscheinung
- LERNEN
- gelernt
- lernen
- geführt
- Gefällt mir
- Limitiert
- Lang
- aussehen
- aussehen wie
- Lounge
- Maschine
- Maschinelles Lernen
- gemacht
- Magie
- um
- Making
- Ihres Materials
- Materie
- Bedeutung
- Nachrichten
- Meta
- könnte
- Million
- Millionen
- Minuten
- Modell
- für
- mehr
- vor allem warme
- geheimnisvoll
- National
- Natürliche
- Natur
- fast
- Neuronen
- Neu
- News
- weiter
- Normalerweise
- of
- bieten
- on
- EINEM
- Andere
- Anders
- Gesamt-
- Papier
- besondere
- Prozent
- Ausführen
- Leistung
- Plato
- Datenintelligenz von Plato
- PlatoData
- Points
- Potenzial
- möglicherweise
- vorhersagen
- vorhergesagt
- Prognose
- Prognosen
- sagt voraus,
- früher
- vorher
- Prozessdefinierung
- anpassen
- Verarbeitung
- Programm
- Beweis
- Proof of Concept
- immobilien
- Proteine
- Proteine
- veröffentlicht
- setzen
- Rennen
- lieber
- Beziehung
- verhältnismäßig
- Release
- merken
- Forschungsprojekte
- Folge
- Die Ergebnisse
- Rockstar
- rund
- Said
- gleich
- Skalieren
- Skalierung
- Szene
- Wissenschaft
- ausgewählt
- Senior
- Sinn
- Reihenfolge
- Sets
- Form
- Formen
- ähnlich
- Einfacher
- Single
- Größen
- LÖSEN
- einige
- Klingen
- Quellen
- Raumfahrt
- spezifisch
- Geschwindigkeit
- State-of-the-art
- Schritt
- Sturm
- stark
- Struktur
- Studie
- so
- Supercomputing
- Systeme und Techniken
- Reden
- Team
- Tech
- Tech-Riese
- Test
- Vielen Dank
- zur Verbesserung der Gesundheitsgerechtigkeit
- Das
- die Welt
- ihr
- Sie
- Thermal-
- Diese
- Dritte
- Durch
- Zeit
- mal
- zu
- gemeinsam
- Training
- trainiert
- WENDE
- letzte
- für
- Verständnis
- Bereiche
- Universum
- Viren
- Weg..
- Wochen
- begrüßt
- bekannt
- Was
- welche
- während
- WHO
- mit
- ohne
- Word
- Worte
- Arbeiten
- zusammenarbeiten
- weltweit wie ausgehandelt und gekauft ausgeführt wird.
- schreiben
- Du
- Ihr
- Zephyrnet
- Zoom