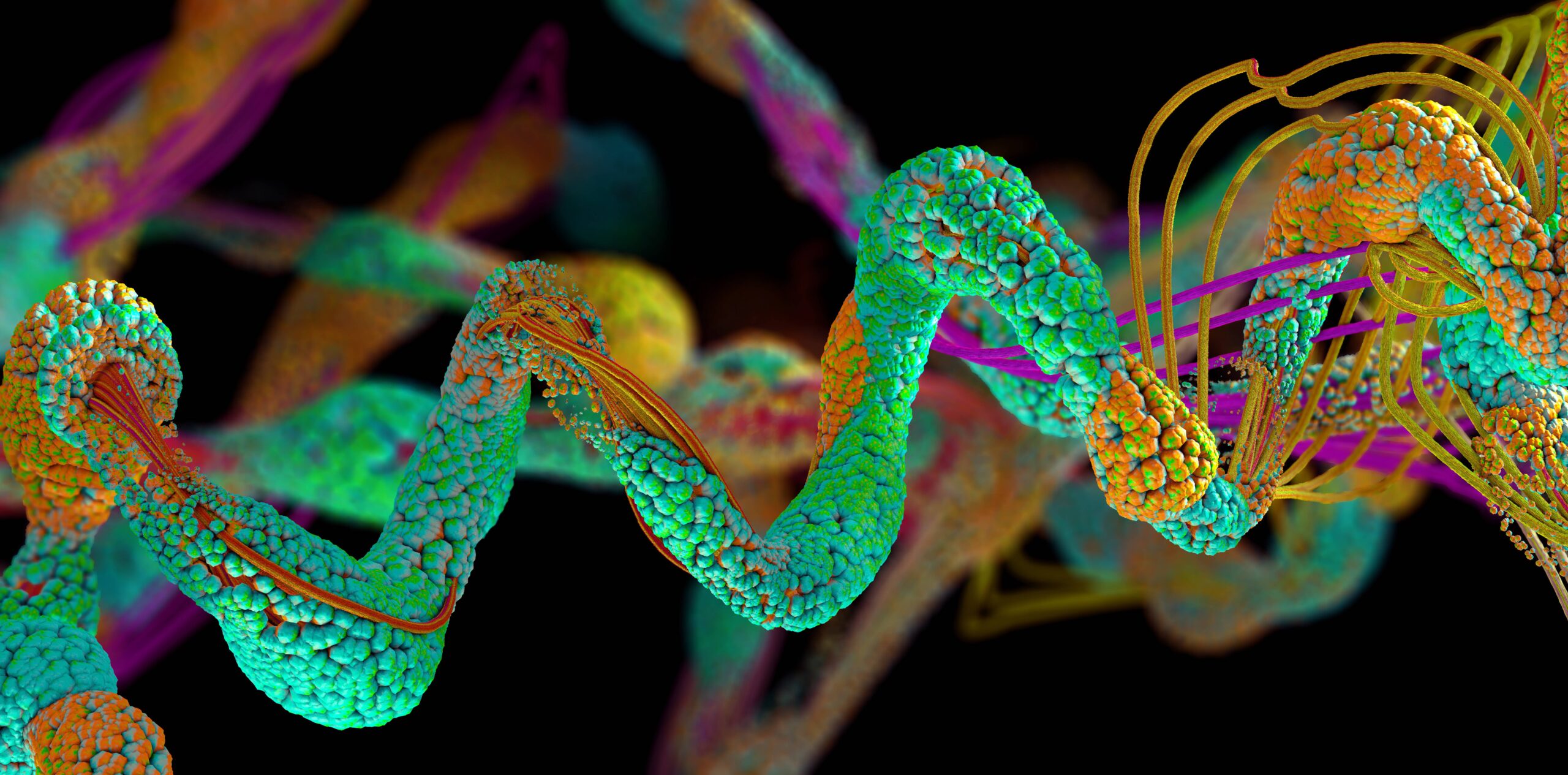
Das KI-gestützte Proteinfaltungsmodell AlphaFold hat mehr als 200 Millionen Proteine vorhergesagt, fast alle der Wissenschaft bekannten Strukturen dieser Art, sagte DeepMind am Donnerstag.
Proteine sind komplexe biologische Moleküle, die in lebenden Organismen aus in der DNA gespeicherten Anweisungen hergestellt werden. Diese Ketten im Nanomaßstab bestehen aus bis zu 20 Arten von Aminosäuren und erfüllen lebenswichtige zelluläre Aufgaben, um alle Arten von Körperfunktionen auszuführen. Die Kenntnis der dreidimensionalen Form von Proteinen ist wichtig, da ihre physikalische Struktur Hinweise darauf gibt, wie sie sich verhält und welchem Zweck sie dient, was uns hilft, Dinge wie die Entwicklung von Medikamenten und die Herstellung von Nachahmerproteinen für diejenigen zu tun, denen sie fehlen.
Einige Proteine sind hilfreich, beispielsweise solche, die an der Verdauung von Nahrung beteiligt sind, während andere schädlich sein können, beispielsweise solche, die am Wachstum von Tumoren beteiligt sind. Es ist jedoch schwierig, ihre komplizierten, zappeligen Formen herauszufinden. Molekularbiologen können Jahre damit verbringen, Experimente durchzuführen, um die Struktur eines Proteins zu entschlüsseln, und AlphaFold kann dies in Minuten tun, je nachdem, wie groß das Molekül aus der Aminosäurezusammensetzung ist.
AlphaFold wurde an Hunderttausenden bekannter Proteinstrukturen trainiert und lernte die Beziehungen zwischen den einzelnen Aminosäuren und den endgültigen Gesamtformen. Bei einer beliebigen Eingangs-Aminosäuresequenz kann das Modell eine 3D-Proteinstruktur vorhersagen. Jetzt hat das Modell fast alle der Wissenschaft bekannten Proteinstrukturen vorhergesagt.
In Zusammenarbeit mit dem European Bioinformatics Institute hat DeepMind seine erweitert AlphaFold Proteinstrukturdatenbank über 200 Millionen 3D-Formen von Proteinen von Tieren bis Pflanzen, Bakterien bis Viren zu enthalten – eine mehr als 200-fache Steigerung von fast einer Million Molekülen auf mindestens 200 Millionen Moleküle in nur einem Jahr.
„Wir hofften, dass diese bahnbrechende Ressource dazu beitragen würde, die wissenschaftliche Forschung und Entdeckung weltweit zu beschleunigen, und dass andere Teams von den Fortschritten, die wir mit AlphaFold gemacht haben, lernen und darauf aufbauen könnten, um weitere Durchbrüche zu schaffen“, sagte Demis Hassibis, Mitbegründer und CEO von DeepMind. sagte in einer Erklärung am Donnerstag.
„Diese Hoffnung ist viel schneller Wirklichkeit geworden, als wir zu träumen gewagt hatten. Nur zwölf Monate später wurde AlphaFold von mehr als einer halben Million Forschern aufgerufen und verwendet, um den Fortschritt bei wichtigen realen Problemen zu beschleunigen, die von der Verschmutzung durch Plastik bis hin zur Antibiotikaresistenz reichen.“
Das Register hat DeepMind um weitere Kommentare gebeten.
AlphaFold hat auch ein großes Potenzial für die Entwicklung neuer Medikamente gezeigt. Die Strukturen helfen Wissenschaftlern, chemische Verbindungen herauszufinden, die an Zielproteine binden können, um diese zu behandeln oder daran zu hindern, pathologische Funktionen auszuführen. Unternehmen wie Insilco Medicine haben experimentierte mit dem Modell, um neue Medikamente zu entdecken; CEO Alex Zhavoronkov sagte Das Register dass der Prozess viel komplizierter ist, als Sie vielleicht denken, und mehrere Schritte umfasst.
Es ist nicht klar, wie genau die Vorhersagen von AlphaFold sind. Die bandartige Struktur eines Proteins ändert oft ihre Form, wenn es mit einem Medikament interagiert, etwas, bei dem AlphaFold Wissenschaftlern nicht helfen kann, da es nicht darauf trainiert ist. Zhavoronkov sagte, das Modell sei ein „ziemlich bemerkenswerter Durchbruch“, war aber angesichts des ganzen Hypes misstrauisch.
„Bis wir eine Struktur für ein neuartiges Ziel in einer großen Krankheit sehen, die über AlphaFold ohne zusätzliche Experimente erhalten wird, ein Molekül, das mit KI – oder anderen Methoden – unter Verwendung dieser vorhergesagten Struktur entworfen, synthetisiert und vollständig getestet und dann in einer hochkarätigen Zeitschrift veröffentlicht wird – [können wir] dann feiern.“
Große Pharmakonzerne möchten, dass Moleküle, die mit Hilfe von KI-Tools wie AlphaFold entwickelt wurden, tatsächlich an Mäusen und Menschen getestet werden. „Reine algorithmische Errungenschaften sind für die Pharmaunternehmen und insbesondere für die Patienten nicht wertvoll“, fügte Zhavoronkov hinzu.
Fabio Urbina, leitender Wissenschaftler bei Collaboration Pharmaceuticals, einem Startup, das maschinelle Lernalgorithmen zur Entwicklung von Medikamenten für seltene genetische Krankheiten einsetzt, sagte, AlphaFold habe sich in seiner Forschung noch nicht als nützlich erwiesen. Urbina verwendet eine andere Technik und konzentriert sich eher auf die Struktur eines potenziellen neuen Arzneimittels als auf ein Zielprotein.
Es bleibt abzuwarten, ob die Proteinstrukturen nützlich genug sein werden, … um uns bei der Entdeckung neuer potenzieller Medikamente für seltene Krankheiten zu helfen
„Das hat mehrere Gründe; Die Proteinstrukturen für viele Wirkstoffziele waren für Forscher oft nicht leicht verfügbar, und Proteininformationen schienen den frühen maschinellen Lernmodellen nicht dabei zu helfen, ihre Vorhersagekraft erheblich zu verbessern“, sagte er Das Register.
„Ich bin vorsichtig optimistisch, dass AlphaFold das erste Problem im Wesentlichen „gelöst“ hat, aber es muss sich noch herausstellen, ob die Proteinstrukturen für unsere nachgelagerte Anwendung zur Verbesserung der Vorhersagekraft des maschinellen Lernens nützlich genug sein werden, um uns bei der Entdeckung neuer potenzieller Medikamente zu helfen für seltene Erkrankungen. Wir haben jedoch zunehmend gesehen, dass Proteinstrukturinformationen im Rahmen neuerer maschineller Lernmethoden berücksichtigt werden, und wir haben darüber nachgedacht, dasselbe zu tun.“
Die Bereitstellung einer Datenbank mit fast allen bekannten Proteinstrukturen, wie DeepMind versprochen hat, bedeutet, dass mehr Wissenschaftler die Ressourcen haben werden, um zu experimentieren und leistungsfähigere KI-Modelle zu bauen, sagte Urbina. „Ich bin vorsichtig optimistisch, aber mit der gesamten verfügbaren Bibliothek an Proteinstrukturen würde ich sagen, dass es gute Chancen gibt, dass AlphaFold-Strukturen in einige unserer Modelle für maschinelles Lernen integriert werden und uns letztendlich helfen können, neuartige Therapeutika zu entdecken. ” ®
- AI
- Kunst
- KI-Kunstgenerator
- KI-Roboter
- künstliche Intelligenz
- Zertifizierung für künstliche Intelligenz
- Künstliche Intelligenz im Bankwesen
- Roboter mit künstlicher Intelligenz
- Roboter mit künstlicher Intelligenz
- Software für künstliche Intelligenz
- Blockchain
- Blockchain-Konferenz ai
- Einfallsreichtum
- dialogorientierte künstliche Intelligenz
- Krypto-Konferenz ai
- Dalls
- tiefe Lernen
- Google Ai
- Maschinelles Lernen
- Plato
- platon ai
- Datenintelligenz von Plato
- Plato-Spiel
- PlatoData
- Platogaming
- Skala ai
- Syntax
- Das Register
- Zephyrnet