Das Entwerfen eines Proteins ist ein bisschen wie das Bauen eines Schranks. Der erste Schritt ist der Aufbau des Rückgrats, das das Protein zusammenhält. Aber dann kommt der schwierige Teil: Herauszufinden, wo Scharniere am Gerüst angebracht werden müssen – das heißt, die besten „Hotspots“ zu finden – um Türen, Regale und andere Anbauteile anzubringen, die den Schrank letztendlich voll funktionsfähig machen.
In gewisser Weise haben Proteine auch Hotspots in ihre Strukturen eingebettet. Getreu ihrem Namen „funktionelle Stellen“ bilden diese faszinierenden Ecken und Winkel komplizierte Docks, an denen sich andere Proteine oder Medikamente festhalten können. Die Standorte sind von zentraler Bedeutung für die Durchführung der meisten unserer grundlegenden biologischen Prozesse. Sie sind auch eine riesige Goldmine für die Entwicklung neuer Behandlungen und Medikamente.
Das Problem? Funktionale Standorte sind schwer zu kartieren. Traditionell mussten Wissenschaftler verdächtige Bereiche auf einem Protein nacheinander mutieren – indem sie eine Aminosäure gegen eine andere austauschten – um präzise Bindungsstellen festzunageln. Wie ein Detektiv, der Hunderte von Verdächtigen überprüft, von denen es viele geben könnte, ist es äußerst mühsam.
A neue Studie in Wissenschaft stürzte das ganze Spielbuch. Unter der Leitung von Dr. David Baker von der University of Washington nutzte ein Team die „Fantasie“ einer KI, um eine Vielzahl von funktionalen Standorten von Grund auf neu zu erfinden. Es ist die „Kreativität“ eines maschinellen Verstandes in Bestform – ein Deep-Learning-Algorithmus, der den allgemeinen Bereich der funktionellen Stelle eines Proteins vorhersagt, dann aber die Struktur weiter formt.
Als Realitätscheck verwendete das Team die neue Software, um Medikamente zur Krebsbekämpfung zu entwickeln und Impfstoffe gegen häufige, wenn auch manchmal tödliche Viren zu entwickeln. In einem Fall entwickelte der digitale Verstand eine Lösung, die beim Testen in isolierten Zellen perfekt zu einem vorhandenen Antikörper gegen ein gewöhnliches Virus passte. Mit anderen Worten, der Algorithmus „bildete“ sich einen Hotspot eines viralen Proteins ein, wodurch es als Ziel für die Entwicklung neuer Behandlungen verwundbar wurde.
Der Algorithmus ist der erste Vorstoß von Deep Learning zum Aufbau von Proteinen um ihre Funktionen herum und öffnet eine Tür zu Behandlungen, die zuvor undenkbar waren. Aber die Software ist nicht auf natürliche Protein-Hotspots beschränkt. „Die Proteine, die wir in der Natur finden, sind erstaunliche Moleküle, aber entworfene Proteine können so viel mehr“, sagte Baker in einer Pressemitteilung. Der Algorithmus „macht Dinge, von denen keiner von uns gedacht hätte, dass er dazu in der Lage wäre.“
Der Protein-Hotspot
Bakers Team ist es nicht fremd, Proteine mit künstlichem Verstand vorherzusagen. Vor einigen Jahren erschütterten sie das Feld der Strukturbiologie, indem sie Rosetta veröffentlichten, eine Software, die die 3D-Struktur eines Proteins allein auf der Grundlage seiner Aminosäuresequenz vorhersagen kann. Sie kartierten außerdem Proteinkomplexe und konstruierten Protein-„Schraubendreher“ von Grund auf neu, um unerwünschte Proteininteraktionen auseinanderzuhebeln. Ende letzten Jahres veröffentlichten sie a Deep Learning-Netzwerk genannt trRosetta, ein KI-„Architekt“, der verallgemeinert, wie sich Ketten von Aminosäuren zu komplizierten Strukturen im Nanomaßstab anordnen.
Lassen Sie uns zurückgehen.
Es ist leicht, sich Proteine als fleischige, sehnige Hühnerflügel vorzustellen, in die ich beiße, während ich diesen Satz tippe. Aber auf molekularer Ebene sind sie viel eleganter. Stellen Sie sich mehrere Lego-Blöcke – Aminosäuren – vor, die von einer Schnur zusammengehalten werden. Drehen Sie es nun herum und drehen Sie die Kette, bis einige Blöcke aufeinander einrasten. Dies bildet eine filigrane Struktur, die oft einer Spirale oder zerknitterten Bettlaken ähnelt. In einigen Proteinen setzen sich diese Bausteine weiter zu Komplexen zusammen – zum Beispiel, indem sie einen Kanal herstellen, der wie eine bewachte Autobahn durch die Schutzmembran einer Zelle führt.
Proteine treiben jeden einzelnen biologischen Prozess an, oft durch eine Kaskade von Wechselwirkungen mit anderen Proteinen oder Medikamenten, die – je nach Partner – ganz unterschiedliche Konsequenzen auslösen können: Soll eine Zelle leben oder sterben? Einen potenziellen Eindringling angreifen oder zurücktreten? Mit anderen Worten, Proteine sind die Bausteine des Lebens, und durch das Analysieren ihrer Struktur können wir uns in das Leben hacken.
Hier ist die Sache: Nicht alle Teile eines Proteins sind gleich. Wenn ein Protein ein menschlicher Körper ist, sind funktionelle Stellen seine „Hände“ – wo es nach einem anderen Protein oder Medikament greift, enzymatische Reaktionen auslöst oder eindringende Krankheitserreger abwehrt. Diese direkt in die Struktur des Proteins eingebetteten Stellen sind schwer festzumachen und noch schwerer nachzubilden.
Die neue Studie ging das Problem mit einer Version von Rosetta an: Ist es einem Computer mit etwas Vorwissen möglich, sich eine Kette von Aminosäuren auszudenken, die sich auf natürliche Weise zu einer funktionellen Stelle falten?
Der Träumer und der Realist
Das Problem mag exotisch erscheinen, aber es gibt ein früheres Beispiel – auf einem anderen Gebiet. Mithilfe eines neuronalen Netzwerks erstellte OpenAI allein aus Textunterschriften eine Vielzahl von Bildern. Ein Ableger des Textgenerators Rockstar AI GPT-3, generierte der DALL·E-Algorithmus fantastische, aber realistisch aussehende Bilder basierend auf einfachen Texteingabeaufforderungen, indem er Muster aus seinem Training erkannte. „Es nimmt die tiefsten, dunkelsten Winkel Ihrer Vorstellungskraft und verwandelt sie in etwas unheimlich Bedeutsames.“ sagte Dr. Hany Farid von der UC Berkeley nach der ersten Veröffentlichung des Tools.
Der Aufbau einer funktionellen Proteinstelle ist ähnlich. Hier sind Aminosäuren die Buchstaben und die funktionelle Stelle des Proteins ist das Bild. „Die Idee ist die gleiche: Neuronale Netze können darauf trainiert werden, Muster in Daten zu erkennen. Einmal trainiert, können Sie ihm eine Eingabeaufforderung geben und sehen, ob es eine elegante Lösung generiert“, sagte Dr. Joseph Watson, einer der Hauptautoren der neuen Arbeit. Anstatt einen Roman zu schreiben, könnte der Algorithmus dabei helfen, das Leben neu zu schreiben.
Das Team begann mit einer früheren Kreation, trRosetta. Es handelt sich um ein neuronales Netzwerk, das ursprünglich entwickelt wurde, um neue Proteine auf der Grundlage von Aminosäuresequenzen zu erfinden und gleichzeitig in der Lage zu sein, ihre Struktur vorherzusagen – einige davon so fremd von natürlichen, dass das Team das Innenleben des Deep Learning als „Halluzination“ bezeichnete. Der Algorithmus schien perfekt: Er konnte sowohl die Aminosäuresequenz als auch die Struktur eines Proteins vorhersagen.
Der Schluckauf? Es hat nicht wirklich funktioniert. Im Gegensatz, das OG der Proteinstrukturvorhersage, RoseTTAFold, performte wie ein Weltmeister. Die Stärke des Algorithmus liegt in seinem Design: Modellieren jeder Aminosäure im Nanomaßstab, Bereitstellen von Koordinaten für jedes Atom. Wie das Anheften eines geografischen Standorts mit Google Maps bietet dies eine gewisse Grundwahrheit für eine Struktur, auf der eine KI weiter riffeln kann – eine Art „eingeschränkte Halluzination“.
Übersetzung? RoseTTAFold kann eine funktionale Struktur vorhersagen – spezifisch für das jeweilige Problem – und eine grobe Skizze als endgültiges Design erstellen.
Dann kam ein weiterer cleverer Trick, der als „Inpainting“ bezeichnet wurde. Hier versteckte das Team Teile der Proteinsequenz oder -struktur. Die Software musste lernen, Informationen aus einer lauten Funküberwachung zu entschlüsseln, bei der man nur die ersten paar Wörter hört, aber versucht, ihre Bedeutung zu verstehen, indem man die Lücken ausfüllt. RoseTTAFold ging das „Problem der Wiederherstellung fehlender Informationen“ mit Begeisterung an, indem es sowohl Aminosäuresequenzen als auch Strukturen automatisch vervollständigte, um eine bestimmte funktionelle Region mit hoher Genauigkeit zu konstruieren.
RoseTTAFold kann die Probleme beim Aufbau von Aminosäuresequenzen angehen und gleichzeitig ein Rückgrat für die Site generieren. Es ist, als würde man Wörter zu Papier bringen: Der Schreiber achtet darauf, dass jeder Buchstabe an der richtigen Stelle steht, und prüft gleichzeitig, ob Grammatik und Bedeutung Sinn ergeben.
Hinterfragen der Natur der Realität
Das Team stellte seine neue Kreation auf die Probe und entwickelte mehrere Arzneimittel- und Impfstoffdesigns, die möglicherweise Viren und Krebs bekämpfen oder bei eisenarmen Gesundheitsproblemen helfen könnten.
Für den Hauptautor Dr. Jue Wang wurde der Algorithmus unerwartet relevant. Während der Arbeit an dem Projekt wurde sein zweijähriger Sohn wegen einer Lungeninfektion durch RSV (Respiratory Syncytial Virus) in die Notaufnahme eingeliefert – ein Virus, das normalerweise erkältungsähnliche Symptome zeigt, aber für Jung und Alt tödlich sein kann Alten.
Zu dieser Zeit verwendete Wang den Algorithmus, um neue Behandlungen zu entwickeln, darunter potenzielle Stellen auf RSV, um Impfstoffe und Medikamente weiter zu testen. Es ist eine relativ gut abgebildete Struktur. Die Software halluzinierte Entwürfe, die zwei Stellen rekapitulierten, an denen Impfstoffe möglicherweise binden könnten. Tests mit halluzinierten Proteinen, die in Bakterien rekonstruiert wurden, griffen schnell auf vorhandene Antikörper zu – ein Zeichen dafür, dass sie funktionsfähig sind und dass der Deep-Learning-Ansatz funktioniert.
Der Vorfall „machte mir klar, dass selbst die ‚Test'-Probleme, an denen wir arbeiteten, eigentlich ziemlich bedeutsam waren“, sagte Wang.
In mehreren zusätzlichen Tests entwarf das Team funktionelle Stellen für ein Enzym, proteinbindende Proteine und Proteine, die an Metallionen greifen – im Grunde, wie Sie Eisen und andere wichtige Metalle absorbieren.
Obwohl stark, gibt es Raum für Wachstum. Die Methode öffnet die Tür zur Entmystifizierung natürlicher Proteine, aber möglicherweise auch zur Entwicklung neuer Proteine für die synthetische Biologie. „Dies sind sehr leistungsstarke neue Ansätze, aber es gibt noch viel Raum für Verbesserungen“, sagte Baker.
Insgesamt ist es ein weiterer Gewinn für Deep Learning und ein fesselndes Beispiel dafür, wie KI und Biologie zusammenwirken können. „Deep Learning transformierte Proteinstrukturvorhersage in den letzten zwei Jahren, wir befinden uns jetzt mitten in einer ähnlichen Transformation des Proteindesigns“, sagte Baker.
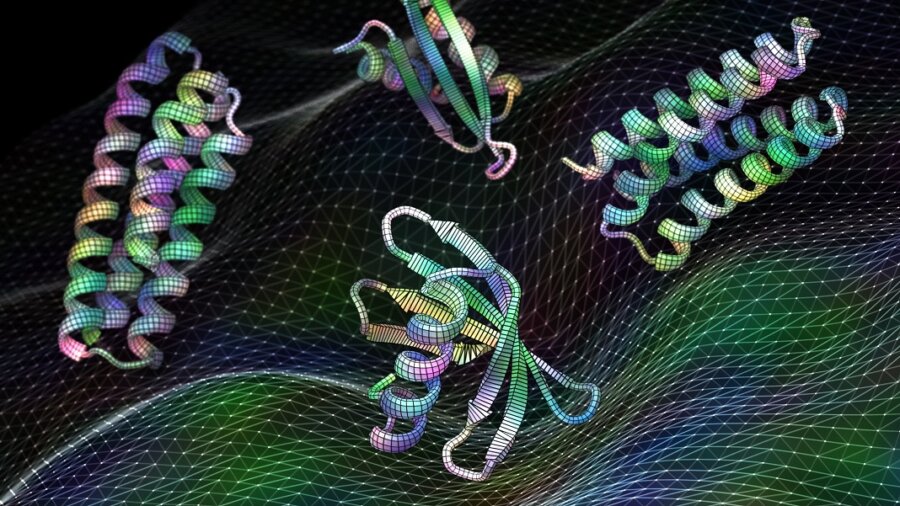
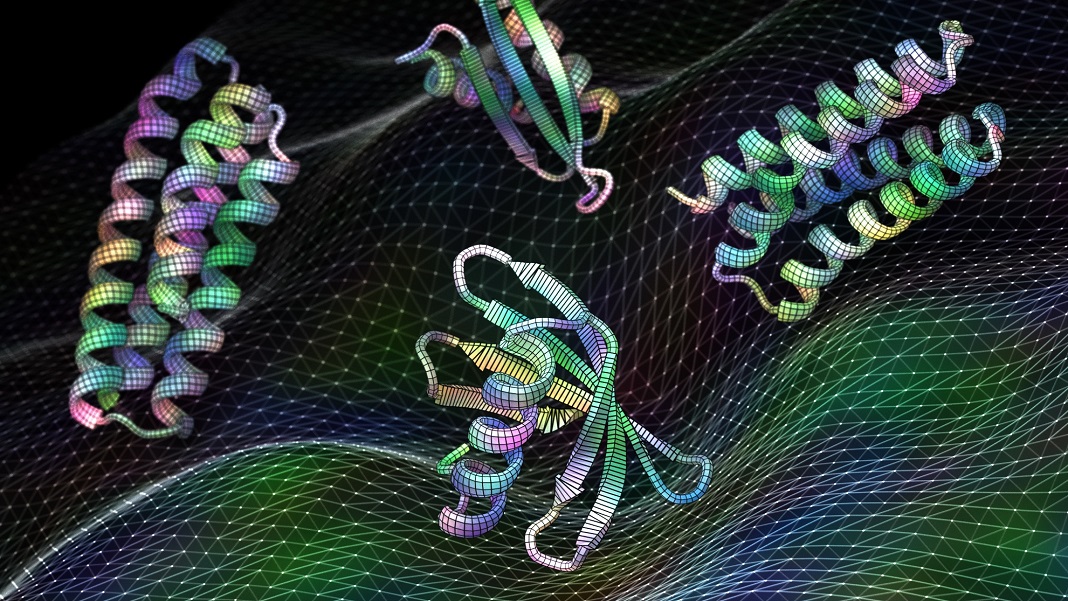
Bildnachweis: Ian C. Haydon/UW-Institut für Proteindesign. Neue Software für künstliche Intelligenz, die auf Proteinstrukturen trainiert wird, kann in Sekundenschnelle funktionelle Proteine erzeugen, einschließlich dieser Impfstoffkandidaten für das Atemwegsvirus RSV.