Αυτή η ανάρτηση γράφτηκε από κοινού με τον Sachin Kadyan, έναν κορυφαίο προγραμματιστή του OpenFold.
Στην ανακάλυψη φαρμάκων, η κατανόηση της τρισδιάστατης δομής των πρωτεϊνών είναι το κλειδί για την αξιολόγηση της ικανότητας ενός φαρμάκου να συνδέεται με αυτό, επηρεάζοντας άμεσα την αποτελεσματικότητά του. Ωστόσο, η πρόβλεψη της μορφής της τρισδιάστατης πρωτεΐνης είναι πολύ περίπλοκη, προκλητική, δαπανηρή και χρονοβόρα και μπορεί να πάρει χρόνια όταν χρησιμοποιούνται παραδοσιακές μέθοδοι όπως η περίθλαση ακτίνων Χ. Η εφαρμογή της μηχανικής μάθησης (ML) για την πρόβλεψη αυτών των δομών μπορεί να επιταχύνει σημαντικά τον χρόνο πρόβλεψης των πρωτεϊνικών δομών—από χρόνια σε ώρες. Αρκετές ερευνητικές ομάδες υψηλού προφίλ έχουν κυκλοφορήσει αλγόριθμους όπως οι AlphaFold3 (AF3), RoseTTAFold και άλλοι. Αυτοί οι αλγόριθμοι αναγνωρίστηκαν από Επιστήμη περιοδικό ως το 2021 Ανακάλυψη της χρονιάς.
Το OpenFold, που αναπτύχθηκε από το Πανεπιστήμιο Κολούμπια, είναι ένα μοντέλο πρόβλεψης πρωτεϊνικής δομής ανοιχτού κώδικα που υλοποιείται με το PyTorch. Το OpenFold είναι μια πιστή αναπαραγωγή του μοντέλου πρόβλεψης πρωτεϊνικής δομής AlphaFold2, ενώ προσφέρει βελτιώσεις απόδοσης σε σχέση με το AlphaFold2. Περιέχει μια σειρά από βελτιστοποιήσεις που σχετίζονται με την προπόνηση και τα συμπεράσματα που εκμεταλλεύονται διαφορετικούς αντισταθμίσεις μνήμης/χρόνου για διαφορετικά μήκη πρωτεΐνης με βάση την εκπαίδευση μοντέλων ή τις διαδρομές συμπερασμάτων. Για προπόνηση, το OpenFold υποστηρίζει βελτιστοποιήσεις FlashAttention που επιταχύνουν το στοιχείο προσοχής της ευθυγράμμισης πολλαπλών ακολουθιών (MSA). Οι βελτιστοποιήσεις FlashAttention μαζί με τη μεταγλώττιση JIT επιταχύνουν τη γραμμή συμπερασμάτων, παρέχοντας διπλάσια απόδοση για μικρότερες αλληλουχίες πρωτεϊνών από το AlphaFold2.
Για μεγαλύτερες πρωτεϊνικές δομές, το OpenFold διαθέτει επιτόπιες βελτιστοποιήσεις προσοχής και χαμηλής μνήμης, οι οποίες υποστηρίζουν προβλέψεις δομών πρωτεΐνης μήκους έως και 4,600 υπολειμμάτων, σε 40 GB A100 που βασίζεται σε GPU Amazon Elastic Compute Cloud (Amazon EC2) περιπτώσεις p4d. Επιπλέον, με τεχνικές βελτιστοποίησης χρήσης μνήμης, όπως η εκφόρτωση CPU, οι επιτόπιες λειτουργίες και το τεμάχιο (διαχωρισμός τανυστών εισόδου), το OpenFold μπορεί να προβλέψει δομές για πολύ μεγάλες πρωτεΐνες που διαφορετικά δεν θα ήταν δυνατές με το AlphaFold. Ο αγωγός ευθυγράμμισης στο OpenFold είναι πιο αποτελεσματικός από το AlphaFold με την αλυσίδα εργαλείων HHBlits/JackHMMER ή τον πολύ πιο γρήγορο αγωγό παραγωγής MSA που βασίζεται σε MMSeqs2.
Το Πανεπιστήμιο Κολούμπια δημοσίευσε δημόσια τα βάρη και τα δεδομένα εκπαίδευσης μοντέλων, που αποτελούνται από 400,000 αρχεία επισκέψεων προτύπου MSAs και PDB70, με άδεια χρήσης. Τα βάρη μοντέλων είναι διαθέσιμα μέσω σεναρίων στο Αποθετήριο GitHub, και οι MSA φιλοξενούνται από το Μητρώο Ανοικτών Δεδομένων στο AWS (RODA). Η χρήση Python και PyTorch για υλοποίηση επιτρέπει στο OpenFold να έχει πρόσβαση σε μια μεγάλη σειρά από λειτουργικές μονάδες και προγραμματιστές ML, διασφαλίζοντας έτσι τη συνεχή βελτίωση και βελτιστοποίησή του.
Σε αυτήν την ανάρτηση, δείχνουμε πώς μπορείτε να αναπτύξετε μοντέλα OpenFold Υπηρεσία Amazon Elastic Kubernetes (Amazon EKS) και πώς να κλιμακώσετε τα συμπλέγματα EKS για να μειώσετε δραστικά τους χρόνους συμπερασμάτων του υπολογισμού του MSA και της δομής πρωτεΐνης. Το Amazon EKS είναι μια διαχειριζόμενη υπηρεσία κοντέινερ για την εκτέλεση και την κλιμάκωση εφαρμογών Kubernetes στο AWS. Με το Amazon EKS, μπορείτε να εκτελέσετε αποτελεσματικά κατανεμημένες εργασίες εκπαίδευσης χρησιμοποιώντας τις πιο πρόσφατες περιπτώσεις EC2 χωρίς να χρειάζεται να εγκαταστήσετε, να χειριστείτε και να διατηρήσετε το δικό σας επίπεδο ελέγχου ή κόμβους. Είναι ένα δημοφιλές ενορχηστρωτής για ροές εργασίας ML και AI και μια ολοένα και πιο δημοφιλής υπηρεσία ενορχήστρωσης κοντέινερ σε μια τυπική αρχιτεκτονική συμπερασμάτων για εφαρμογές όπως μηχανές συστάσεων, ανάλυση συναισθήματος και κατάταξη διαφημίσεων που πρέπει να εξυπηρετούν μεγάλο αριθμό μοντέλων, με συνδυασμό κλασικής ML και βαθιάς μάθησης ( DL) μοντέλα.
Δείχνουμε την απόδοση αυτής της αρχιτεκτονικής για την εκτέλεση υπολογισμών στοίχισης και συμπερασμάτων στο δημοφιλές σύνολο δεδομένων ανοιχτού κώδικα Cameo. Εκτέλεση αυτού του φόρτου εργασίας από άκρη σε άκρη και στις 92 πρωτεΐνες που είναι διαθέσιμες στο Καμέα Το σύνολο δεδομένων θα διαρκέσει συνολικά 8 ώρες, το οποίο περιλαμβάνει τη λήψη των απαιτούμενων δεδομένων, τον υπολογισμό ευθυγράμμισης και τους χρόνους συμπερασμάτων.
Επισκόπηση λύσεων
Προχωράμε στη δημιουργία ενός συμπλέγματος EKS χρησιμοποιώντας Amazon FSx για λάμψη ως κατανεμημένο σύστημα αρχείων μας. Σας δείχνουμε πώς να κάνετε λήψη των απαραίτητων εικόνων, αρχείων μοντέλων, εικόνων κοντέινερ και αρχείων δήλωσης .yaml. Δείχνουμε επίσης πώς μπορείτε να εξυπηρετήσετε το μοντέλο χρησιμοποιώντας το FastAPI για να προβλέψετε τη δομή της τρισδιάστατης πρωτεΐνης. Το βήμα MSA στη ροή εργασιών αναδίπλωσης πρωτεΐνης είναι υπολογιστικά εντατικό και μπορεί να αντιπροσωπεύει το μεγαλύτερο μέρος του χρόνου συμπερασμάτων. Σε αυτήν την ανάρτηση, δείχνουμε πώς να ενορχηστρώνουμε πολλές εργασίες Kubernetes παράλληλα για να χρησιμοποιούμε συμπλέγματα σε κλίμακα για να επιταχύνουμε το βήμα MSA. Τέλος, παρέχουμε συγκρίσεις απόδοσης για διαφορετικές περιπτώσεις υπολογιστών και πώς μπορείτε να παρακολουθείτε τη χρήση της CPU και της GPU.
Μπορείτε να χρησιμοποιήσετε την αρχιτεκτονική αναφοράς σε αυτήν την ανάρτηση για να δοκιμάσετε διαφορετικούς αλγόριθμους αναδίπλωσης, να δοκιμάσετε υπάρχοντα προεκπαιδευμένα μοντέλα σε νέα δεδομένα ή να διαθέσετε αποδοτικά OpenFold API για ευρύτερη χρήση στον οργανισμό σας.
Ρυθμίστε το σύμπλεγμα EKS με ένα σύστημα αρχείων FSx for Luster
Χρησιμοποιούμε αυς-ντο-εκς, ένα έργο ανοιχτού κώδικα που παρέχει μια μεγάλη συλλογή από εύχρηστα και διαμορφώσιμα σενάρια και εργαλεία που σας επιτρέπουν να παρέχετε συμπλέγματα EKS και να εκτελέσετε τα συμπεράσματά σας. Για να δημιουργήσετε το σύμπλεγμα χρησιμοποιώντας το αποθετήριο aws-do-eks, ακολουθήστε τα βήματα στο αποθετήριο GitHub για να ρυθμίσετε και να εκκινήσετε το σύμπλεγμα EKS. Εάν λάβετε σφάλμα κατά τη δημιουργία του συμπλέγματος, ελέγξτε για αυτούς τους πιθανούς λόγους:
- Εάν οι ομάδες κόμβων απέτυχαν να δημιουργηθούν λόγω ανεπαρκούς χωρητικότητας, ελέγξτε τη διαθεσιμότητα των παρουσιών στην περιοχή που ζητήσατε και τα όρια χωρητικότητας.
- Ελέγξτε ότι ο καθορισμένος τύπος παρουσίας είναι διαθέσιμος ή υποστηρίζεται σε ένα δεδομένο AZ.
- Δημιουργία συμπλέγματος EKS AWS CloudFormation οι στοίβες μπορεί να μην έχουν διαγραφεί σωστά. Ίσως χρειαστεί να ελέγξετε τις ενεργές στοίβες CloudFormation για να δείτε εάν η διαγραφή της στοίβας απέτυχε.
Αφού δημιουργηθεί το σύμπλεγμα, χρειάζεστε τη διεπαφή γραμμής εντολών kubectl (CLI) στην παρουσία EC2 για να εκτελέσετε λειτουργίες Kubernetes. Σε μια παρουσία Linux, εκτελέστε την ακόλουθη εντολή για να εγκαταστήσετε το kubectl CLI. Αναφέρομαι στο διαθέσιμες εντολές για τυχόν προσαρμοσμένες απαιτήσεις.
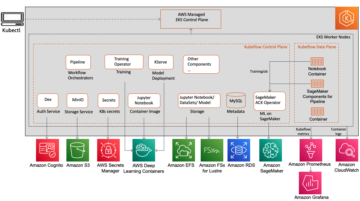
Ένα τυπικό σύμπλεγμα EKS στο AWS μοιάζει με το παρακάτω σχήμα.
Χρειαζόμαστε ένα επεκτάσιμο κοινόχρηστο σύστημα αρχείων στο οποίο μπορούν να έχουν πρόσβαση όλοι οι κόμβοι υπολογιστών στο σύμπλεγμα EKS. Το FSx for Luster είναι ένα πλήρως διαχειριζόμενο σύστημα αρχείων υψηλής απόδοσης που παρέχει λανθάνοντες χρόνους κάτω του χιλιοστού του δευτερολέπτου, απόδοση έως και εκατοντάδες GB/s και εκατομμύρια IOPS. Για να προσαρτήσετε το σύστημα αρχείων FSx for Luster στο σύμπλεγμα EKS, ανατρέξτε στο Δημιουργία Συστημάτων Αρχείων και Αντιγραφή Δεδομένων.
Μπορείτε να δημιουργήσετε το σύστημα αρχείων FSx for Luster στη Ζώνη Διαθεσιμότητας όπου βρίσκεται το μεγαλύτερο μέρος του υπολογισμού σας για να παρέχει τις χαμηλότερες καθυστερήσεις. Το σύστημα αρχείων μπορεί να προσπελαστεί από κόμβους ή λοβούς σε οποιαδήποτε Ζώνη Διαθεσιμότητας. Για απλότητα, σε αυτό το παράδειγμα, κρατήσαμε τους κόμβους στην ίδια Ζώνη Διαθεσιμότητας.
Λήψη δεδομένων OpenFold και αρχείων μοντέλων
Αντιγράψτε τα τεχνουργήματα και τις τράπεζες δεδομένων πρωτεϊνών που απαιτούνται για συμπέρασμα από το Απλή υπηρεσία αποθήκευσης Amazon κάδοι (Amazon S3). s3://aws-batch-architecture-for-alphafold-public-artifacts/ και s3://pdbsnapshots/ στο σύστημα αρχείων FSx for Luster που ρυθμίστηκε στο προηγούμενο βήμα. Η βάση δεδομένων αποτελείται από παραμέτρους AlphaFold, δεδομένα ανάλυσης Microbiome από το MGnify, Big Fantastic Database (BFD), βάση δεδομένων Protein Data Bank, αρχεία mmCIF και βάσεις δεδομένων PDB SeqRes. Τα σενάρια για λήψη και αποσυμπίεση των δεδομένων είναι διαθέσιμα στο download-openfold-data/scripts ντοσιέ. Χρησιμοποιήστε το αρχείο .yaml fsx-data-prep-pod.yaml για να εκτελέσετε μια εργασία Kubernetes για λήψη των δεδομένων. Μπορείτε να ξεκινήσετε πολλές εργασίες Kubernetes για να επιταχύνετε αυτή τη διαδικασία, επειδή η λήψη του αρχείου μπορεί να είναι χρονοβόρα και να διαρκέσει περίπου 4 ώρες. Ολοκληρώστε τα παρακάτω βήματα για να κάνετε λήψη όλων των δεδομένων στο FSx:
Σε αυτό το παράδειγμα, ο κοινός μας φάκελος FSx for Luster είναι /fsx-shared, που δημιουργήθηκε μετά την προσάρτηση του τόμου FSx for Luster στο σύμπλεγμα EKS. Όταν ολοκληρωθεί η εργασία, θα πρέπει να δείτε τους παρακάτω φακέλους στο fsx-shared φάκελο.
Κλωνοποιήστε το Αρχεία μοντέλου OpenFold και κατεβάστε τα σε έναν κάδο S3 και από εκεί σε ένα σύστημα αρχείων FSx for Luster χρησιμοποιώντας τα προηγούμενα βήματα. Το παρακάτω στιγμιότυπο οθόνης δείχνει τα επτά αρχεία που θα πρέπει να υπάρχουν στο σύστημα αρχείων FSX αφού ολοκληρώσετε τη λήψη.
Δημιουργήστε ένα αρχείο OpenFold Docker και ένα αρχείο δήλωσης .yaml
Έχουμε παράσχει ένα Αρχείο OpenFold Docker που μπορείτε να χρησιμοποιήσετε για να δημιουργήσετε ένα βασικό κοντέινερ που περιέχει όλες τις απαραίτητες εξαρτήσεις για την εκτέλεση του OpenFold. Για να εκτελέσετε το συμπέρασμα OpenFold με προεκπαιδευμένα μοντέλα OpenFold, πρέπει να εκτελέσετε τον ακόλουθο κώδικα:
Η run_pretrained_openfold.py Ο κώδικας που παρέχεται στο αποθετήριο OpenFold GitHub είναι ένας κώδικας συμπερασμάτων από άκρο σε άκρο που λαμβάνει εισόδους χρήστη και υπολογίζει ευθυγραμμίσεις εάν χρειάζεται χρησιμοποιώντας δυαδικά αρχεία jackhmmer και hhsuite, φορτώνει το μοντέλο OpenFold και εκτελεί συμπεράσματα. Περιλαμβάνει επίσης άλλες λειτουργίες, όπως χαλάρωση πρωτεΐνης, ανίχνευση μοντέλων και πολλαπλών μοντέλων, για να αναφέρουμε μερικές. Εκτελέστε το run_pretrained_openfold.py κώδικας σε ένα pod Kubernetes χρησιμοποιώντας το αρχείο .yaml ως εξής:
Αναπτύξτε μοντέλα OpenFold ως υπηρεσίες και δοκιμάστε τη λύση
Για να αναπτύξετε διακομιστές μοντέλων OpenFold ως API, πρέπει να ολοκληρώσετε τα ακόλουθα βήματα:
- Ενημέρωση inference_config.properties με πληροφορίες όπως όνομα μοντέλου OpenFold, διαδρομή προς τον κατάλογο ευθυγράμμισης, αριθμό μοντέλων που θα αναπτυχθούν ανά διακομιστή, τύπος παρουσίας διακομιστή μοντέλου, αριθμός διακομιστών και θύρα διακομιστή.
- Δημιουργήστε την εικόνα Docker με
build.sh. - Σπρώξτε την εικόνα Docker με
push.sh. - Αναπτύξτε τα μοντέλα με
deploy.sh.
Εάν χρειάζεται να προσαρμόσετε τα OpenFold API, χρησιμοποιήστε το fastapi-server.py αρχείο, το οποίο διαθέτει όλες τις κρίσιμες λειτουργίες που απαιτούνται για τη φόρτωση μοντέλων OpenFold, τον υπολογισμό των MSA και την εκτέλεση συμπερασμάτων.
Αρχικοποιήστε το model_config, template_featurizer, data_processor, να feature_processor αγωγών στο fastapi-server.py καλώντας τις αντίστοιχες κλάσεις τους. ο precompute_alignment Το API παίρνει μια ετικέτα πρωτεΐνης και μια ακολουθία ως προαιρετικές παραμέτρους και δημιουργεί μια στοίχιση εάν δεν υπάρχει ήδη. Η μεταβλητή alignment_dir καθορίζει τη θέση όπου αποθηκεύονται όλες οι ευθυγραμμίσεις. Το precompute_alignment API δημιουργεί τοπικούς καταλόγους ευθυγράμμισης χρησιμοποιώντας τις ετικέτες κάθε αλληλουχίας πρωτεΐνης. Για αυτόν τον λόγο, βεβαιωθείτε ότι οι ετικέτες κάθε πρωτεΐνης είναι μοναδικές. Όταν ολοκληρωθεί η εκτέλεση του API, το bfd_uniclust_hits.a3m, mgnify_hits.a3m, pdb70_hits.hhr, να uniref90_hits.a3m Τα αρχεία δημιουργούνται στον τοπικό κατάλογο ευθυγράμμισης.
Τηλεφώνησε στο openfold_predictions API συμπερασμάτων, το οποίο λαμβάνει μια ετικέτα πρωτεΐνης, μια αλληλουχία και ένα αναγνωριστικό μοντέλου. Αφού εντοπιστεί ο κατάλογος τοπικής ευθυγράμμισης, δημιουργείται ένα επεξεργασμένο λεξικό χαρακτηριστικών, το οποίο δίνει μια παρτίδα εισόδου. Στη συνέχεια, εκτελείται μια προκαταρκτική κλήση συμπερασμάτων με το μοντέλο για να δώσει την έξοδο, η οποία πρέπει να υποβληθεί σε μετεπεξεργασία με το prep_output λειτουργούν για να αποδώσουν μια μη χαλαρή πρωτεΐνη.
Όταν ο fastapi-server.py εκτελείται ο κώδικας, φορτώνει πολλά μοντέλα OpenFold σε κάθε GPU σε πολλές παρουσίες. Για να παρακολουθούμε ποιο μοντέλο φορτώνεται σε κάθε GPU, χρειαζόμαστε ένα παγκόσμιο λεξικό μοντέλων που αποθηκεύει τα αναγνωριστικά μοντέλων κάθε μοντέλου. Πρέπει να καθορίσετε ποιο αρχείο σημείου ελέγχου θέλετε να χρησιμοποιήσετε και τον αριθμό των μοντέλων που θα φορτωθούν ανά GPU, και αυτά τα μοντέλα φορτώνονται όταν εκτελείται το κοντέινερ, όπως φαίνεται στον ακόλουθο κώδικα:
Το αρχείο inference_config.properties έχει εισόδους που πρέπει να συμπληρώσετε, συμπεριλαμβανομένου του αρχείου σημείου ελέγχου που θα χρησιμοποιήσετε, του τύπου παρουσίας για συμπέρασμα, του αριθμού των διακομιστών μοντέλων και του αριθμού των μοντέλων που θα φορτωθούν ανά διακομιστή. Επιπλέον, περιλαμβάνει άλλες εισόδους που αντιστοιχούν σε ορίσματα εισόδου στο run_pretrained_openfold.py κωδικός, όπως ο αριθμός των CPU, config_preset, κι αλλα. Εάν χρειάζεται να προσθέσετε επιπλέον λειτουργίες, όπως χαλάρωση προσθήκης πρωτεΐνης, μπορείτε να προσθέσετε σχετικές παραμέτρους στο inference_config.properties και να κάνετε σχετικές αλλαγές στο fastapi-server.py κώδικας. Εάν καθορίσετε μοντέλα που θα εκτελούνται σε GPU και, για παράδειγμα, δύο διακομιστές μοντέλων με δύο μοντέλα που θα αναπτυχθούν ανά διακομιστή, θα αναπτυχθούν τέσσερις εφαρμογές Kubernetes (δείτε παρακάτω).
Είναι σημαντικό να καθορίσετε τον προεπιλεγμένο χώρο ονομάτων, διαφορετικά μπορεί να υπάρξουν περιπλοκές στην πρόσβαση στο FSx για κοινόχρηστους τόμους Luster από υπολογιστικούς πόρους σε ένα προσαρμοσμένο περιβάλλον χώρου ονομάτων.
Η φάκελο ανάπτυξης παρέχει το πρότυπο αρχείο μανιφέστου .yaml για την ανάπτυξη του μοντέλου OpenFold ως υπηρεσία και ένα σενάριο κελύφους generate-yaml.sh που δημιουργεί ένα αρχείο .yaml για κάθε υπηρεσία σε έναν συγκεκριμένο φάκελο. Για παράδειγμα, εάν καθορίσετε δύο διακομιστές μοντέλου και τύπο παρουσίας p3.2xlarge, openfold-gpu-0.yaml και openfold-gpu-1.yaml τα αρχεία δημιουργούνται στο app-openfold-gpu-p3.2xlarge ντοσιέ. Στη συνέχεια, μπορείτε να αναπτύξετε τις υπηρεσίες μοντέλου ως εξής:
kubectl apply -f app-openfold-gpu-p3.2xlargeΜετά την ανάπτυξη των υπηρεσιών, μπορείτε να προβάλετε τις αναπτυγμένες υπηρεσίες, όπως φαίνεται στο παρακάτω στιγμιότυπο οθόνης.
Εκτέλεση υπολογισμού ευθυγράμμισης
Η έκθεση της λειτουργικότητας υπολογισμού ευθυγράμμισης ως API μπορεί να έχει ορισμένες συγκεκριμένες περιπτώσεις χρήσης, αλλά πρέπει να είμαστε σε θέση να χρησιμοποιούμε βέλτιστα το σύμπλεγμα EKS, έτσι ώστε ο υπολογισμός της στοίχισης να μπορεί να γίνει με παράλληλο τρόπο. Δεν χρειαζόμαστε ακριβές παρουσίες που βασίζονται σε GPU για τον υπολογισμό της ευθυγράμμισης, επομένως πρέπει να προσθέσουμε παρουσίες έντασης μνήμης ή υπολογισμού με μεγάλο αριθμό CPU. Αφού δημιουργήσουμε ένα σύμπλεγμα EKS, μπορούμε να δημιουργήσουμε μια νέα ομάδα κόμβων εκτελώντας το eks-nodegroup-create.sh script και μπορούμε να κλιμακώσουμε τις παρουσίες από την ομάδα αυτόματης κλίμακας στην κονσόλα Amazon EC2 αφού βεβαιωθούμε ότι οι παρουσίες βρίσκονται στην ίδια Ζώνη Διαθεσιμότητας με το FSx για το Lustre. Επειδή ο υπολογισμός ευθυγράμμισης είναι πιο εντατικός στη μνήμη, προσθέσαμε παρουσίες r6 στο σύμπλεγμα EKS.
Η καμέα Ο φάκελος περιέχει όλα τα σχετικά σενάρια (αρχείο Docker, κώδικας Python, σενάρια δημιουργίας, ώθησης και κελύφους, και αρχείο δήλωσης .yaml) που δείχνουν τον τρόπο εκτέλεσης της υπολογιστικής στοίχισης σε ένα αρχείο FASTA ακολουθιών πρωτεϊνών. Για να εκτελέσετε τον υπολογισμό ευθυγράμμισης στο προσαρμοσμένο σύνολο δεδομένων FASTA, ολοκληρώστε τα ακόλουθα βήματα:
- Αποθηκεύστε το αρχείο FASTA στο φάκελο FSx.
- Δημιουργήστε ένα προσωρινό αρχείο FASTA για κάθε αλληλουχία πρωτεΐνης και αποθηκεύστε το στον φάκελο FSx. Για το σύνολο δεδομένων Cameo, αυτό γίνεται εκτελώντας
kubectl apply -f temp-fasta.yamlστοcameo-fastasφάκελο. - Ενημερώστε το
alignment_dirδιαδρομή στοprecompute_alignments.pyκώδικα, ο οποίος καθορίζει τον φάκελο προορισμού για την αποθήκευση των στοίχισης. - Δημιουργήστε και σπρώξτε την εικόνα Docker σε Μητρώο εμπορευματοκιβωτίων Amazon Elastic (ECR Amazon).
- Ενημερώστε το
run-cameo.yamlαρχείο με τον τύπο παρουσίας και τη διαδρομή προς την εικόνα Docker στο Amazon ECR και τον αριθμό των CPU, εάν χρειάζεται. - Ενημέρωση
run-grid.pyμε τις διαδρομές από τα βήματα 1 και 2. Αυτός ο κώδικας λαμβάνει τοrun-cameo.yamlαρχείο ως πρότυπο, δημιουργεί ένα αρχείο .yaml για κάθε εργασία υπολογισμού στοίχισης και τα αποθηκεύει στοcameo-yamlsφάκελο. - Τέλος, υποβάλετε όλες τις εργασίες τρέχοντας
kubectl apply -f cameo-yamls.
Η precompute_alignments.py Ο κώδικας φορτώνει ένα αρχείο FASTA με αλληλουχίες πρωτεϊνών. ο run-cameo.yaml Το αρχείο που εμφανίζεται στον παρακάτω κώδικα χρειάζεται απλώς να καθορίσει τον τύπο παρουσίας, την προδιαγραφή κοινόχρηστης βάσης τόμου και ορίσματα όπως ο αριθμός των CPU για τον υπολογισμό της ευθυγράμμισης:
Ανάλογα με τη διαθεσιμότητα των υπολογιστικών κόμβων στο σύμπλεγμα, θα μπορούσατε να υποβάλετε πολλές εργασίες Kubernetes παράλληλα. Ανάλογα με τις ανάγκες σας, θα μπορούσατε να έχετε μία ή περισσότερες αποκλειστικές παρουσίες που βασίζονται σε CPU. Αφού δημιουργήσετε μια ομάδα κόμβων τύπου παρουσίας CPU, μπορείτε εύκολα να την αυξήσετε ή να μειώσετε με μη αυτόματο τρόπο από την κονσόλα Amazon EC2. Εάν προκύψει ανάγκη για αυτόματη κλιμάκωση συμπλέγματος, αυτό θα μπορούσε επίσης να είναι δυνατό με το aws-do-eks πλαίσιο, αλλά θα περιλαμβανόταν σε μεταγενέστερη επανάληψη αυτής της λύσης.
Δοκιμές απόδοσης
Έχουμε δοκιμάσει την απόδοση της αρχιτεκτονικής μας στον ανοιχτό κώδικα Καμέα σύνολο δεδομένων. Αυτό το σύνολο δεδομένων έχει συνολικά 92 πρωτεΐνες διαφορετικού μήκους. Η ακόλουθη γραφική παράσταση δείχνει ένα ιστόγραμμα των μηκών αλληλουχίας, το οποίο έχει διάμεσο μήκος ακολουθίας 236 και τέσσερις ακολουθίες μεγαλύτερες από 600.
Δημιουργήσαμε αυτό το γράφημα με τον ακόλουθο κώδικα:
Ο υπολογισμός της ευθυγράμμισης είναι έντασης μνήμης και όχι εντατικών υπολογισμών, πράγμα που σημαίνει ότι η χρήση παρουσιών βελτιστοποιημένης μνήμης θα έχει μεγαλύτερη απόδοση κόστους από τις βελτιστοποιημένες περιπτώσεις υπολογισμού. Για τις δοκιμές μας, επιλέξαμε τα στιγμιότυπα r6i.xlarge, τα οποία διαθέτουν 4 vCPU και 32 GB μνήμης, και ένα pod αποσπάστηκε για μία εργασία υπολογισμού ευθυγράμμισης για κάθε ακολουθία πρωτεΐνης.
Ο παρακάτω πίνακας δείχνει τα αποτελέσματα για τις εργασίες υπολογισμού ευθυγράμμισης. Βλέπουμε ότι με 92 περιπτώσεις r6i.xlarge, θα μπορούσαμε να ολοκληρώσουμε τον υπολογισμό ευθυγράμμισης για 92 πρωτεΐνες για λιγότερο από $60. Για αναφορά, συγκρίναμε 1 παράδειγμα c6i.12xlarge με ένα μόνο pod που χρειάστηκε πάνω από 2 ημέρες για να ολοκληρωθεί ο υπολογισμός.
| Τύπος συμβολής | Συνολική διαθέσιμη μνήμη | Σύνολο διαθέσιμων vCPU | Ζητήθηκε Pod Memory | Ζητήθηκαν Pod CPU | Αριθμός λοβών | Χρονομετρημένος | Ωριαίο κόστος κατ' απαίτηση | συνολικό κόστος |
| r6i.xlarge | 32 GB | 4 | 16GB | 4 | 92 | 2.5 ώρες | $ 0.252 / ώρα | $58 |
| c6i.12xlarge | 96 GB | 48 | Προεπιλογή | 4 | 1 | 49 ώρες, 43 λεπτά | $ 2.04 / ώρα | $101 |
Η ακόλουθη γραφική παράσταση δείχνει τον χρόνο υπολογισμού ευθυγράμμισης έναντι των μηκών αλληλουχίας πρωτεΐνης.
Οι παρακάτω γραφικές παραστάσεις δείχνουν τη μέγιστη χρήση της CPU των 92×4 = 368 vCPU στην ομάδα αυτόματης κλίμακας r6i.xlarge. Η κάτω πλοκή είναι απλώς η συνέχεια της πάνω. Βλέπουμε ότι οι CPU χρησιμοποιήθηκαν για τη μέγιστη χωρητικότητά τους και σταδιακά πέφτουν στο 0 όταν τελειώσουν όλες οι εργασίες.
Τέλος, μετά τον υπολογισμό των MSA, μπορούμε να εκτελέσουμε το συμπέρασμα καλώντας τα API του διακομιστή μοντέλου. Ο παρακάτω πίνακας δείχνει τους συνολικούς χρόνους συμπερασμάτων στο σύνολο δεδομένων Cameo για περιπτώσεις p3.2xlarge έναντι g4dn.xlarge. Με το μηχάνημα p3.2xlarge, ο υπολογισμός MSA σε 92 πρωτεΐνες του συνόλου δεδομένων Cameo μπορεί να γίνει τρεις φορές πιο γρήγορα από το μηχάνημα g4dn.xlarge.
| Τύπος συμβολής | Αριθμός GPU | Τύπος GPU | Σύνολο διαθέσιμων vCPU | Μνήμη CPU | Μνήμη GPU | Συνολικός χρόνος συμπερασμάτων στο σύνολο δεδομένων Cameo | Ωριαίο κόστος κατ' απαίτηση | συνολικό κόστος |
| σελ.3.2xlarge | 1 | Tesla V100 | 8 | 61 GiB | 16 | 1.36 ώρες | $ 3.06 / ώρα | $4 |
| g4dn.xlarge | 1 | Tesla T4 | 4 | 16 GiB | 16 GiB | 3.95 ώρες | $ 0.526 / ώρα | $2 |
Η ακόλουθη γραφική παράσταση δείχνει τον συνολικό χρόνο που απαιτείται για τη φόρτωση των αρχείων MSA και την πραγματοποίηση συμπερασμάτων σε μια παρουσία p3.2xlarge και μια παρουσία g4dn.xlarge ως συνάρτηση του μήκους της αλληλουχίας πρωτεΐνης. Για ακολουθίες μεγαλύτερες από 200, ο χρόνος συμπερασμάτων με το παράδειγμα p3.2xlarge είναι τρεις φορές ταχύτερος από το παράδειγμα g4dn.xlarge, ενώ για μικρότερες ακολουθίες είναι 1–2 φορές ταχύτερος.
εκκαθάριση
Είναι σημαντικό να περιστρέφονται πόροι μετά την εκπαίδευση του μοντέλου, προκειμένου να αποφευχθούν τα κόστη που σχετίζονται με την εκτέλεση περιπτώσεων αδράνειας. Με κάθε σενάριο που δημιουργεί πόρους, το GitHub repo παρέχει ένα αντίστοιχο σενάριο για τη διαγραφή τους. Για να καθαρίσουμε τις ρυθμίσεις μας, πρέπει να διαγράψουμε το σύστημα αρχείων FSx πριν διαγράψουμε το σύμπλεγμα, επειδή σχετίζεται με ένα υποδίκτυο στο VPC του συμπλέγματος. Για να διαγράψετε το σύστημα αρχείων FSx, εκτελέστε την ακόλουθη εντολή μέσα από το FSX φάκελο:
Σημειώστε ότι αυτό όχι μόνο θα διαγράψει τον μόνιμο τόμο, αλλά θα διαγράψει επίσης το σύστημα αρχείων FSx και όλα τα δεδομένα στο σύστημα αρχείων θα χαθούν.
Όταν ολοκληρωθεί αυτό το βήμα, μπορούμε να διαγράψουμε το σύμπλεγμα χρησιμοποιώντας το ακόλουθο σενάριο στο εξ φάκελο:
Αυτό θα διαγράψει όλα τα υπάρχοντα pod, θα καταργήσει το σύμπλεγμα και θα διαγράψει το VPC που δημιουργήθηκε στην αρχή.
Συμπέρασμα
Σε αυτήν την ανάρτηση, δείξαμε πώς να χρησιμοποιήσετε ένα σύμπλεγμα EKS για την εκτέλεση συμπερασμάτων με μοντέλα OpenFold. Έχουμε δημοσιεύσει τις οδηγίες στο AWS EKS Architecture For OpenFold Inference GitHub repo, όπου μπορείτε να βρείτε οδηγίες βήμα προς βήμα για το πώς να δημιουργήσετε ένα σύμπλεγμα EKS, να προσαρτήσετε ένα κοινό σύστημα αρχείων σε αυτό, να κατεβάσετε δεδομένα OpenFold, να εκτελέσετε υπολογισμούς MSA και να αναπτύξετε μοντέλα OpenFold ως API. Για περισσότερες πληροφορίες σχετικά με το OpenFold, επισκεφτείτε το αποθετήριο OpenFold GitHub.
Σχετικά με τους συγγραφείς
 Shubha Kumbadakone είναι ειδικός του Sr. GTM για αυτοδιαχειριζόμενη μηχανική εκμάθηση με εστίαση σε λογισμικό και εργαλεία ανοιχτού κώδικα. Έχει περισσότερα από 17 χρόνια εμπειρίας στην υποδομή cloud και τη μηχανική εκμάθηση και βοηθά τους πελάτες να δημιουργήσουν την κατανεμημένη εκπαίδευση και τα συμπεράσματά τους σε κλίμακα για τα μοντέλα ML τους στο AWS. Κατέχει επίσης ένα δίπλωμα ευρεσιτεχνίας για έναν αλγόριθμο προσωρινής αποθήκευσης για γρήγορη συνέχιση από την αδρανοποίηση για κινητά συστήματα.
Shubha Kumbadakone είναι ειδικός του Sr. GTM για αυτοδιαχειριζόμενη μηχανική εκμάθηση με εστίαση σε λογισμικό και εργαλεία ανοιχτού κώδικα. Έχει περισσότερα από 17 χρόνια εμπειρίας στην υποδομή cloud και τη μηχανική εκμάθηση και βοηθά τους πελάτες να δημιουργήσουν την κατανεμημένη εκπαίδευση και τα συμπεράσματά τους σε κλίμακα για τα μοντέλα ML τους στο AWS. Κατέχει επίσης ένα δίπλωμα ευρεσιτεχνίας για έναν αλγόριθμο προσωρινής αποθήκευσης για γρήγορη συνέχιση από την αδρανοποίηση για κινητά συστήματα.
 Ανκούρ Σριβαστάβα είναι αρχιτέκτονας Sr. Solutions στην ομάδα ML Frameworks. Επικεντρώνεται στην παροχή βοήθειας στους πελάτες με αυτοδιαχειριζόμενη κατανεμημένη εκπαίδευση και εξαγωγή συμπερασμάτων σε κλίμακα για το AWS. Η εμπειρία του περιλαμβάνει βιομηχανική προγνωστική συντήρηση, ψηφιακά δίδυμα, πιθανοτική βελτιστοποίηση σχεδίασης και έχει ολοκληρώσει τις διδακτορικές του σπουδές από τη Μηχανολογία στο Πανεπιστήμιο Rice και τη μεταδιδακτορική έρευνα από το Ινστιτούτο Τεχνολογίας της Μασαχουσέτης.
Ανκούρ Σριβαστάβα είναι αρχιτέκτονας Sr. Solutions στην ομάδα ML Frameworks. Επικεντρώνεται στην παροχή βοήθειας στους πελάτες με αυτοδιαχειριζόμενη κατανεμημένη εκπαίδευση και εξαγωγή συμπερασμάτων σε κλίμακα για το AWS. Η εμπειρία του περιλαμβάνει βιομηχανική προγνωστική συντήρηση, ψηφιακά δίδυμα, πιθανοτική βελτιστοποίηση σχεδίασης και έχει ολοκληρώσει τις διδακτορικές του σπουδές από τη Μηχανολογία στο Πανεπιστήμιο Rice και τη μεταδιδακτορική έρευνα από το Ινστιτούτο Τεχνολογίας της Μασαχουσέτης.
- AI
- αι τέχνη
- ι γεννήτρια τέχνης
- ρομπότ ai
- Υπηρεσία Amazon Elastic Kubernetes
- Εκμάθηση μηχανών του Αμαζονίου
- τεχνητή νοημοσύνη
- πιστοποίηση τεχνητής νοημοσύνης
- τεχνητή νοημοσύνη στον τραπεζικό τομέα
- ρομπότ τεχνητής νοημοσύνης
- ρομπότ τεχνητής νοημοσύνης
- λογισμικό τεχνητής νοημοσύνης
- Μηχανική εκμάθηση AWS
- blockchain
- συνέδριο blockchain ai
- Coingenius
- συνομιλητική τεχνητή νοημοσύνη
- κρυπτοσυνεδριο αι
- του νταλ
- βαθιά μάθηση
- έχεις google
- μάθηση μηχανής
- Πλάτων
- πλάτων αι
- Πληροφορία δεδομένων Plato
- Παιχνίδι Πλάτωνας
- Πλάτωνα δεδομένα
- platogaming
- κλίμακα αι
- σύνταξη
- zephyrnet