Valgud juhivad paljusid bioloogilisi protsesse, nagu ensüümide aktiivsus, molekulaarne transport ja raku tugi. Valgu kolmemõõtmeline struktuur annab ülevaate selle funktsioonist ja sellest, kuidas see interakteerub teiste biomolekulidega. Eksperimentaalsed meetodid valgu struktuuri määramiseks, nagu röntgenkristallograafia ja NMR-spektroskoopia, on kallid ja aeganõudvad.
Seevastu hiljuti väljatöötatud arvutusmeetodid suudavad kiiresti ja täpselt ennustada valgu struktuuri selle aminohappejärjestuse põhjal. Need meetodid on kriitilised valkude jaoks, mida on raske eksperimentaalselt uurida, näiteks membraanivalgud, mis on paljude ravimite sihtmärgid. Üks tuntud näide sellest on AlfaFold, sügaval õppimisel põhinev algoritm, mida tähistatakse selle täpsete prognooside eest.
ESMFold on veel üks ülitäpne, sügaval õppimisel põhinev meetod, mis on välja töötatud valgu struktuuri ennustamiseks selle aminohappejärjestuse põhjal. ESMFold kasutab selgroona suurt valgukeele mudelit (pLM) ja töötab otsast lõpuni. Erinevalt AlphaFold2-st ei vaja see otsingut ega Mitme järjestuse joondamine (MSA) samm, samuti ei tugine see prognooside genereerimiseks välistele andmebaasidele. Selle asemel koolitas arendusmeeskond mudelit miljonite UniRefi valgujärjestuste põhjal. Treeningu ajal töötas mudel välja tähelepanumustrid, mis elegantselt esindavad järjestuse aminohapete vahelisi evolutsioonilisi interaktsioone. Selline pLM-i kasutamine MSA asemel võimaldab kuni 60 korda kiiremat ennustusaega kui teised tipptasemel mudelid.
Selles postituses kasutame Hugging Face with eelkoolitatud ESMFoldi mudelit Amazon SageMaker ennustada raske ahela struktuuri trastuzumabon monoklonaalne antikeha esmakordselt välja töötanud Genentech raviks HER2-positiivne rinnavähk. Selle valgu struktuuri kiire ennustamine võib olla kasulik, kui teadlased soovivad testida järjestuse modifikatsioonide mõju. See võib potentsiaalselt kaasa tuua patsientide elulemuse paranemise või kõrvaltoimete vähenemise.
See postitus sisaldab Jupyteri märkmiku ja sellega seotud skriptide näidet GitHubi hoidla.
Eeldused
Soovitame seda näidet käivitada an Amazon SageMaker Studio märkmik PyTorch 1.13 Python 3.9 protsessoriga optimeeritud kujutise käitamine ml.r5.xlarge eksemplaritüübil.
Visualiseerige trastuzumabi eksperimentaalset struktuuri
Alustuseks kasutame biopython raamatukogu ja abiskript trastuzumabi struktuuri allalaadimiseks saidilt RCSB valgu andmepank:
Järgmisena kasutame py3Dmol teek struktuuri visualiseerimiseks interaktiivse 3D-visualisatsioonina:
Järgmine joonis kujutab valgu andmepangast (PDB) pärinevat 3D-valgu struktuuri 1N8Z. Sellel pildil on trastuzumabi kerge ahel kuvatud oranžina, raske ahel on sinine (muutuva piirkonnaga helesinisega) ja HER2 antigeen on roheline.
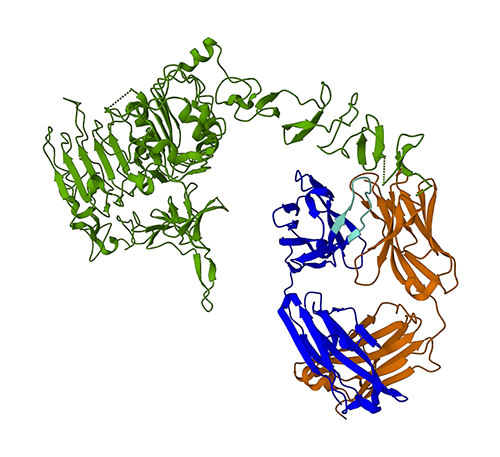
Esmalt kasutame ESMFoldi raske ahela (ahel B) struktuuri ennustamiseks selle aminohappejärjestuse põhjal. Seejärel võrdleme ennustust ülaltoodud eksperimentaalselt määratud struktuuriga.
ESMFoldi abil ennustage trastuzumabi raske ahela struktuur selle järjestuse põhjal
Kasutame ESMFoldi mudelit raske ahela struktuuri ennustamiseks ja võrdleme seda katsetulemusega. Alustuseks kasutame Studios eelehitatud märkmikukeskkonda, mis sisaldab mitut olulist teeki, nt PyTorch, eelinstallitud. Kuigi saaksime sülearvuti analüüsi jõudluse parandamiseks kasutada kiirendatud eksemplari tüüpi, kasutame selle asemel kiirendamata eksemplari ja käivitame protsessoris ESMFoldi prognoosi.
Esiteks laadime eelnevalt koolitatud ESMFoldi mudeli ja tokenisaatori Kallistamine Face Hub:
Järgmisena kopeerime mudeli oma seadmesse (antud juhul CPU-sse) ja määrame mõned mudeli parameetrid:
Valgujärjestuse analüüsiks ettevalmistamiseks peame selle märgistama. See tõlgib aminohapete sümbolid (EVQLV…) ESMFoldi mudelile arusaadavasse numbrivormingusse (6,19,5,10,19,…):
Järgmisena kopeerime tokeniseeritud sisendi režiimi, teeme ennustuse ja salvestame tulemuse faili:
See võtab kiirendamata eksemplaritüübi puhul aega umbes 3 minutit, näiteks r5.
ESMFoldi ennustuse täpsust saame kontrollida, võrreldes seda eksperimentaalse struktuuriga. Teeme seda kasutades US-Align tööriist, mille on välja töötanud Michigani ülikooli Zhang Lab:
| PDB-ahel1 | PDB-ahel2 | TM-skoor |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
. malli modelleerimise skoor (TM-skoor) on valgustruktuuride sarnasuse hindamise mõõdik. Hinne 1.0 näitab täiuslikku sobivust. Skoorid üle 0.7 näitavad, et valkudel on sama selgroog. Hinded üle 0.9 näitavad, et valgud on funktsionaalselt vahetatavad allkasutamiseks. Meie puhul, kui saavutame TM-Score 0.802, oleks ESMFoldi ennustus tõenäoliselt sobiv selliste rakenduste jaoks nagu struktuuri hindamine või ligandi sidumise katsed, kuid ei pruugi sobida sellisteks kasutusjuhtudeks nagu molekulaarne asendus mis nõuavad väga suurt täpsust.
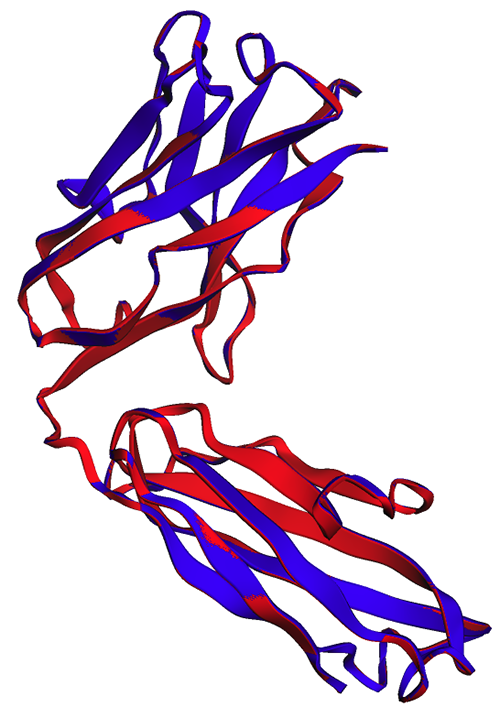
Saame selle tulemuse kinnitada joondatud struktuuride visualiseerimisega. Need kaks struktuuri näitavad suurt, kuid mitte täiuslikku kattumist. Valgu struktuuri prognoosimine on kiiresti arenev valdkond ja paljud uurimisrühmad töötavad välja üha täpsemaid algoritme!
Juurutage ESMFold SageMakeri järelduse lõpp-punktina
Mudeli järeldamise käivitamine sülearvutis sobib katsetamiseks, kuid mis siis, kui teil on vaja mudelit rakendusega integreerida? Või MLOps torujuhe? Sel juhul on parem võimalus juurutada oma mudel järelduse lõpp-punktina. Järgmises näites juurutame ESMFoldi kiirendatud eksemplari SageMakeri reaalajas järelduse lõpp-punktina. SageMakeri reaalajas lõpp-punktid pakuvad skaleeritavat, kulutõhusat ja turvalist viisi masinõppe (ML) mudelite juurutamiseks ja hostimiseks. Automaatse skaleerimisega saate kohandada lõpp-punkti käitavate eksemplaride arvu, et see vastaks teie rakenduse nõudmistele, optimeerides kulusid ja tagades kõrge kättesaadavuse.
Eelnevalt ehitatud SageMaker konteiner näo kallistamiseks muudab süvaõppemudelite juurutamise tavapäraste ülesannete jaoks lihtsaks. Uudsete kasutusjuhtumite jaoks, nagu valgu struktuuri ennustamine, peame aga määratlema kohandamise inference.py skript mudeli laadimiseks, ennustuse käivitamiseks ja väljundi vormindamiseks. See skript sisaldab suures osas sama koodi, mida kasutasime oma märkmikus. Samuti loome a requirements.txt faili, et määratleda mõned Pythoni sõltuvused, mida meie lõpp-punkt kasutada. Meie loodud faile näete lehel GitHubi hoidla.
Järgmisel joonisel on trastuzumabi raske ahela eksperimentaalne (sinine) ja prognoositud (punane) struktuur väga sarnane, kuid mitte identne.

Pärast seda, kui oleme loonud failis vajalikud failid code kataloogi, juurutame oma mudeli SageMakeri abil HuggingFaceModel klass. See kasutab eelehitatud konteinerit, et lihtsustada Hugging Face mudelite SageMakeris juurutamist. Pange tähele, et lõpp-punkti loomiseks võib kuluda 10 minutit või rohkem, olenevalt saadavusest ml.g4dn eksemplari tüübid meie piirkonnas.
Kui lõpp-punkti juurutamine on lõppenud, saame valgujärjestuse uuesti esitada ja kuvada ennustuse paar esimest rida:
Kuna kasutasime oma lõpp-punkti kiirendatud eksemplari, peaks ennustus võtma vaid mõne sekundi. Tulemuse iga rida vastab ühele aatomile ja sisaldab aminohappe identiteeti, kolme ruumikoordinaati ja pLDDT skoor esindab ennustuse usaldust selles kohas.
| PB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARN_X | CARN_Y | CARN_Z | TÄITUS | PLDDT | ATOM_ID |
| ATOM | 1 | N | G.L.U. | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | G.L.U. | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | G.L.U. | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | G.L.U. | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | G.L.U. | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | G.L.U. | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | G.L.U. | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | G.L.U. | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | G.L.U. | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Kasutades sama meetodit nagu varem, näeme, et märkmik ja lõpp-punkti ennustused on identsed.
| PDB-ahel1 | PDB-ahel2 | TM-skoor |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
Nagu on näidatud järgmisel joonisel, näitavad ESMFoldi ennustused, mis on genereeritud sülearvutis (punane) ja lõpp-punkti järgi (sinine), täiuslikku joondust.
Koristage
Edasiste tasude vältimiseks kustutame oma järelduse lõpp-punkti ja testiandmed:
kokkuvõte
Valgu struktuuri arvutuslik ennustamine on valkude funktsiooni mõistmiseks oluline vahend. Lisaks alusuuringutele on sellistel algoritmidel nagu AlphaFold ja ESMFold palju rakendusi meditsiinis ja biotehnoloogias. Nende mudelite loodud struktuursed teadmised aitavad meil paremini mõista, kuidas biomolekulid interakteeruvad. See võib seejärel viia patsientidele paremate diagnostikavahendite ja ravimeetoditeni.
Selles postituses näitame, kuidas juurutada ESMFoldi valgukeele mudelit Hugging Face Hubist skaleeritava järelduse lõpp-punktina, kasutades SageMakerit. Lisateavet Hugging Face mudelite SageMakeris juurutamise kohta leiate aadressilt Kasutage Hugging Face'i koos Amazon SageMakeriga. Samuti leiate rohkem valguteaduse näiteid Suurepärane AWS-i valguanalüüs GitHubi repo. Palun jätke meile kommentaar, kui on muid näiteid, mida soovite näha!
Autoritest
 Brian lojaalne on AI/ML lahenduste vanemarhitekt ettevõtte Amazon Web Services ülemaailmses tervishoiu- ja bioteaduste meeskonnas. Tal on enam kui 17-aastane kogemus biotehnoloogia ja masinõppe vallas ning ta soovib kirglikult aidata klientidel genoomseid ja proteoomilisi probleeme lahendada. Vabal ajal meeldib talle süüa teha ja sõprade ja perega süüa.
Brian lojaalne on AI/ML lahenduste vanemarhitekt ettevõtte Amazon Web Services ülemaailmses tervishoiu- ja bioteaduste meeskonnas. Tal on enam kui 17-aastane kogemus biotehnoloogia ja masinõppe vallas ning ta soovib kirglikult aidata klientidel genoomseid ja proteoomilisi probleeme lahendada. Vabal ajal meeldib talle süüa teha ja sõprade ja perega süüa.
 Shamika Ariyawansa on AI/ML spetsialistilahenduste arhitekt Amazon Web Servicesi ülemaailmses tervishoiu- ja bioteaduste meeskonnas. Ta teeb kirglikult koostööd klientidega, et kiirendada nende AI ja ML kasutuselevõttu, pakkudes tehnilisi juhiseid ning aidates neil uuendusi teha ja AWS-is turvalisi pilvelahendusi luua. Väljaspool tööd armastab ta suusatada ja maastikul sõita.
Shamika Ariyawansa on AI/ML spetsialistilahenduste arhitekt Amazon Web Servicesi ülemaailmses tervishoiu- ja bioteaduste meeskonnas. Ta teeb kirglikult koostööd klientidega, et kiirendada nende AI ja ML kasutuselevõttu, pakkudes tehnilisi juhiseid ning aidates neil uuendusi teha ja AWS-is turvalisi pilvelahendusi luua. Väljaspool tööd armastab ta suusatada ja maastikul sõita.
 Yanjun Qi on rakendusteaduste vanemjuht AWS-i masinõppelahenduste laboris. Ta teeb uuendusi ja rakendab masinõpet, et aidata AWS-i klientidel kiirendada nende tehisintellekti ja pilve kasutuselevõttu.
Yanjun Qi on rakendusteaduste vanemjuht AWS-i masinõppelahenduste laboris. Ta teeb uuendusi ja rakendab masinõpet, et aidata AWS-i klientidel kiirendada nende tehisintellekti ja pilve kasutuselevõttu.
- SEO-põhise sisu ja PR-levi. Võimenduge juba täna.
- PlatoAiStream. Web3 andmete luure. Täiustatud teadmised. Juurdepääs siia.
- Tuleviku rahapaja Adryenn Ashley. Juurdepääs siia.
- Ostke ja müüge IPO-eelsete ettevõtete aktsiaid koos PREIPO®-ga. Juurdepääs siia.
- Allikas: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :on
- :on
- :mitte
- $ UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- MEIST
- üle
- AC
- kiirendama
- kiirendatud
- täpsus
- täpne
- täpselt
- saavutamisel
- tegevus
- lisamine
- Vastuvõtmine
- AI
- AI / ML
- algoritm
- algoritme
- joondatud
- joondumine
- Ka
- Kuigi
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- analüüs
- ja
- Teine
- mistahes
- taotlus
- rakendused
- rakendatud
- asjakohane
- OLEME
- AS
- Hindamine
- At
- aatom
- tähelepanu
- Automaatne
- kättesaadavus
- vältima
- AWS
- AWS-i masinõpe
- Selgroog
- Pank
- põhiline
- BE
- enne
- alustama
- Parem
- vahel
- siduv
- biotehnoloogia
- sinine
- ehitama
- kuid
- by
- CAN
- vähk
- multifilm
- juhul
- juhtudel
- tähistati
- kett
- väljakutseid
- koormuste
- kontrollima
- klass
- Cloud
- pilve adopteerimine
- kood
- värv
- tuleb
- kommentaar
- ühine
- võrdlema
- võrrelda
- täitma
- usaldus
- Konteiner
- kontrast
- vastab
- kuluefektiivne
- kulud
- võiks
- looma
- loodud
- kriitiline
- Kliendid
- andmed
- andmebaasid
- kuupäev Kellaaeg
- sügav
- sügav õpe
- Deepmind
- Kraad
- nõudmisi
- Olenevalt
- juurutada
- lähetatud
- juurutamine
- kasutuselevõtu
- Määrama
- kindlaksmääratud
- arenenud
- arenev
- & Tarkvaraarendus
- seade
- raske
- Ekraan
- do
- ei
- Ei tee
- lae alla
- ajam
- Narkootikumide
- ajal
- iga
- lihtne
- mõju
- mõju
- võimaldab
- lõpp
- Lõpp-punkt
- tagades
- keskkond
- näide
- näited
- kallis
- kogemus
- katseid
- väline
- äärmiselt
- nägu
- pere
- kiiremini
- vähe
- vähem
- väli
- Joonis
- fail
- Faile
- leidma
- lõpp
- esimene
- Järel
- eest
- formaat
- sõbrad
- Alates
- funktsioon
- edasi
- tekitama
- loodud
- GitHub
- Globaalne
- Green
- juhised
- Olema
- he
- tervishoid
- raske
- aitama
- aidates
- Suur
- tema
- võõrustaja
- Kuidas
- Kuidas
- aga
- HTML
- HTTPS
- Keskus
- Kallistav Nägu
- identiques
- Identity
- if
- pilt
- import
- oluline
- parandama
- paranenud
- in
- hõlmab
- näitama
- näitab
- info
- uuendama
- sisend
- ülevaade
- teadmisi
- Näiteks
- selle asemel
- integreerima
- suhelda
- interaktsioonid
- interaktiivne
- interaktiivne
- sisse
- IT
- ITS
- labor
- keel
- suur
- viima
- õppimine
- Lahkuma
- raamatukogud
- Raamatukogu
- elu
- Maaülikooli
- valgus
- nagu
- Tõenäoliselt
- koormus
- liising
- lookup
- masin
- masinõpe
- tegema
- TEEB
- juht
- palju
- Vastama
- mai..
- meditsiin
- Vastama
- meetod
- meetodid
- meetriline
- Michigan
- miljonid
- protokoll
- ML
- MLOps
- viis
- mudel
- mudelid
- Muudatused
- molekulaarne
- rohkem
- palju
- nimi
- vajalik
- Vajadus
- NIH
- märkmik
- romaan
- number
- esemeid
- vananenud
- of
- on
- ONE
- ainult
- tegutseb
- optimeerimine
- valik
- or
- oranž
- OS
- Muu
- meie
- väljund
- väljaspool
- parameetrid
- kirglik
- patsient
- patsientidel
- mustrid
- täiuslik
- jõudlus
- torujuhe
- Platon
- Platoni andmete intelligentsus
- PlatoData
- palun
- post
- potentsiaalselt
- ennustada
- ennustada
- prognoosimine
- ennustus
- Ennustused
- Valmistama
- varem
- protsess
- Protsessid
- Valk
- Valgud
- anda
- annab
- pakkudes
- Python
- pütorch
- Qi
- kiiresti
- kiiresti
- kiiresti arenev valdkond
- reaalajas
- soovitama
- Red
- piirkond
- seotud
- lootma
- esindama
- esindavad
- esindab
- nõudma
- teadustöö
- Teadlased
- kaasa
- ROW
- jooks
- jooksmine
- salveitegija
- SageMakeri järeldus
- sama
- Säästa
- skaalautuvia
- ketendamine
- teadus
- TEADUSED
- skoor
- Hinded
- skripte
- sekundit
- kindlustama
- vaata
- vanem
- Jada
- Teenused
- komplekt
- mitu
- Jaga
- ta
- peaks
- näitama
- näidatud
- külg
- sarnane
- lihtsustama
- ühekordne
- lahendus
- Lahendused
- LAHENDAGE
- mõned
- ruumiline
- spetsialist
- Spektroskoopia
- kiirus
- algus
- modernne
- Samm
- struktuuriline
- struktuur
- stuudio
- Uuring
- selline
- sobiv
- toetama
- ellujäämine
- Võtma
- võtab
- eesmärgid
- ülesanded
- meeskond
- meeskonnad
- Tehniline
- test
- kui
- et
- .
- oma
- Neile
- SIIS
- Seal.
- Need
- see
- kolm
- kolmemõõtmeline
- aeg
- aega võttev
- korda
- et
- märkima
- märgistatud
- tööriist
- töövahendid
- tõrvik
- koolitatud
- koolitus
- trafod
- transportida
- ravi
- kaks
- tüüp
- liigid
- mõistma
- mõistmine
- Ülikool
- erinevalt
- us
- kasutama
- Kasutatud
- kasutamine
- KINNITAGE
- väga
- vaade
- visualiseerimine
- W
- tagaotsitav
- Tee..
- we
- web
- veebiteenused
- hästi tuntud
- M
- Wikipedia
- will
- koos
- WordPress
- Töö
- töötab
- oleks
- röntgen
- sa
- Sinu
- sephyrnet