Lääkeyhtiöiden, jotka hakevat lupaa säänteleviltä virastoilta, kuten Yhdysvaltain elintarvike- ja lääkevirastolta (FDA) tai Japanin lääke- ja lääkinnällisten laitteiden virastolta (PMDA) myydäkseen lääkkeitään markkinoilla, on toimitettava todisteet siitä, että heidän lääkkeensä on turvallinen ja tehokas sille aiotulla tavalla. käyttää. Lääkäreiden, tilastotieteilijöiden, kemistien, farmakologien ja muiden kliinisten tutkijoiden ryhmä tarkastelee kliinisen tutkimuksen toimittamista koskevia tietoja ja ehdotetut merkinnät. Jos katsauksessa todetaan, että on olemassa riittävästi tilastollista näyttöä sen osoittamiseksi, että lääkkeen terveyshyödyt ovat riskejä suuremmat, lääke hyväksytään myyntiin.
Kliinisen tutkimuksen toimituspaketti koostuu taulukkotiedoista, analyysitiedoista, tutkimuksen metatiedoista ja tilastoraporteista, jotka koostuvat tilastotaulukoista, luetteloista ja kuvista. Yhdysvaltain FDA:n tapauksessa sähköinen yhteinen tekninen asiakirja (eCTD) on vakiomuoto hakemusten, muutosten, täydennysten ja raporttien lähettämiseen FDA:n biologisten arvioinnin ja tutkimuksen keskukselle (CBER) ja Center for Drug Evaluation and Researchille ( CDER). FDA:n ja japanilaisen PMDA:n osalta lakisääteinen vaatimus on toimittaa taulukkotiedot CDISC Standard Data Bulation Model (SDTM), analyysitiedot CDISC Analysis Dataset Model (ADaM) -mallissa ja kokeilumetatiedot CDISC Define-XML:ssä (perustuu toimintatietomalliin). (ODM)).
Tässä viestissä näytämme, kuinka voimme käyttää RStudioa Amazon Sage Maker tällaisten säännösten mukaisten toimitusten luomiseksi. Tässä postauksessa kuvataan kliinisen tutkimuksen toimitusprosessi, kuinka voimme niellä kliinisten tutkimusten tutkimustietoja, taulukoida ja analysoida tiedot ja sitten luoda tilastoraportteja – yhteenvetotaulukoita, tietoluetteloita ja kuvioita (TLF). Tämän menetelmän avulla lääkeasiakkaat voivat yhdistää saumattomasti AWS-ympäristöönsä tallennettuihin kliinisiin tietoihin, käsitellä niitä R:n avulla ja nopeuttaa kliinisen tutkimuksen tutkimusprosessia.
Lääkkeiden kehitysprosessi
Lääkekehitysprosessi voidaan jakaa laajasti viiteen päävaiheeseen, kuten seuraavasta kuvasta näkyy.
Kestää keskimäärin 10–15 vuotta ja noin 1–3 miljardia dollaria, ennen kuin yksi lääke saa onnistuneen hyväksynnän noin 10,000 XNUMX potentiaalisen molekyylin joukosta. Tutkimuksen alkuvaiheessa (lääkekehitysvaiheessa) tunnistetaan lupaavia lääkekandidaatteja, jotka siirtyvät edelleen prekliiniseen tutkimukseen. Prekliinisen vaiheen aikana tutkijat yrittävät selvittää lääkkeen myrkyllisyyttä suorittamalla vitro kokeet laboratoriossa ja in vivo eläinkokeita. Prekliinisen testauksen jälkeen lääkkeet siirtyvät kliinisen kokeen tutkimusvaiheeseen, jossa niitä on testattava ihmisillä niiden turvallisuuden ja tehokkuuden varmistamiseksi. Tutkijat suunnittelevat kliiniset tutkimukset ja yksityiskohtaisesti tutkimussuunnitelman kliinisen tutkimuksen protokollassa. Ne määrittelevät eri kliinisen tutkimuksen vaiheet – pienistä vaiheen 1 tutkimuksista lääkkeiden turvallisuuden ja annostuksen määrittämiseksi, suurempiin vaiheen 2 tutkimuksiin lääkkeiden tehon ja sivuvaikutusten määrittämiseksi, vielä suurempiin vaiheiden 3 ja 4 tutkimuksiin lääkkeiden tehon, turvallisuuden ja haittavaikutusten seurantaan. Onnistuneiden kliinisten ihmiskokeiden jälkeen lääkesponsori jättää uuden lääkehakemuksen (NDA) lääkkeen markkinoimiseksi. Sääntelyviranomaiset tarkistavat kaikki tiedot, työskentelevät sponsorin kanssa reseptimerkintöjen tiedoissa ja hyväksyvät lääkkeen. Lääkkeen hyväksymisen jälkeen sääntelyvirastot tarkistavat markkinoille saattamisen jälkeiset turvallisuusraportit varmistaakseen tuotteen täydellisen turvallisuuden.
Vuonna 1997 Clinical Data Interchange Standards Consortium (CDISC), globaali voittoa tavoittelematon organisaatio, joka koostuu lääkeyhtiöistä, CRO:ista, biotekniikasta, akateemisista laitoksista, terveydenhuollon tarjoajista ja valtion virastoista, perustettiin vapaaehtoisryhmänä. CDISC on julkaissut tietostandardeja virtaviivaistaakseen tiedonkulkua keräämisestä lähetysten kautta ja helpottaa tiedonvaihtoa kumppanien ja palveluntarjoajien välillä. CDISC on julkaissut seuraavat standardit:
- CDASH (Clinical Data Acquisition Standards Harmonization) – Kerättyjä tietoja koskevat standardit
- SDTM (Study Data Bulation Model) – Taulukkotietojen toimittamista koskevat standardit
- ADaM (analyysitietomalli) – Analyysitietojen standardit
- LÄHETÄ (standardi ei-kliinisen tiedon vaihdolle) – Standardit ei-kliinisille tiedoille
- PRM (Protocol Representation Model) – Protokollan standardit
Nämä standardit voivat auttaa koulutettuja arvioijia analysoimaan tietoja tehokkaammin ja nopeammin vakiotyökalujen avulla, mikä lyhentää lääkkeiden hyväksymisaikoja. Yhdysvaltain FDA:n ja Japanin PMDA:n lakisääteinen vaatimus on toimittaa kaikki taulukkotiedot SDTM-muodossa.
R kliinisten tutkimusten tutkimustoimituksista
SAS ja R ovat kaksi eniten käytettyä tilastoanalyysiohjelmistoa lääketeollisuudessa. Kun CDISC aloitti SDTM-standardien kehittämisen, SAS oli lähes yleisessä käytössä lääketeollisuudessa ja FDA:ssa. R on kuitenkin saavuttamassa valtavaa suosiota nykyään, koska se on avoimen lähdekoodin, ja uusia paketteja ja kirjastoja lisätään jatkuvasti. Opiskelijat käyttävät R:tä ensisijaisesti akateemisissa ja tutkimuksissaan, ja he ottavat tämän R:n tuntemuksen käyttöön töissään. R tarjoaa myös tukea uusille teknologioille, kuten edistyneille syväoppimisen integraatioille.
Pilvipalveluntarjoajista, kuten AWS, on nyt tullut lääkeasiakkaiden suosikkialusta infrastruktuurinsa isännöimiseen. AWS tarjoaa myös hallittuja palveluita, kuten SageMaker, mikä tekee koneoppimismallien (ML) luomisesta, kouluttamisesta ja käyttöönotosta pilvessä vaivatonta. SageMaker mahdollistaa myös pääsyn RStudio IDE:hen mistä tahansa verkkoselaimen kautta. Tässä viestissä kerrotaan, kuinka tilasto-ohjelmoijat ja biostatistit voivat syöttää kliinisen tietonsa R-ympäristöön, kuinka R-koodia voidaan ajaa ja kuinka tulokset tallennetaan. Tarjoamme koodinpätkät, joiden avulla kliinisen kokeen datatutkijat voivat syöttää XPT-tiedostoja R-ympäristöön, luoda R-datakehyksiä SDTM:lle ja ADaM:lle ja lopuksi luoda TLF:n, joka voidaan tallentaa Amazonin yksinkertainen tallennuspalvelu (Amazon S3) esineen säilytysämpäri.
RStudio SageMakerissa
2 AWS yhteistyössä RStudio PBC ilmoitti - yleinen saatavuus RStudio SageMakerissa, alan ensimmäinen täysin hallittu RStudio Workbench IDE pilvessä. Voit nyt tuoda nykyisen RStudio-lisenssisi helposti siirtääksesi itsehallitut RStudio-ympäristösi SageMakeriin muutamalla yksinkertaisella vaiheella. Saat lisätietoja tästä jännittävästä yhteistyöstä tutustumalla RStudion julkaiseminen Amazon SageMakerissa.
RStudio-työpöydän lisäksi RStudio-paketti R-kehittäjille tarjoaa myös RStudio Connectin ja RStudio Package Managerin. RStudio Connect on suunniteltu antamaan datatieteilijöille mahdollisuus julkaista oivalluksia, kojetauluja ja verkkosovelluksia. Sen avulla on helppoa jakaa tietotieteilijöiden monimutkaisesta työstä saatuja ML- ja datatieteen näkemyksiä ja antaa ne päättäjien käsiin. RStudio Connect tekee myös sisällön ylläpidosta ja hallinnasta yksinkertaista ja skaalautuvaa laajaan kulutukseen.
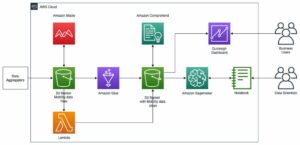
Ratkaisun yleiskatsaus
Seuraavissa osioissa keskustelemme siitä, kuinka voimme tuoda raakadataa etävarastosta tai S3-säilöstä SageMakerin RStudiossa. On myös mahdollista muodostaa yhteys suoraan Amazon Relational Database -palvelu (Amazon RDS) ja tietovarastot, kuten Amazonin punainen siirto (Ks. R:n yhdistäminen Amazon Redshiftiin) suoraan RStudiosta; tämä ei kuitenkaan kuulu tämän viestin piiriin. Kun tiedot on haettu muutamasta eri lähteestä, käsittelemme sen ja luomme R tietokehystä taulukolle. Sitten muunnamme taulukon datakehyksen RTF-tiedostoksi ja tallennamme tulokset takaisin S3-ämpäriin. Näitä tulosteita voidaan sitten mahdollisesti käyttää viranomaistoimituksiin edellyttäen, että viestissä käytetyt R-paketit on validoitu käytettäviksi viranomaistoimituksia varten.
Ota RStudio käyttöön SageMakerissa
Katso ohjeet RStudion määrittämisestä SageMakerissa ympäristössäsi Aloita RStudion käyttö SageMakerissa. Varmista, että SageMakerin RStudion suoritusroolilla on pääsy tietojen lataamiseen ja lähettämiseen S3-säilöyn, johon tiedot on tallennettu. Lisätietoja R-pakettien hallinnasta ja analyysin julkaisemisesta SageMakerin RStudion avulla on kohdassa SageMaker for Data Scientists -palvelussa julkistetaan täysin hallinnoitu RStudio.
Siirrä tiedot RStudioon
Tässä vaiheessa haemme tietoja eri lähteistä, jotta ne ovat käytettävissä R-istunnossamme. Tuomme tietoja SAS XPT -muodossa; prosessi on kuitenkin samanlainen, jos haluat syöttää tietoja muissa muodoissa. Yksi RStudion käytön eduista SageMakerissa on, että jos lähdetiedot on tallennettu AWS-tileillesi, SageMaker voi käyttää tietoja natiivisti käyttämällä AWS-henkilöllisyyden ja käyttöoikeuksien hallinta (IAM) roolit.
Käytä etätietovarastoon tallennettuja tietoja
Tässä vaiheessa tuomme ADaM-tiedot kohteesta FDA:n GitHub-arkisto. Luomme paikallisen hakemiston nimeltä data RStudio-ympäristössä tietojen tallentamiseen ja demografisten tietojen lataamiseen (dm.xpt) etävarastosta. Tässä yhteydessä paikallinen hakemisto viittaa yksityiseen Amazon EFS -tallennustilaan luotuun hakemistoon, joka on oletuksena liitetty R-istuntoympäristöösi. Katso seuraava koodi:
Kun tämä vaihe on valmis, näet dm.xpt ladataan navigoimalla kohteeseen Asiakirjat, tiedot, dm.xpt.
Käytä Amazon S3:een tallennettuja tietoja
Tässä vaiheessa lataamme tilillemme S3-ämpäriin tallennetut tiedot. Olemme kopioineet sisällön FDA:n GitHub-arkistosta S3-säihöön nimeltä aws-sagemaker-rstudio tälle esimerkille. Katso seuraava koodi:
Kun vaihe on valmis, näet pp.xpt ladataan navigoimalla kohteeseen Asiakirjat, tiedot, pp.xpt.
Käsittele XPT-tietoja
Nyt kun meillä on SAS XPT -tiedostoja saatavilla R-ympäristössä, meidän on muunnettava ne R-tietokehyksiksi ja käsiteltävä ne. Käytämme haven kirjasto XPT-tiedostojen lukemiseen. Yhdistämme CDISC SDTM -tietojoukot dm ja pp luodaksesi ADPP-tietojoukon. Sitten luomme yhteenvetotilastotaulukon käyttämällä ADPP-tietokehystä. Yhteenvetotaulukko viedään sitten RTF-muodossa.
Ensin XPT-tiedostot luetaan käyttämällä read_xpt satamakirjaston toiminto. Sitten analyysitietojoukko luodaan käyttämällä sqldf toiminto sqldf kirjasto. Katso seuraava koodi:
Sitten luodaan lähtötietokehys käyttämällä funktioita kohteesta Tplyr ja dplyr kirjastot:
Lähtötietokehys tallennetaan sitten RTF-tiedostona RStudio-ympäristön tulostuskansioon:
Lataa lähdöt Amazon S3:een
Kun tulos on luotu, laitamme tiedot takaisin S3-ämpäriin. Voimme saavuttaa tämän luomalla SageMaker-istunnon uudelleen, jos istunto ei ole jo aktiivinen, ja lataamalla tuloskansion sisällön S3-säihöön käyttämällä session$upload_data toiminto:
Näiden vaiheiden avulla olemme saaneet tietoja, käsitelleet niitä ja ladaneet tulokset toimitettavaksi valvontaviranomaisille.
Puhdistaa
Sinun on lopetettava nykyinen istunto välttääksesi tahattomat kulut. Valitse sivun oikeasta yläkulmasta virtakuvake. Tämä pysäyttää automaattisesti taustalla olevan ilmentymän ja lopettaa siten tahattomien laskentakulujen syntymisen.
Haasteet
Viestissä on kuvattu vaiheet S3-säilöyn tai etävarastosta tallennetun raakadatan nielemiseen. Kliiniseen tutkimukseen on kuitenkin olemassa monia muita raakadatalähteitä, pääasiassa eCRF-tiedot (elektroniset tapausraporttilomakkeet), jotka on tallennettu EDC-järjestelmiin (elektroninen tiedonkeruu), kuten Oracle Clinical, Medidata Rave, OpenClinica tai Snowflake; laboratoriotiedot; tiedot eCOA:sta (kliininen tulosarviointi) ja ePRO:sta (elektroniset potilaiden raportoimat tulokset); reaalimaailman data sovelluksista ja lääketieteellisistä laitteista; ja sairaaloiden sähköiset terveystiedot (EHR). Merkittävää esikäsittelyä on tehtävä ennen kuin nämä tiedot voidaan ottaa käyttöön viranomaistoimituksia varten. Liitinten rakentaminen eri tietolähteisiin ja niiden kerääminen keskitettyyn tietovarastoon (CDR) tai kliiniseen datajärveen, samalla kun ylläpidetään asianmukaista pääsynhallintaa, asettaa merkittäviä haasteita.
Toinen ratkaiseva haaste on säännösten noudattaminen. Viranomaistulosten luomiseen käytettävän tietokonejärjestelmän on oltava asianmukaisten määräysten, kuten 21 CFR Part 11, HIPAA, GDPR tai muiden GxP-vaatimusten tai ICH-ohjeiden mukainen. Tämä tarkoittaa työskentelyä validoidussa ja pätevässä ympäristössä, jossa on käytössä pääsyn, suojauksen, varmuuskopioinnin ja tarkastettavuuden hallinta. Tämä tarkoittaa myös, että kaikki R-paketit, joita käytetään säädöstenmukaisten toimitustulosten luomiseen, on validoitava ennen käyttöä.
Yhteenveto
Tässä viestissä näimme, että jotkin eCTD-lähetyksen tärkeimmistä suorituksista olivat CDISC SDTM, ADaM-tietojoukot ja TLF. Tässä viestissä esitettiin vaiheet, jotka tarvitaan näiden säädösten mukaisten toimitusten luomiseen siirtämällä ensin tiedot muutamasta lähteestä SageMakerin RStudioon. Sitten näimme kuinka voimme käsitellä syötetyt tiedot XPT-muodossa; muuntaa se R-datakehyksiksi luodaksesi SDTM, ADaM ja TLF; ja lopuksi lataa tulokset S3-ämpäriin.
Toivomme, että postauksessa esitettyjen laajojen ideoiden avulla tilasto-ohjelmoijat ja biostatistikot voivat helposti visualisoida kliinisen kokeen tutkimustietojen lataamisen, käsittelyn ja analysoinnin päästä päähän SageMakerin RStudioon ja käyttää oppimista mukautetun työnkulku, joka sopii säädöstenmukaisille toimituksillesi.
Tuleeko mieleen muita sovelluksia, joissa RStudio voisi auttaa tutkijoita, tilastotieteilijöitä ja R-ohjelmoijia helpottamaan elämäänsä? Haluaisimme kuulla ideoistasi! Ja jos sinulla on kysyttävää, jaa ne kommenttiosiossa.
Esittelymateriaalit
Lisätietoja on seuraavissa linkeissä:
Tietoja kirjoittajista
 Rohit Banga on maailmanlaajuisen kliinisen kehityksen alan asiantuntija Lontoossa, Isossa-Britanniassa. Hän on koulutukseltaan biostatistikko ja auttaa Healthcare- ja LifeScience-asiakkaita ottamaan käyttöön innovatiivisia kliinisiä kehitysratkaisuja AWS:ssä. Hän on intohimoinen siitä, kuinka datatiedettä, tekoälyä/ML:ää ja uusia teknologioita voidaan käyttää ratkaisemaan todellisia liiketoimintaongelmia Healthcare- ja LifeScience-alalla. Vapaa-ajallaan Rohit harrastaa hiihtoa, grillausta ja aikaa perheen ja ystävien kanssa.
Rohit Banga on maailmanlaajuisen kliinisen kehityksen alan asiantuntija Lontoossa, Isossa-Britanniassa. Hän on koulutukseltaan biostatistikko ja auttaa Healthcare- ja LifeScience-asiakkaita ottamaan käyttöön innovatiivisia kliinisiä kehitysratkaisuja AWS:ssä. Hän on intohimoinen siitä, kuinka datatiedettä, tekoälyä/ML:ää ja uusia teknologioita voidaan käyttää ratkaisemaan todellisia liiketoimintaongelmia Healthcare- ja LifeScience-alalla. Vapaa-ajallaan Rohit harrastaa hiihtoa, grillausta ja aikaa perheen ja ystävien kanssa.
 Georgios Schinas on AI/ML:n erikoisratkaisuarkkitehti EMEA-alueella. Hänen kotipaikkansa on Lontoossa ja hän tekee läheistä yhteistyötä Iso-Britannian ja Irlannin asiakkaiden kanssa. Georgios auttaa asiakkaita suunnittelemaan ja ottamaan käyttöön koneoppimissovelluksia AWS:n tuotannossa. Hän on erityisen kiinnostunut MLOps-käytännöistä ja antaa asiakkaille mahdollisuuden suorittaa koneoppimista mittakaavassa. Vapaa-ajallaan hän nauttii matkustamisesta, ruoanlaitosta ja viettämisestä ystävien ja perheen kanssa.
Georgios Schinas on AI/ML:n erikoisratkaisuarkkitehti EMEA-alueella. Hänen kotipaikkansa on Lontoossa ja hän tekee läheistä yhteistyötä Iso-Britannian ja Irlannin asiakkaiden kanssa. Georgios auttaa asiakkaita suunnittelemaan ja ottamaan käyttöön koneoppimissovelluksia AWS:n tuotannossa. Hän on erityisen kiinnostunut MLOps-käytännöistä ja antaa asiakkaille mahdollisuuden suorittaa koneoppimista mittakaavassa. Vapaa-ajallaan hän nauttii matkustamisesta, ruoanlaitosta ja viettämisestä ystävien ja perheen kanssa.
- AI
- ai taide
- ai taiteen generaattori
- ai robotti
- Amazon Sage Maker
- Amazon Simple Storage Service (S3)
- tekoäly
- tekoälyn sertifiointi
- tekoäly pankkitoiminnassa
- tekoäly robotti
- tekoälyrobotit
- tekoälyohjelmisto
- AWS-koneoppiminen
- blockchain
- blockchain-konferenssi ai
- coingenius
- keskustelullinen tekoäly
- kryptokonferenssi ai
- dall's
- syvä oppiminen
- google ai
- Keskitaso (200)
- Life Sciences
- koneoppiminen
- Platon
- plato ai
- Platonin tietotieto
- Platon peli
- PlatonData
- platopeliä
- mittakaava ai
- syntaksi
- zephyrnet