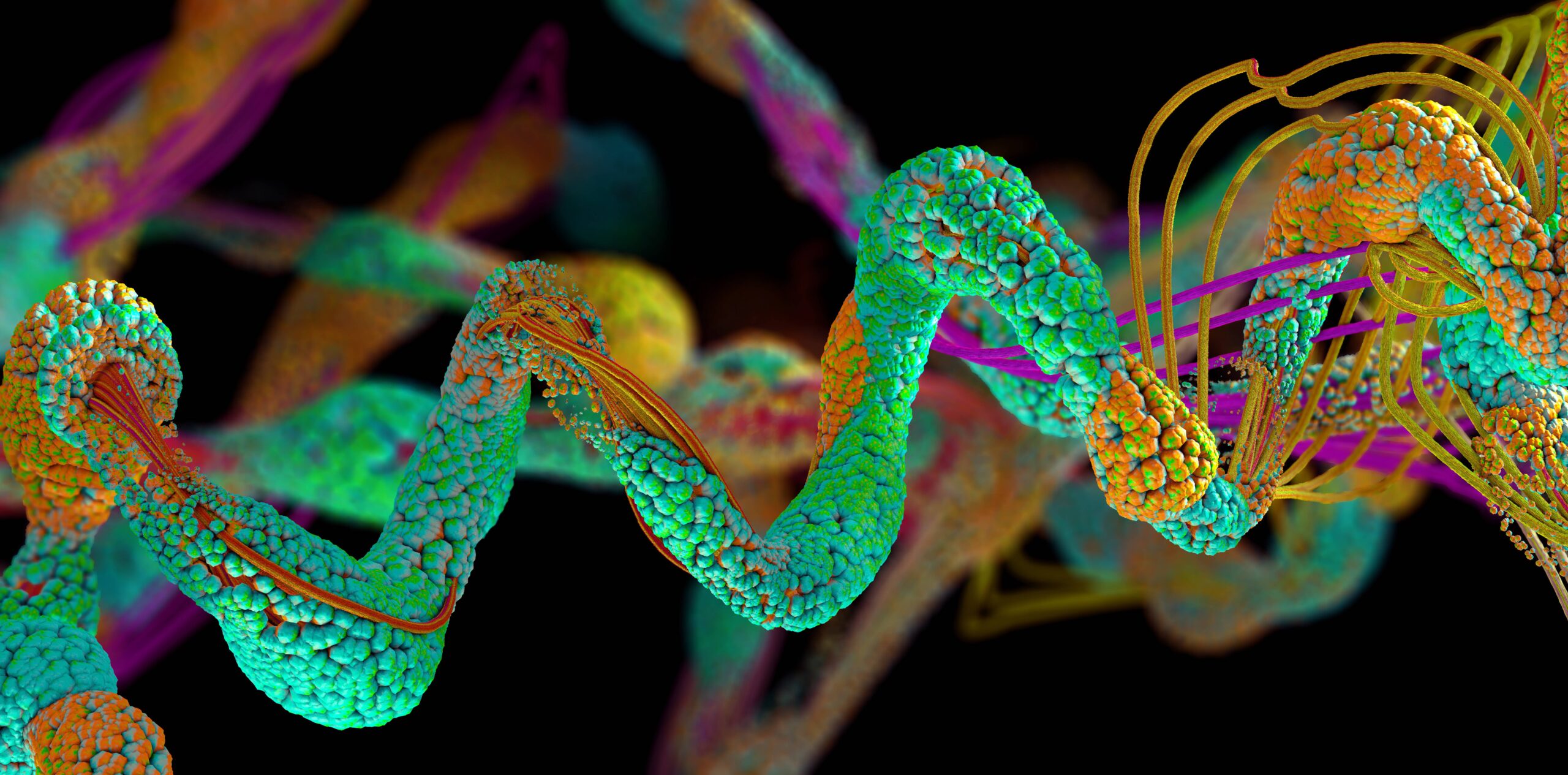
Le modèle de repliement des protéines alimenté par l'IA AlphaFold a prédit plus de 200 millions de protéines, presque toutes ces structures connues de la science, a déclaré DeepMind jeudi.
Les protéines sont des molécules biologiques complexes produites dans des organismes vivants à partir d'instructions stockées dans l'ADN. Fabriquées à partir de pas moins de 20 types d'acides aminés, ces chaînes à l'échelle nanométrique effectuent des tâches cellulaires vitales pour effectuer toutes sortes de fonctions corporelles. Connaître la forme tridimensionnelle des protéines est important car sa structure physique fournit des indices sur la façon dont elle se comporte et à quoi elle sert, ce qui nous aide à faire des choses comme développer des médicaments et créer des protéines imitatrices pour ceux qui en manquent.
Certaines protéines sont utiles, telles que celles impliquées dans la digestion des aliments, tandis que d'autres peuvent être nocives, telles que celles impliquées dans la croissance des tumeurs. Il est cependant difficile de comprendre leurs formes sinueuses compliquées. Les biologistes moléculaires peuvent passer des années à mener des expériences pour déchiffrer la structure d'une protéine, et AlphaFold peut le faire en quelques minutes, selon la taille de la molécule, à partir de la composition en acides aminés.
AlphaFold a été formé sur des centaines de milliers de structures protéiques connues et a appris les relations entre les acides aminés constitutifs et les formes globales finales. Étant donné une séquence d'acides aminés d'entrée arbitraire, le modèle peut prédire une structure de protéine 3D. Maintenant, le modèle a prédit presque toutes les structures protéiques connues de la science.
En collaboration avec l'Institut européen de bioinformatique, DeepMind a élargi sa Base de données sur la structure des protéines AlphaFold contenir plus de 200 millions de formes 3D de protéines, des animaux aux plantes, des bactéries aux virus - une augmentation de plus de 200 fois, passant de près d'un million de molécules à au moins 200 millions de molécules en seulement un an.
"Nous espérions que cette ressource révolutionnaire aiderait à accélérer la recherche et la découverte scientifiques à l'échelle mondiale, et que d'autres équipes pourraient apprendre et s'appuyer sur les progrès que nous avons réalisés avec AlphaFold pour créer de nouvelles percées", Demis Hassibis, co-fondateur et PDG de DeepMind, a affirmé Valérie Plante. dans une déclaration jeudi.
« Cet espoir est devenu réalité bien plus rapidement que nous n'avions osé le rêver. À peine douze mois plus tard, AlphaFold a été consulté par plus d'un demi-million de chercheurs et utilisé pour accélérer les progrès sur d'importants problèmes du monde réel allant de la pollution plastique à la résistance aux antibiotiques.
Le registre a demandé à DeepMind des commentaires supplémentaires.
AlphaFold a également montré un grand potentiel pour la conception de nouveaux médicaments. Les structures aident les scientifiques à comprendre les composés chimiques qui peuvent se lier aux protéines cibles pour les traiter ou les empêcher d'exercer des fonctions pathologiques. Des entreprises comme Insilco Medicine ont expérimenté avec le modèle pour découvrir de nouveaux médicaments ; Le PDG Alex Zhavoronkov a déclaré Le registre que le processus est beaucoup plus compliqué que vous ne le pensez et comporte plusieurs étapes.
La précision des prédictions d'AlphaFold n'est pas claire. La structure en forme de ruban d'une protéine change souvent de forme lorsqu'elle interagit avec un médicament, ce qu'AlphaFold ne peut pas aider les scientifiques car il n'est pas formé à cela. Zhavoronkov a déclaré que le modèle est une "percée assez remarquable", mais se méfiait de tout le battage médiatique.
"Jusqu'à ce que nous voyions une structure pour une nouvelle cible dans une grande maladie obtenue via AlphaFold sans aucune expérience supplémentaire, une molécule conçue à l'aide de l'IA - ou d'autres méthodes - utilisant cette structure prédite, synthétisée et testée jusqu'au bout, puis publiée dans un journal de haut niveau – [nous pouvons] alors célébrer.
Les grandes entreprises pharmaceutiques veulent voir des molécules conçues à l'aide d'outils d'IA comme AlphaFold réellement testées sur des souris et des humains. "Les réalisations algorithmiques pures ne sont pas précieuses pour les sociétés pharmaceutiques et en particulier pour les patients", a ajouté Zhavoronkov.
Fabio Urbina, scientifique principal chez Collaboration Pharmaceuticals, une startup utilisant des algorithmes d'apprentissage automatique pour développer des médicaments pour les maladies génétiques rares, a déclaré qu'AlphaFold ne s'est pas encore avéré utile dans ses recherches. Urbina utilise une technique différente et se concentre davantage sur la structure d'un nouveau médicament potentiel plutôt que sur une protéine cible.
Il reste à voir si les structures protéiques seront suffisamment utiles… pour nous aider à découvrir de nouveaux médicaments potentiels pour les maladies rares
«C'est pour plusieurs raisons; les structures protéiques de nombreuses cibles médicamenteuses n'étaient souvent pas facilement disponibles pour les chercheurs, et les informations sur les protéines ne semblaient pas aider les premiers modèles d'apprentissage automatique à améliorer leur pouvoir prédictif par une marge significative », a-t-il déclaré. Le registre.
"Je suis prudemment optimiste qu'AlphaFold a essentiellement" résolu "le premier problème, mais il reste à voir si les structures protéiques seront suffisamment utiles pour notre application en aval d'amélioration de la puissance prédictive de l'apprentissage automatique pour nous aider à découvrir de nouveaux médicaments potentiels. pour les maladies rares. Cependant, nous avons de plus en plus vu les informations structurelles des protéines prises en compte dans le cadre des nouvelles méthodes d'apprentissage automatique, et nous avons pensé à faire de même.
Rendre disponible une base de données avec presque toutes les structures protéiques connues, comme DeepMind l'a promis, signifie que davantage de scientifiques auront les ressources pour expérimenter et construire des modèles d'IA plus puissants, a déclaré Urbina. «Je suis prudemment optimiste, mais avec toute la bibliothèque de structures protéiques disponible, je dirais qu'il y a de fortes chances que les structures AlphaFold soient incorporées dans certains de nos modèles d'apprentissage automatique et puissent finalement nous aider à découvrir de nouvelles thérapies. ” ®
- AI
- art de l'IA
- générateur d'art ai
- robot IA
- intelligence artificielle
- certification en intelligence artificielle
- intelligence artificielle en banque
- robot d'intelligence artificielle
- robots d'intelligence artificielle
- logiciel d'intelligence artificielle
- blockchain
- conférence blockchain ai
- cognitif
- intelligence artificielle conversationnelle
- crypto conférence ai
- de dall
- l'apprentissage en profondeur
- google ai
- machine learning
- Platon
- platon ai
- Intelligence des données Platon
- Jeu de Platon
- PlatonDonnées
- jeu de platogamie
- échelle ai
- syntaxe
- Le registre
- zéphyrnet