La transcriptomique spatiale et la protéomique fournissent des informations complémentaires qui ont transformé notre compréhension des processus biologiques complexes. Cependant, l'intégration expérimentale de ces modalités est limitée.
Une nouvelle méthode appelée Spatial PrOtein and Transcriptome Sequencing (SPOTS) peut éclairer les identités et les activités des cellules dans un organe ou une tumeur avec une résolution sans précédent. Développé par des chercheurs de Weill Cornell médecine, NewYork-Presbyterian et le New York Genome Center, SPOTS peut éclairer les identités et les activités des cellules d'un organe ou d'une tumeur avec une résolution sans précédent.
La technique préserve les informations sur l'emplacement précis des cellules tout en enregistrant les schémas d'activité des gènes et la présence de substances essentielles. protéines dans les cellules à travers des échantillons de tissus. Cela permet de créer des « cartes » complexes et riches en données des organes, y compris les organes malades et les tumeurs, ce qui peut être extrêmement utile dans la recherche fondamentale et clinique.
Co-auteur principal de l'étude, le Dr Dan Landau, professeur agrégé de médecine à la Division d'hématologie et d'oncologie médicale et membre du Sandra et Edward Meyer Cancer Center de Weill Cornell Medicine et membre principal du corps professoral du New York Genome Center, dit, "Cette technologie est passionnante car elle nous permet de cartographier l'organisation spatiale des tissus, y compris les types de cellules, les activités cellulaires et les interactions cellule à cellule, comme jamais auparavant."
La nouvelle approche fait partie d’une initiative plus vaste menée par des scientifiques et des ingénieurs visant à créer des moyens plus efficaces de « voir » au niveau microscopique le fonctionnement des organes et des tissus. Ces dernières années ont vu des progrès significatifs dans la recherche, notamment dans les méthodes de profilage de l'activité génétique et d'autres couches de données dans des cellules individuelles ou de petits groupes de cellules. cellules. Les informations concernant les emplacements d'origine des cellules profilées dans les tissus doivent cependant être récupérées, car ces approches nécessitent fréquemment la décomposition des tissus et la séparation des cellules de leurs voisines. La nouvelle technique enregistre également ces données spatiales, et ce avec une excellente résolution.
La méthode est basée en partie sur la technologie 10x Genomics existante. Il utilise des lames de verre adaptées à l'imagerie d'échantillons de tissus avec des méthodes pathologiques ordinaires basées sur un microscope, mais est également recouverte de milliers de molécules sondes particulières.
Le « code-barres » chimique de chaque molécule sonde identifie son emplacement bidimensionnel sur la lame. Les molécules sondes sur la lame saisissent le ARN messagers (ARNm), essentiellement les transcrits de gènes activés, provenant de cellules voisines, lorsqu'un échantillon de tissu finement tranché est placé sur la lame, ses cellules deviennent perméables. Des anticorps de conception sont utilisés dans la procédure et se fixent aux molécules sondes et aux protéines d'intérêt uniques dans le tissu.
Les chercheurs peuvent identifier rapidement et automatiquement les ARNm collectés et les protéines choisies et les cartographier avec précision à leur emplacement d'origine dans l'échantillon de tissu. Les cartes produites peuvent être considérées indépendamment ou par rapport à l'imagerie pathologique de routine de l'échantillon.
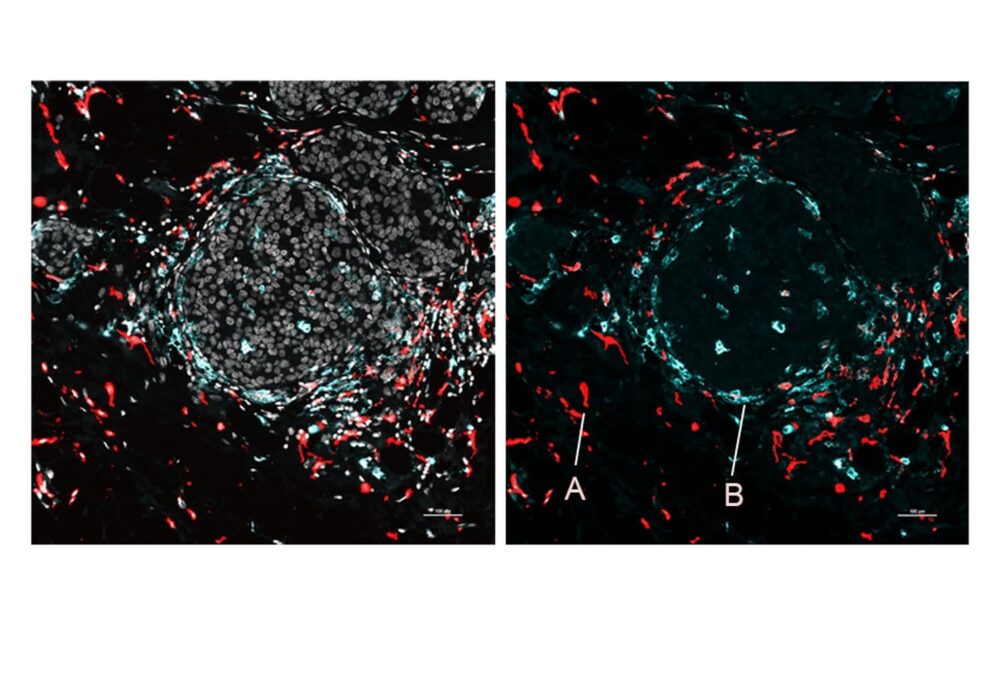
Sur les tissus d'une rate de souris saine, l'équipe a utilisé SPOTS pour montrer l'architecture fonctionnelle complexe de cet organe, y compris des groupes de différents types de cellules, leurs états fonctionnels et la manière dont ces états ont été modifiés en fonction de l'emplacement des cellules.
Les chercheurs ont également utilisé SPOTS pour cartographier la structure cellulaire d'une tumeur du sein de souris, soulignant ainsi son potentiel d'utilisation dans recherche sur le cancer. La carte générée montrait des macrophages et des cellules immunitaires dans deux états différents, chacun indiqué par un marqueur protéique différent : une condition était active et combattait les tumeurs, tandis que l'autre était immunosuppressive et établissait une barrière pour protéger la tumeur.
Dr Landau, oncologue au NewYork-Presbyterian/Weill Cornell Medical Center, a affirmé Valérie Plante., "Nous avons pu constater que ces deux sous-ensembles de macrophages se trouvent dans différentes zones de la tumeur et interagissent avec différentes cellules, et cette différence dans le microenvironnement est probablement à l'origine de leurs états d'activité distincts."
"De tels détails sur l'environnement immunitaire de la tumeur - des détails qui ne peuvent souvent pas être résolus en raison de la rareté des cellules immunitaires dans les tumeurs - pourraient aider à expliquer pourquoi certains patients réagissent à thérapie de renforcement immunitaire et d’autres non, et pourraient ainsi éclairer la conception de futures immunothérapies.
« Cette version initiale de SPOTS a une résolution spatiale telle que chaque « pixel » de l’ensemble de données résultant résume les informations sur l’activité des gènes pour au moins plusieurs cellules. Cependant, les chercheurs espèrent bientôt limiter cette résolution à des cellules uniques tout en ajoutant d’autres couches d’informations cellulaires clés.
Journal de référence:
- Ben-Chetrit, N., Niu, X., Swett, AD et al. Intégration du profilage spatial du transcriptome entier avec des marqueurs protéiques. Nat Biotechnologie (2023). EST CE QUE JE: 10.1038/s41587-022-01536-3