AmazonSageMakerパイプライン 機械学習 (ML) のユースケース向けに設計された継続的インテグレーションおよび継続的デリバリー (CI/CD) サービスです。 これを使用して、エンドツーエンドの ML ワークフローを作成、自動化、および管理できます。 時間、労力、リソースを必要とする ML プロセスの各ステップを調整するという課題に取り組みます。 使いやすくするために、必要に応じてカスタマイズできる複数のテンプレートが用意されています。
フル マネージドの画像およびビデオ分析サービスも、コンピューター ビジョン ソリューションの採用を加速させています。 AWS は、事前にトレーニングされた完全マネージド型の AWS AI サービスを提供しています。 Amazonの再認識 API 呼び出しを使用してコンピューター ビジョン アプリケーションに統合でき、ML の経験は必要ありません。 に画像を提供するだけです。 Amazon 認識 API また、事前定義されたラベルに従って必要なオブジェクトを識別できます。 また、ユース ケースに固有のカスタム ラベルを提供し、ML の専門知識を必要とするオーバーヘッドをほとんどまたはまったく必要とせずに、カスタマイズされたコンピューター ビジョン モデルを構築することもできます。
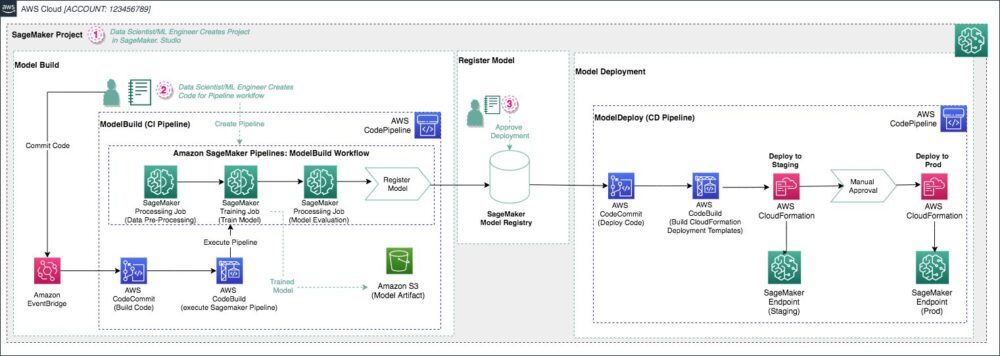
この投稿では、特定のコンピューター ビジョンの問題である皮膚病変の分類に取り組み、既存のテンプレートをカスタマイズしてこのタスクに合わせてパイプラインを使用します。 正確な皮膚病変の分類は、がん疾患の早期診断に役立ちます。 しかし、異なる種類の皮膚病変の間には高い類似性があるため、医療分野では困難な作業です。 パイプラインを使用すると、さまざまな既存のモデルとアルゴリズムを利用して、最小限の労力と時間でエンド ツー エンドの実稼働パイプラインを確立できます。
ソリューションの概要
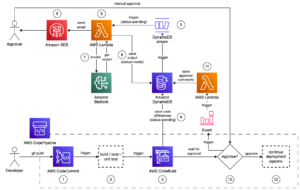
この投稿では、Pipelines を使用してエンドツーエンドのパイプラインを構築し、一般的な色素性皮膚病変の皮膚鏡画像を分類します。 私たちは、 Amazon SageMakerスタジオ プロジェクトテンプレート モデルを構築、トレーニング、および展開するためのMLOpsテンプレート および次のコード GitHubリポジトリ. 結果のアーキテクチャを次の図に示します。
このパイプラインでは、10000 の皮膚鏡画像で構成される HAM10000 (「Human Against Machine with 10,015 training images」) データセットを使用します。 当面のタスクは、コンピューター ビジョンの分野におけるマルチクラス分類です。 このデータセットは、色素性病変の領域で最も重要な XNUMX つの診断カテゴリを示しています: 光線性角化症および上皮内癌またはボーエン病 (akiec)、基底細胞がん(bcc)、良性角化症様病変(日光黒子または脂漏性角化症および扁平苔癬のような角化症、 bkl)、皮膚線維腫(df)、メラノーマ(mel)、メラノサイト母斑 (nv)、および血管病変(血管腫、角化血管腫、化膿性肉芽腫および出血、 vasc).
モデルの入力の形式については、 RecordIO フォーマット。 これは、継続的な読み取りのために画像データをまとめて保存するコンパクトな形式であるため、より高速で効率的なトレーニングを行うことができます。 さらに、HAM10000 データセットを使用する際の課題の XNUMX つは、クラスの不均衡です。 次の表は、クラスの分布を示しています。
| CLASS | アキエック | BCC | BKL | df | 蜂蜜 | nv | バスク |
| 画像数 | 327 | 514 | 1099 | 115 | 1113 | 6705 | 142 |
| トータル | 10015 | ||||||
この問題に対処するために、ランダムな変換 (トリミング、反転、ミラーリング、回転など) を使用してデータセットを拡張し、すべてのクラスがほぼ同じ数の画像を持つようにします。
この前処理ステップでは MXNet と OpenCV を使用するため、ビルド済みの MXNet コンテナー イメージを使用します。 残りの依存関係は、 requirements.txt ファイル。 カスタム イメージを作成して使用する場合は、 を参照してください。 イメージ構築CI / CDパイプラインを使用してAmazonSageMakerプロジェクトを作成します.
トレーニング ステップでは、SageMaker 組み込みの Scikit Docker イメージから利用可能な推定器を使用してイメージを分類し、パラメーターを次のように設定します。
コンテナ イメージの詳細については、次を参照してください。 画像分類アルゴリズム.
スタジオ プロジェクトを作成する
Studio のセットアップ方法の詳細については、次を参照してください。 クイックセットアップを使用して Amazon SageMaker ドメインにオンボード. プロジェクトを作成するには、次の手順を実行します。
- Studio で、 プロジェクト のメニュー SageMakerリソース メニュー。
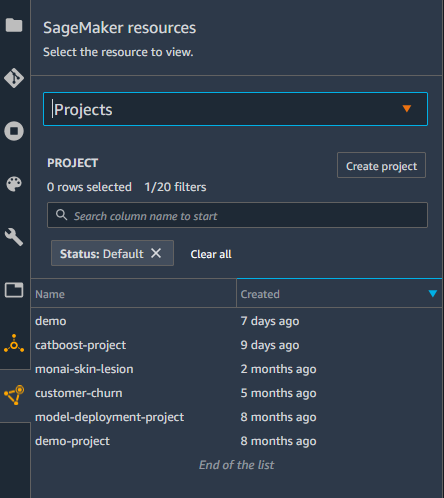
プロジェクトページで、事前設定された SageMaker MLOps テンプレートを起動できます。 - 選択する モデルの構築、トレーニング、および展開のためのMLOpsテンプレート.
- 選択する プロジェクトテンプレートを選択します.
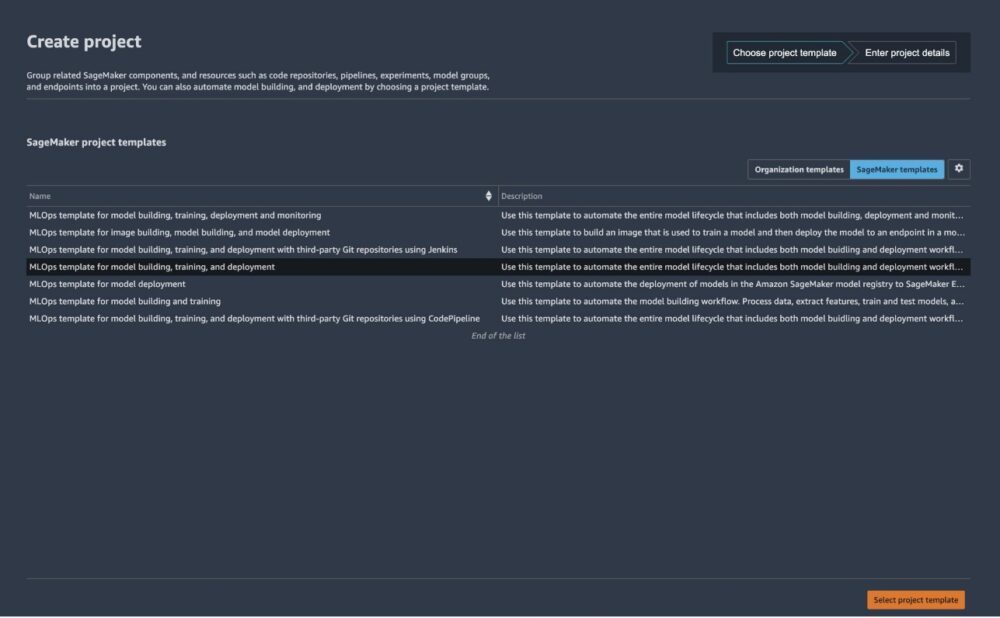
- プロジェクト名と簡単な説明を入力します。
- 選択する プロジェクトを作成する.
プロジェクトの作成には数分かかります。
データセットを準備する
データセットを準備するには、次の手順を実行します。
- に行く ハーバード・データバース.
- 選択する データセットへのアクセスをクリックし、クリエイティブ コモンズ 表示 - 非営利 4.0 国際パブリック ライセンスのライセンスを確認してください。
- ライセンスに同意する場合は、 元の形式の郵便番号 ZIPファイルをダウンロードします。
- 作る Amazon シンプル ストレージ サービス (Amazon S3) バケットを開き、で始まる名前を選択します
sagemaker(これにより、SageMaker は追加のアクセス許可なしでバケットにアクセスできます)。 - セキュリティのベスト プラクティスとして、アクセス ログと暗号化を有効にすることができます。
- アップロード
dataverse_files.zipバケツに。 - 後で使用するために S3 バケットのパスを保存します。
- 後で使用するために、データを保存したバケットの名前と、後続のフォルダーの名前を書き留めます。
データ前処理の準備
前処理ステップで MXNet と OpenCV を使用しているため、ビルド済みの MXNet Docker イメージを使用し、残りの依存関係を requirements.txt ファイル。 これを行うには、それをコピーして下に貼り付ける必要があります pipelines/skin セクションに sagemaker--modelbuild リポジトリ。 さらに、 MANIFEST.in と同じレベルのファイル setup.py、Pythonに含めるように指示します requirements.txt ファイル。 詳細については、 MANIFEST.in、 参照する MANIFEST.in を使用してソース配布にファイルを含める. どちらのファイルも GitHubリポジトリ.
パイプライン テンプレートを変更する
Pipelines テンプレートを更新するには、次の手順を実行します。
- デフォルトのバケット内にフォルダーを作成します。
- Studio 実行ロールが、デフォルトのバケットとデータセットを含むバケットにアクセスできることを確認してください。
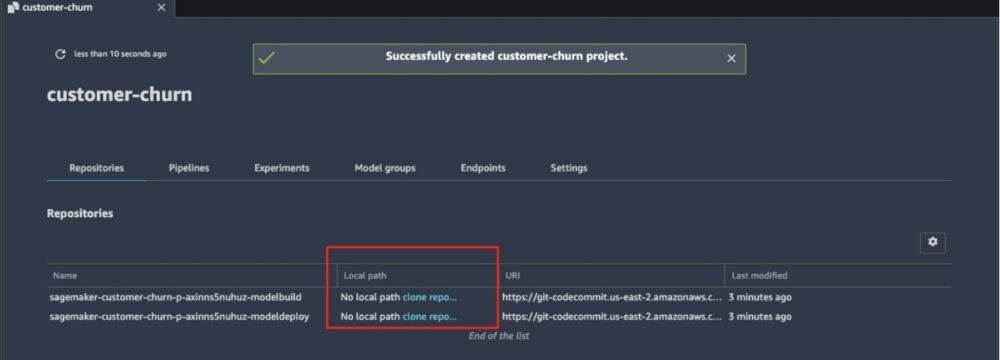
- プロジェクトのリストから、作成したプロジェクトを選択します。
- ソフトウェア設定ページで、下図のように リポジトリ タブで、ハイパーリンクを選択してローカルに複製します AWS コードコミット リポジトリをローカルの Studio インスタンスにコピーします。
- に移動します
pipelines内のディレクトリsagemaker--modelbuildディレクトリの名前を変更しますabaloneへのディレクトリskin. - Video Cloud Studioで
codebuild-buildspec.yml内のファイルsagemaker--modelbuildディレクトリから実行パイプライン パスを変更します。run-pipeline —module-name pipelines.abalone.pipeline(15 行目) を次のように変更します。 - ファイルを保存します。
- ファイルを置き換える
pipelines.py,preprocess.py、およびevaluate.pyからのファイルを含むパイプライン ディレクトリに GitHubリポジトリ.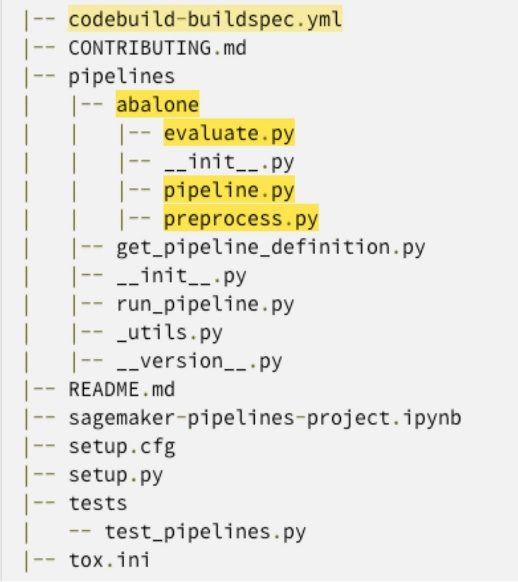
- 更新する
preprocess.pyS183 の場所 (SKIN_CANCER_BUCKET) とフォルダ名 (SKIN_CANCER_BUCKET_PATH) をアップロードした場所dataverse_files.zip記録:skin_cancer_bucket=””skin_cancer_bucket_path=””skin_cancer_files=””skin_cancer_files_ext=””
前の例では、データセットは次の場所に保存されます。 s3://monai-bucket-skin-cancer/skin_cancer_bucket_prefix/dataverse_files.zip.
パイプラインの実行をトリガーする
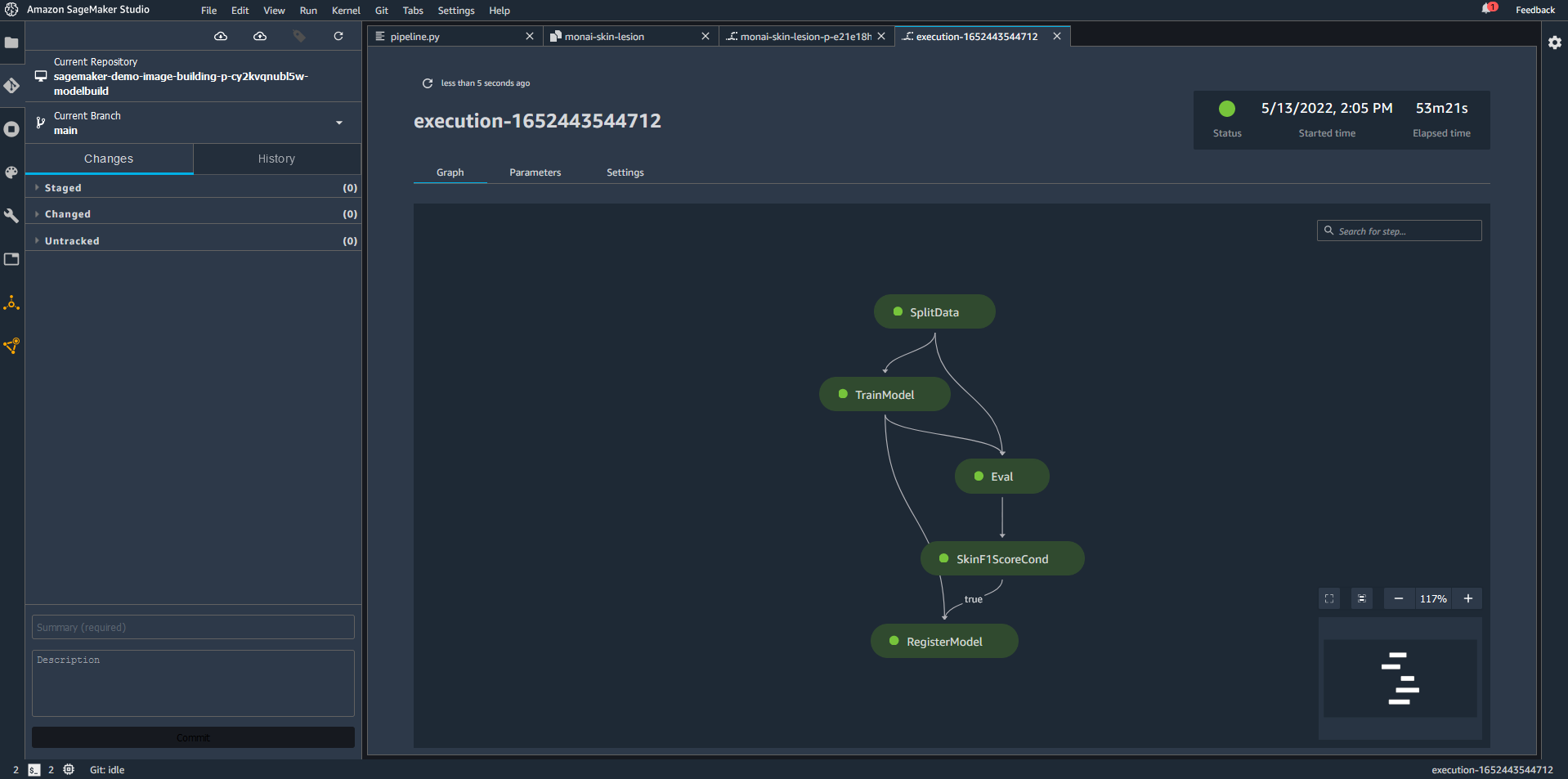
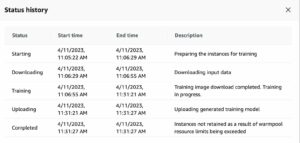
コミットされた変更を CodeCommit リポジトリにプッシュすると (Studio ソース管理タブで実行)、新しいパイプラインの実行がトリガーされます。 アマゾンイベントブリッジ コミットのイベント モニター。 SageMaker プロジェクト内のパイプラインを選択することで、実行を監視できます。 次のスクリーンショットは、正常に実行されたパイプラインの例を示しています。
- 変更をコミットするには、左ペインの Git セクションに移動します。
- 関連するすべての変更をステージングします。 追跡する必要はありません
-checkpointファイル。 にエントリを追加できます。.gitignoreファイル*checkpoint.*それらを無視します。 - 概要、名前、メールアドレスを提供して、変更をコミットします。
- 変更をプッシュします。
- プロジェクトに戻り、 パイプライン のセクションから無料でダウンロードできます。
- 進行中のパイプラインを選択すると、パイプラインのステップが表示されます。
これにより、現在実行中のステップを監視できます。パイプラインが表示されるまでに数分かかる場合があります。 パイプラインの実行を開始するには、CI/CD で定義された手順codebuild-buildspec.yml正常に実行する必要があります。 これらのステップのステータスを確認するには、次を使用できます。 AWS コードビルド。 詳細については、を参照してください。 AWS コードビルド (AMS SSPS). - パイプラインが完了したら、プロジェクト ページに戻り、 モデルグループ タブをクリックして、モデル成果物に添付されたメタデータを調べます。
- すべてが良さそうな場合は、 ステータスの更新 タブを開き、モデルを手動で承認します。デフォルトは
ModelApprovalStatusに設定されていますPendingManualApproval。 モデルの精度が60%を超える場合、モデルはモデルレジストリに追加されますが、手動による承認が完了するまで展開されません。 - に移動します エンドポイント SageMaker コンソールのページで、ステージングエンドポイントが作成されていることを確認できます。数分後、エンドポイントが
InService状態。 - エンドポイントを本番環境にデプロイするには、
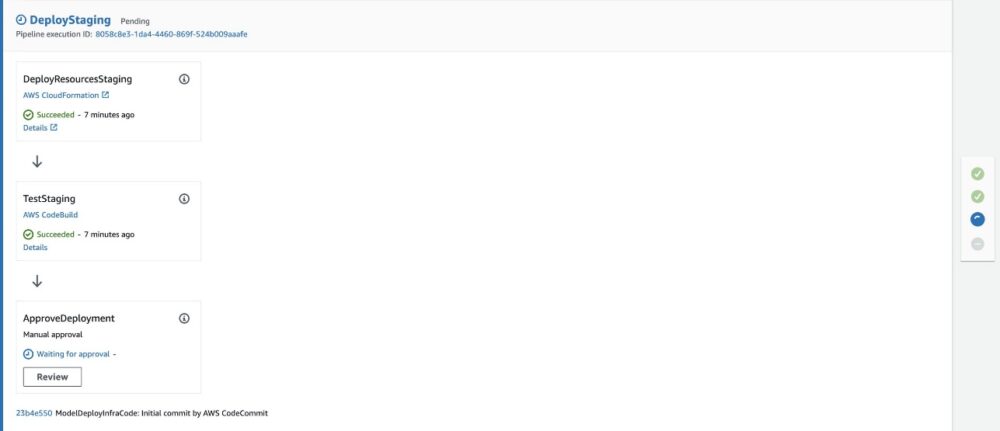
CodePipelineコンソールで、sagemaker--modeldeploy現在進行中のパイプライン。 - の終了時
DeployStaging段階で、展開を手動で承認する必要があります。
このステップの後、本番エンドポイントが SageMaker にデプロイされていることを確認できます エンドポイント ページ。 しばらくすると、エンドポイントが次のように表示されます。 InService.
クリーンアップ
SageMaker プロジェクトによって作成されたすべてのリソースを簡単にクリーンアップできます。
- Studio のナビゲーション ペインで、 SageMakerリソース.
- 選択する プロジェクト ドロップダウン メニューからプロジェクトを選択します。
- ソフトウェア設定ページで、下図のように メニュー、選択 削除 関連するすべてのリソースを削除します。
結果と次のステップ
Pipelines を使用して、HAM10000 データセットの組み込みモデルを使用して皮膚病変を分類するためのエンドツーエンドの MLOps フレームワークを作成することに成功しました。 リポジトリで提供されているパラメーターについて、テスト セットで次の結果が得られました。
| メトリック | 精度 | リコール | F1スコア |
| 値 | 0.643 | 0.8 | 0.713 |
ハイパーパラメータの微調整、データ拡張のための変換の追加、Synthetic Minority Oversampling Technique (SMOTE) や Generative Adversarial Networks (GAN) などの他の方法を使用して、モデルのパフォーマンスをさらに向上させることができます。 さらに、組み込みの SageMaker Docker イメージを使用するか、独自のコンテナを SageMaker で動作するように適合させることにより、トレーニングに独自のモデルまたはアルゴリズムを使用できます。 詳細については、次を参照してください。 SageMakerでのDockerコンテナの使用.
パイプラインに機能を追加することもできます。 モニタリングを含める場合は、 モデルの構築、トレーニング、デプロイ、モニタリング用の MLOps テンプレート SageMaker プロジェクトを作成するときのテンプレート。 結果のアーキテクチャには、追加の監視手順があります。 または、既存のサードパーティ Git リポジトリがある場合は、 Jenkins を使用したサードパーティの Git リポジトリを使用したモデルの構築、トレーニング、デプロイ用の MLOps テンプレート プロジェクトを作成し、モデル構築リポジトリとモデル展開リポジトリの両方に情報を提供します。 これにより、既存のコードを利用できるようになり、SageMaker と Git の統合にかかる時間や労力を節約できます。 ただし、このオプションでは、 AWS コードスター 接続が必要です。
まとめ
この投稿では、Studio と自動化されたパイプラインを使用してエンドツーエンドの ML ワークフローを作成する方法を示しました。 ワークフローには、データセットの取得、ML モデルにアクセス可能な場所への保存、前処理用のコンテナー イメージの構成、ボイラープレート コードの変更が含まれます。 次に、パイプラインをトリガーする方法、パイプラインがたどるステップ、およびそれらがどのように機能するかを示しました。 また、モデルのパフォーマンスを監視し、モデルをエンドポイントにデプロイする方法についても説明しました。
これらのタスクのほとんどは、包括的な ML IDE として機能し、そのようなモデルの開発と展開を加速する Studio 内で実行されました。
このソリューションは、肌の分類タスクに限定されません。 SageMaker 組み込みアルゴリズムまたは事前トレーニング済みモデルのいずれかを使用して、任意の分類または回帰タスクに拡張できます。
著者について
 マリエム・クティリ AWS Professional Services Globals の AI/ML コンサルタントであり、Health Care and Life Science (HCLS) チームの一員です。 彼女は、さまざまな問題に対する ML ソリューションの構築に情熱を傾けており、常に新しい機会や取り組みに積極的に取り組んでいます。 彼女はドイツのミュンヘンに住んでおり、旅行や世界の他の地域の発見に熱心です。
マリエム・クティリ AWS Professional Services Globals の AI/ML コンサルタントであり、Health Care and Life Science (HCLS) チームの一員です。 彼女は、さまざまな問題に対する ML ソリューションの構築に情熱を傾けており、常に新しい機会や取り組みに積極的に取り組んでいます。 彼女はドイツのミュンヘンに住んでおり、旅行や世界の他の地域の発見に熱心です。
 ヤシン・ザーフォーリ AWS のプロフェッショナル サービスの AI/ML コンサルタントです。 彼は、グローバル企業の顧客がクラウドで AI/ML ソリューションを構築および展開して、ビジネス上の課題を克服できるよう支援しています。 余暇には、スポーツをしたり観戦したり、世界中を旅行したりしています。
ヤシン・ザーフォーリ AWS のプロフェッショナル サービスの AI/ML コンサルタントです。 彼は、グローバル企業の顧客がクラウドで AI/ML ソリューションを構築および展開して、ビジネス上の課題を克服できるよう支援しています。 余暇には、スポーツをしたり観戦したり、世界中を旅行したりしています。
 フォティノス・キリアキデス AWS のプロフェッショナル サービスの AI/ML エンジニアです。 彼は、テクノロジーを使用して顧客に価値を提供し、ビジネスの成果を達成することに情熱を注いでいます。 ロンドンを拠点に、余暇にはランニングや探検を楽しんでいます。
フォティノス・キリアキデス AWS のプロフェッショナル サービスの AI/ML エンジニアです。 彼は、テクノロジーを使用して顧客に価値を提供し、ビジネスの成果を達成することに情熱を注いでいます。 ロンドンを拠点に、余暇にはランニングや探検を楽しんでいます。
 アンナ・ザパイシチコワ AI/ML の ProServe コンサルタントであり、Amazon Healthcare TFC のメンバーでした。 彼女はテクノロジーと、テクノロジーがヘルスケアに与える影響に情熱を注いでいます。 彼女のバックグラウンドは、保険、自動車、ヘルスケアなどのさまざまな分野における顧客の問題に対する MLOps と AI を活用したソリューションの構築です。
アンナ・ザパイシチコワ AI/ML の ProServe コンサルタントであり、Amazon Healthcare TFC のメンバーでした。 彼女はテクノロジーと、テクノロジーがヘルスケアに与える影響に情熱を注いでいます。 彼女のバックグラウンドは、保険、自動車、ヘルスケアなどのさまざまな分野における顧客の問題に対する MLOps と AI を活用したソリューションの構築です。