단백질은 효소 활동, 분자 수송 및 세포 지원과 같은 많은 생물학적 과정을 주도합니다. 단백질의 XNUMX차원 구조는 단백질의 기능과 다른 생체 분자와 상호 작용하는 방법에 대한 통찰력을 제공합니다. X선 결정학 및 NMR 분광법과 같은 단백질 구조를 결정하기 위한 실험적 방법은 비용이 많이 들고 시간이 많이 걸립니다.
반면에 최근에 개발된 계산 방법은 아미노산 서열에서 단백질의 구조를 빠르고 정확하게 예측할 수 있습니다. 이러한 방법은 많은 약물의 표적인 막 단백질과 같이 실험적으로 연구하기 어려운 단백질에 중요합니다. 이에 대한 잘 알려진 예는 다음과 같습니다. 알파 폴드, 정확한 예측으로 유명한 딥 러닝 기반 알고리즘.
에스엠폴드 아미노산 서열에서 단백질 구조를 예측하기 위해 개발된 또 다른 매우 정확한 딥 러닝 기반 방법입니다. ESMFold는 대형 단백질 언어 모델(pLM)을 백본으로 사용하고 종단 간 작동합니다. AlphaFold2와 달리 조회 또는 다중 시퀀스 정렬 (MSA) 단계이며 예측을 생성하기 위해 외부 데이터베이스에 의존하지도 않습니다. 대신 개발팀은 UniRef의 수백만 개의 단백질 서열에 대해 모델을 훈련했습니다. 훈련하는 동안 모델은 시퀀스에서 아미노산 간의 진화적 상호 작용을 우아하게 나타내는 주의 패턴을 개발했습니다. MSA 대신 pLM을 사용하면 다른 최첨단 모델보다 최대 60배 빠른 예측 시간이 가능합니다.
이 게시물에서는 Hugging Face의 사전 훈련된 ESMFold 모델을 사용합니다. 아마존 세이지 메이커 의 중쇄 구조를 예측하기 위해 트라 스투 주맙은 단일 클론 항체 처음 개발 한 제넨텍 치료를 위해 HER2 양성 유방암. 이 단백질의 구조를 신속하게 예측하는 것은 연구자들이 서열 변형의 효과를 시험하고자 할 때 유용할 수 있습니다. 이것은 잠재적으로 환자 생존율을 향상시키거나 부작용을 줄일 수 있습니다.
이 게시물은 다음에서 예제 Jupyter 노트북 및 관련 스크립트를 제공합니다. GitHub 저장소.
사전 조건
다음에서 이 예제를 실행하는 것이 좋습니다. 아마존 세이지 메이커 스튜디오 수첩 ml.r1.13.xlarge 인스턴스 유형에서 PyTorch 3.9 Python 5 CPU 최적화 이미지 실행.
trastuzumab의 실험 구조 시각화
시작하려면 biopython 라이브러리 및 도우미 스크립트에서 trastuzumab 구조를 다운로드합니다. RCSB 단백질 데이터 뱅크:
다음으로 우리는 py3Dmol 대화형 3D 시각화로 구조를 시각화하는 라이브러리:
다음 그림은 PDB(Protein Data Bank)의 3D 단백질 구조 1N8Z를 나타냅니다. 이 이미지에서 trastuzumab 경쇄는 주황색으로 표시되고 중쇄는 파란색(가변 영역은 밝은 파란색), HER2 항원은 녹색으로 표시됩니다.
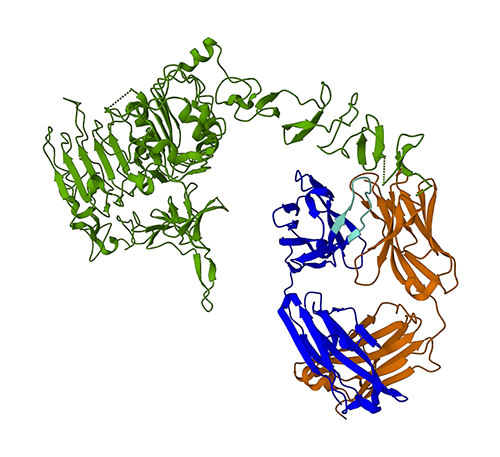
먼저 ESMFold를 사용하여 아미노산 서열에서 중쇄(체인 B)의 구조를 예측합니다. 그런 다음 예측을 위에 표시된 실험적으로 결정된 구조와 비교할 것입니다.
ESMFold를 사용하여 시퀀스에서 trastuzumab 중쇄 구조 예측
ESMFold 모델을 사용하여 중쇄의 구조를 예측하고 실험 결과와 비교해 봅시다. 시작하려면 다음과 같은 몇 가지 중요한 라이브러리와 함께 제공되는 Studio의 미리 빌드된 노트북 환경을 사용합니다. 파이 토치, 사전 설치됨. 가속화된 인스턴스 유형을 사용하여 노트북 분석의 성능을 향상시킬 수 있지만 대신 비가속 인스턴스를 사용하고 CPU에서 ESMFold 예측을 실행합니다.
먼저 사전 훈련된 ESMFold 모델과 토크나이저를 로드합니다. 허깅 페이스 허브:
다음으로 모델을 장치(이 경우 CPU)에 복사하고 일부 모델 매개변수를 설정합니다.
분석을 위해 단백질 서열을 준비하려면 이를 토큰화해야 합니다. 이는 아미노산 기호(EVQLV…)를 ESMFold 모델이 이해할 수 있는 숫자 형식(6,19,5,10,19,…)으로 변환합니다.
다음으로 토큰화된 입력을 모드에 복사하고 예측하고 결과를 파일에 저장합니다.
이 작업은 r3와 같은 비가속 인스턴스 유형에서 약 5분이 소요됩니다.
ESMFold 예측의 정확성을 실험 구조와 비교하여 확인할 수 있습니다. 우리는 이것을 사용하여 미국 정렬 University of Michigan의 Zhang Lab에서 개발한 도구:
| PDB체인1 | PDB체인2 | TM 점수 |
| 데이터/예측.pdb:A | 데이터/experimental.pdb:B | 0.802 |
XNUMXD덴탈의 템플릿 모델링 점수 (TM-점수)는 단백질 구조의 유사성을 평가하기 위한 메트릭입니다. 1.0의 점수는 완벽한 일치를 나타냅니다. 0.7 이상의 점수는 단백질이 동일한 백본 구조를 공유함을 나타냅니다. 0.9 이상의 점수는 단백질이 기능적으로 교환 가능 다운스트림 사용을 위해. TM-Score 0.802를 달성한 경우 ESMFold 예측은 구조 스코어링 또는 리간드 결합 실험과 같은 애플리케이션에 적합할 수 있지만 다음과 같은 사용 사례에는 적합하지 않을 수 있습니다. 분자 교체 매우 높은 정확도를 요구합니다.
정렬된 구조를 시각화하여 이 결과를 확인할 수 있습니다. 두 구조는 높지만 완벽하지는 않은 중첩 정도를 보여줍니다. 단백질 구조 예측은 빠르게 발전하는 분야이며 많은 연구팀이 더욱 정확한 알고리즘을 개발하고 있습니다!
ESMFold를 SageMaker 추론 엔드포인트로 배포
노트북에서 모델 추론을 실행하는 것은 실험에 적합하지만 모델을 애플리케이션과 통합해야 하는 경우에는 어떻게 해야 할까요? 아니면 MLOps 파이프라인? 이 경우 더 나은 옵션은 모델을 추론 엔드포인트로 배포하는 것입니다. 다음 예에서는 ESMFold를 가속화된 인스턴스에 SageMaker 실시간 추론 엔드포인트로 배포합니다. SageMaker 실시간 엔드포인트는 기계 학습(ML) 모델을 배포하고 호스팅하는 확장 가능하고 비용 효율적이며 안전한 방법을 제공합니다. 자동 조정을 사용하면 엔드포인트를 실행하는 인스턴스 수를 조정하여 애플리케이션의 요구 사항을 충족하고 비용을 최적화하고 고가용성을 보장할 수 있습니다.
미리 만들어진 Hugging Face용 SageMaker 컨테이너 일반적인 작업을 위한 딥 러닝 모델을 쉽게 배포할 수 있습니다. 그러나 단백질 구조 예측과 같은 새로운 사용 사례의 경우 사용자 지정을 정의해야 합니다. inference.py 모델을 로드하고, 예측을 실행하고, 출력 형식을 지정하는 스크립트입니다. 이 스크립트에는 노트북에서 사용한 것과 동일한 코드가 많이 포함되어 있습니다. 우리는 또한 requirements.txt 사용할 엔드포인트에 대한 일부 Python 종속성을 정의하는 파일입니다. 당신은 우리가 만든 파일을 볼 수 있습니다 GitHub 저장소.
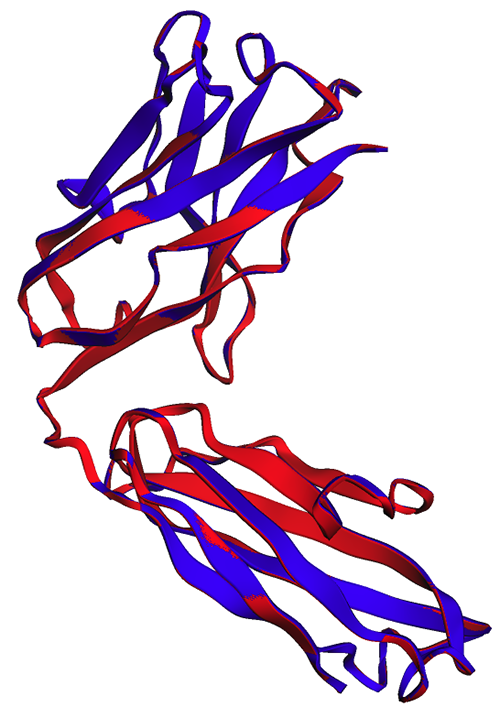
다음 그림에서 trastuzumab 중쇄의 실험적(파란색) 및 예측된(빨간색) 구조는 매우 유사하지만 동일하지는 않습니다.

필요한 파일을 생성한 후 code 디렉터리에서 SageMaker를 사용하여 모델을 배포합니다. HuggingFaceModel 수업. 이것은 사전 구축된 컨테이너를 사용하여 Hugging Face 모델을 SageMaker에 배포하는 프로세스를 단순화합니다. 가용성에 따라 끝점을 만드는 데 10분 이상 걸릴 수 있습니다. ml.g4dn 우리 지역의 인스턴스 유형.
엔드포인트 배포가 완료되면 단백질 시퀀스를 다시 제출하고 예측의 처음 몇 행을 표시할 수 있습니다.
엔드포인트를 가속화된 인스턴스에 배포했기 때문에 예측에는 몇 초밖에 걸리지 않습니다. 결과의 각 행은 단일 원자에 해당하며 아미노산 ID, XNUMX개의 공간 좌표 및 pLDDT 점수 해당 위치에서의 예측 신뢰도를 나타냅니다.
| PDB_그룹 | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | 장바구니_X | 장바구니_Y | CARTN_Z | 점유율 | PLDDT | ATOM_ID |
| ATOM | 1 | N | 글루 | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | 글루 | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | 글루 | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | 글루 | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | 글루 | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | 글루 | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | 글루 | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | 글루 | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | 글루 | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
이전과 동일한 방법을 사용하면 노트북과 엔드포인트 예측이 동일함을 알 수 있습니다.
| PDB체인1 | PDB체인2 | TM 점수 |
| 데이터/endpoint_prediction.pdb:A | 데이터/예측.pdb:A | 1.0 |
다음 그림에서 볼 수 있듯이 노트북 내(빨간색)와 엔드포인트(파란색)에서 생성된 ESMFold 예측은 완벽한 정렬을 보여줍니다.
정리
추가 요금이 부과되지 않도록 추론 엔드포인트와 테스트 데이터를 삭제합니다.
요약
전산 단백질 구조 예측은 단백질의 기능을 이해하는 데 중요한 도구입니다. 기본 연구 외에도 AlphaFold 및 ESMFold와 같은 알고리즘은 의학 및 생명 공학 분야에서 많은 응용 분야를 가지고 있습니다. 이러한 모델에서 생성된 구조적 통찰력은 생체 분자가 상호 작용하는 방식을 더 잘 이해하는 데 도움이 됩니다. 이것은 환자를 위한 더 나은 진단 도구와 치료법으로 이어질 수 있습니다.
이 게시물에서는 Hugging Face Hub의 ESMFold 단백질 언어 모델을 SageMaker를 사용하여 확장 가능한 추론 엔드포인트로 배포하는 방법을 보여줍니다. SageMaker에서 Hugging Face 모델 배포에 대한 자세한 내용은 다음을 참조하십시오. Amazon SageMaker에서 포옹 얼굴 사용. 또한 더 많은 단백질 과학 사례를 찾을 수 있습니다. AWS의 놀라운 단백질 분석 GitHub 저장소. 보고 싶은 다른 예가 있으면 의견을 남겨주세요!
저자에 관하여
 브라이언 로열 Amazon Web Services 글로벌 의료 및 생명 과학 팀의 수석 AI/ML 솔루션 설계자입니다. 그는 생명 공학 및 기계 학습 분야에서 17년 이상의 경력을 보유하고 있으며 고객이 게놈 및 단백질체 문제를 해결하도록 돕는 데 열정적입니다. 여가 시간에는 친구 및 가족과 함께 요리하고 식사하는 것을 즐깁니다.
브라이언 로열 Amazon Web Services 글로벌 의료 및 생명 과학 팀의 수석 AI/ML 솔루션 설계자입니다. 그는 생명 공학 및 기계 학습 분야에서 17년 이상의 경력을 보유하고 있으며 고객이 게놈 및 단백질체 문제를 해결하도록 돕는 데 열정적입니다. 여가 시간에는 친구 및 가족과 함께 요리하고 식사하는 것을 즐깁니다.
 샤 미카 아리 야 완사 Amazon Web Services 글로벌 의료 및 생명 과학 팀의 AI/ML 전문 솔루션 아키텍트입니다. 그는 기술 지침을 제공하고 고객이 AWS에서 안전한 클라우드 솔루션을 혁신하고 구축하도록 지원함으로써 AI 및 ML 채택을 가속화하기 위해 고객과 열정적으로 협력합니다. 일 외에는 스키와 오프로드를 좋아합니다.
샤 미카 아리 야 완사 Amazon Web Services 글로벌 의료 및 생명 과학 팀의 AI/ML 전문 솔루션 아키텍트입니다. 그는 기술 지침을 제공하고 고객이 AWS에서 안전한 클라우드 솔루션을 혁신하고 구축하도록 지원함으로써 AI 및 ML 채택을 가속화하기 위해 고객과 열정적으로 협력합니다. 일 외에는 스키와 오프로드를 좋아합니다.
 치 얀준 AWS Machine Learning Solution Lab의 선임 응용 과학 관리자입니다. 그녀는 AWS 고객이 AI 및 클라우드 채택 속도를 높일 수 있도록 기계 학습을 혁신하고 적용합니다.
치 얀준 AWS Machine Learning Solution Lab의 선임 응용 과학 관리자입니다. 그녀는 AWS 고객이 AI 및 클라우드 채택 속도를 높일 수 있도록 기계 학습을 혁신하고 적용합니다.
- SEO 기반 콘텐츠 및 PR 배포. 오늘 증폭하십시오.
- PlatoAiStream. Web3 데이터 인텔리전스. 지식 증폭. 여기에서 액세스하십시오.
- 미래 만들기 w Adryenn Ashley. 여기에서 액세스하십시오.
- PREIPO®로 PRE-IPO 회사의 주식을 사고 팔 수 있습니다. 여기에서 액세스하십시오.
- 출처: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :있다
- :이다
- :아니
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- 소개
- 위의
- AC
- 가속
- 가속 된
- 정확한
- 정확히
- 달성
- 활동
- 또한
- 양자
- AI
- AI / ML
- 연산
- 알고리즘
- 정렬 된
- 조정
- 또한
- 이기는하지만
- 아마존
- 아마존 세이지 메이커
- Amazon Web Services
- an
- 분석
- 및
- 다른
- 어떤
- 어플리케이션
- 어플리케이션
- 적용된
- 적당한
- 있군요
- AS
- 평가
- At
- 원자
- 주의
- Automatic
- 유효성
- 피하기
- AWS
- AWS 기계 학습
- 등뼈
- 은행
- 기본
- BE
- 전에
- 시작하다
- 더 나은
- 사이에
- 제본
- 생명 공학
- 파란색
- 빌드
- 비자 면제 프로그램에 해당하는 국가의 시민권을 가지고 있지만
- by
- CAN
- 게자리
- 만화
- 케이스
- 가지 경우
- 유명한
- 체인
- 과제
- 요금
- 검사
- 수업
- 클라우드
- 클라우드 채택
- 암호
- 색
- 제공
- 본문
- 공통의
- 비교
- 비교
- 완전한
- 자신
- 컨테이너
- 대조
- 대응
- 비용 효율적인
- 비용
- 수
- 만들
- 만든
- 임계
- 고객
- 데이터
- 데이터베이스
- 날짜 시간
- 깊은
- 깊은 학습
- Deepmind
- 도
- 요구
- 의존
- 배포
- 배포
- 배치
- 전개
- 결정
- 결정된
- 개발
- 개발
- 개발
- 장치
- 어려운
- 디스플레이
- do
- 하지
- 하지 않습니다
- 다운로드
- 드라이브
- 약물
- ...동안
- 마다
- 쉽게
- 효과
- 효과
- 수
- end
- 종점
- 보장
- 환경
- 예
- 예
- 비싼
- 경험
- 실험
- 외부
- 매우
- 페이스메이크업
- 가족
- 빠른
- 를
- 적은
- 들
- 그림
- 입양 부모로서의 귀하의 적합성을 결정하기 위해 미국 이민국에
- 파일
- Find
- 끝
- 먼저,
- 수행원
- 럭셔리
- 체재
- 친구
- 에
- 기능
- 추가
- 생성
- 생성
- GitHub의
- 글로벌
- 초록색
- 지도
- 있다
- he
- 건강 관리
- 무거운
- 도움
- 도움이
- 높은
- 그의
- 주인
- 방법
- How To
- 그러나
- HTML
- HTTPS
- 허브
- 포옹 얼굴
- 같은
- 통합 인증
- if
- 영상
- import
- 중대한
- 개선
- 개선하는
- in
- 포함
- 표시
- 표시
- 정보
- 받아 들여 쓰다
- 입력
- 통찰력
- 통찰력
- 예
- 를 받아야 하는 미국 여행자
- 통합
- 상호 작용하는
- 상호 작용
- 대화형
- 인터렉티브
- 으로
- IT
- 그
- 실험실
- 언어
- 넓은
- 리드
- 배우기
- 휴가
- 도서관
- 도서관
- 생활
- 생명과학
- 빛
- 처럼
- 아마도
- 하중
- 위치
- 조회
- 기계
- 기계 학습
- 확인
- 제작
- 매니저
- .
- 경기
- XNUMX월..
- 의학
- 소개
- 방법
- 방법
- 메트릭
- 미시간
- 수백만
- 회의록
- ML
- MLOps
- 모드
- 모델
- 모델
- 수정
- 분자의
- 배우기
- 많은
- name
- 필요한
- 필요
- NIH
- 수첩
- 소설
- 번호
- 사물
- 시대에 뒤 떨어진
- of
- on
- ONE
- 만
- 운영
- 최적화
- 선택권
- or
- 주황색
- OS
- 기타
- 우리의
- 출력
- 외부
- 매개 변수
- 열렬한
- 환자
- 환자
- 패턴
- 완전한
- 성능
- 관로
- 플라톤
- 플라톤 데이터 인텔리전스
- 플라토데이터
- 부디
- 게시하다
- 잠재적으로
- 예측
- 예측
- 예측
- 예측
- 예측
- Prepare
- 이전에
- 방법
- 프로세스
- 단백질
- 단백질
- 제공
- 제공
- 제공
- Python
- 파이 토치
- Qi
- 빨리
- 빠르게
- 빠르게 발전하는 분야
- 실시간
- 권하다
- 빨간색
- 지방
- 관련
- 의지하다
- 대표
- 대표
- 대표
- 필요
- 연구
- 연구원
- 결과
- 열
- 달리기
- 달리는
- 현자
- SageMaker 추론
- 같은
- 찜하기
- 확장성
- 스케일링
- 과학
- 과학
- 점수
- 점수
- 스크립트
- 초
- 안전해야합니다.
- 참조
- 연장자
- 순서
- 서비스
- 세트
- 몇몇의
- 공유
- 그녀
- 영상을
- 표시
- 표시
- 측면
- 비슷한
- 단순화
- 단일
- 해결책
- 솔루션
- 풀다
- 일부
- 공간의
- 전문가
- 분광학
- 속도
- 스타트
- 최첨단
- 단계
- 구조상의
- 구조
- 스튜디오
- 교육과정
- 이러한
- 적당한
- SUPPORT
- 살아남기
- 받아
- 소요
- 목표
- 작업
- 팀
- 팀
- 테크니컬
- test
- 보다
- 그
- XNUMXD덴탈의
- 그들의
- 그들
- 그때
- 그곳에.
- Bowman의
- 이
- 세
- 삼차원의
- 시간
- 시간이 많이 걸리는
- 시대
- 에
- 토큰 화하다
- 토큰 화 된
- 수단
- 검색을
- 토치
- 훈련 된
- 트레이닝
- 변압기
- 수송
- 치료
- 두
- 유형
- 유형
- 이해
- 이해
- 대학
- 같지 않은
- us
- 사용
- 익숙한
- 사용
- 유효 기간
- 대단히
- 관측
- 심상
- W
- 원
- 방법..
- we
- 웹
- 웹 서비스
- 잘 알려진
- 뭐
- 위키 백과
- 의지
- 과
- 워드프레스(WordPress)
- 작업
- 일
- 겠지
- 엑스선
- 자신의
- 너의
- 제퍼 넷