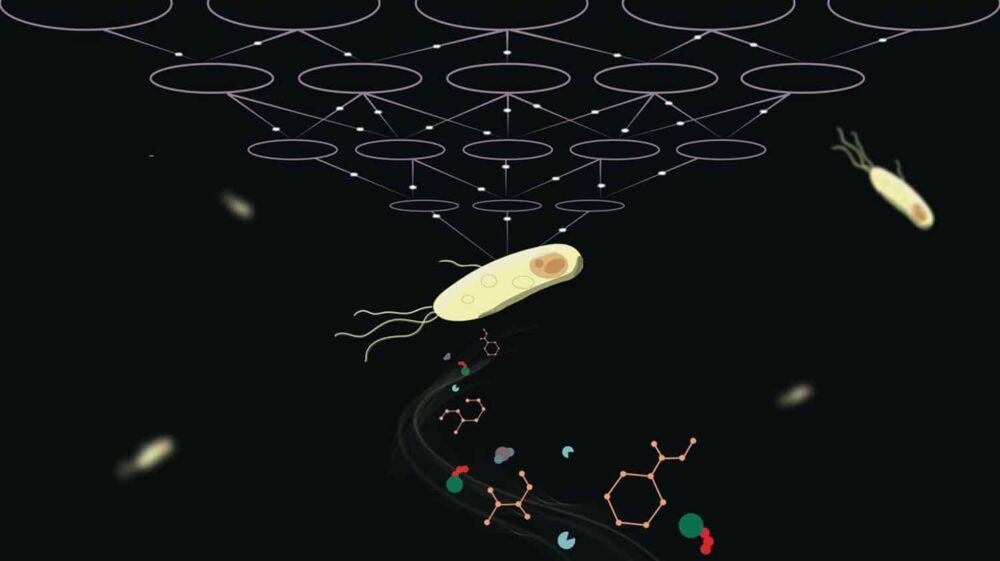
Alle levende ting krever metabolisme. Måten en organisme metaboliserer næringsstoffer er en kompleks prosess, og å simulere de kjemiske prosessene som holder livet i gang er en vanskelig utfordring.
Teoretisk kan prosedyren representeres av matematiske ligninger med parametere som er spesifikke for hver organisme. Men praktisk å bestemme disse parametrene er imidlertid en komplisert sak på grunn av mangelen på eksperimentelle data.
Forskere trenger generelt mye eksperimentell data og prosessorkraft for å finne disse parameterne. EPFL forskere foreslo et dypt læringsbasert beregningsrammeverk som reproduserer de dynamiske metabolske egenskapene observert i celler. Rammeverket kalt REKINDLE kan bane vei for mer effektiv og nøyaktig modellering av metabolske prosesser.
Ljubisa Miskovic fra EPFLs Laboratory of Computational Systems Biotechnology og co-PI i studien sa, "REKINDLE vil tillate forskningsmiljøet å redusere beregningsinnsatsen for å generere kinetiske modeller med flere størrelsesordener. Det vil også bidra til å postulere nye hypoteser ved å integrere biokjemiske data i disse modellene, belyse eksperimentelle observasjoner og styre nye terapeutiske oppdagelser og bioteknologiske design."
Subham Choudhury, den første forfatteren av studien, sa: "Det overordnede målet med metabolsk modellering er å beskrive cellulær metabolsk oppførsel i en slik grad at forståelse og forutsigelse av effektene av variasjoner i cellulære tilstander og miljøforhold pålitelig kan testes for et bredt spekter av studier innen helse, bioteknologi og systemer og syntetisk biologi. Vi håper at REKINDLE legger til rette for å bygge metabolske modeller for det bredere samfunnet.»
Teknikken har direkte bioteknologiske anvendelser fordi kinetiske modeller er avgjørende for en rekke undersøkelser, inkludert de om bioproduksjon, medikamentmålretting, interaksjoner mellom mikrober og bioremediering.
Choudhury sa, «REKINDLE bruker standard, mye brukte Python-biblioteker som gjør det tilgjengelig og enkelt å bruke. Vårt hovedmål med denne studien er å bane vei for å gjøre denne typen modelleringsinnsats åpen kildekode og tilgjengelig, slik at alle i det syntetiske og systembiologiske miljøet kan bruke dem til sitt eget forskningsmål, uansett hva de måtte være.»
Tidsreferanse:
- Choudhury, S., Moret, M., Salvy, P. et al. Rekonstruere kinetiske modeller for dynamiske studier av metabolisme ved bruk av generative kontradiktoriske nettverk. Nat Mach Intell 4, 710–719 (2022). GJØR JEG: 10.1038 / s42256-022-00519-y