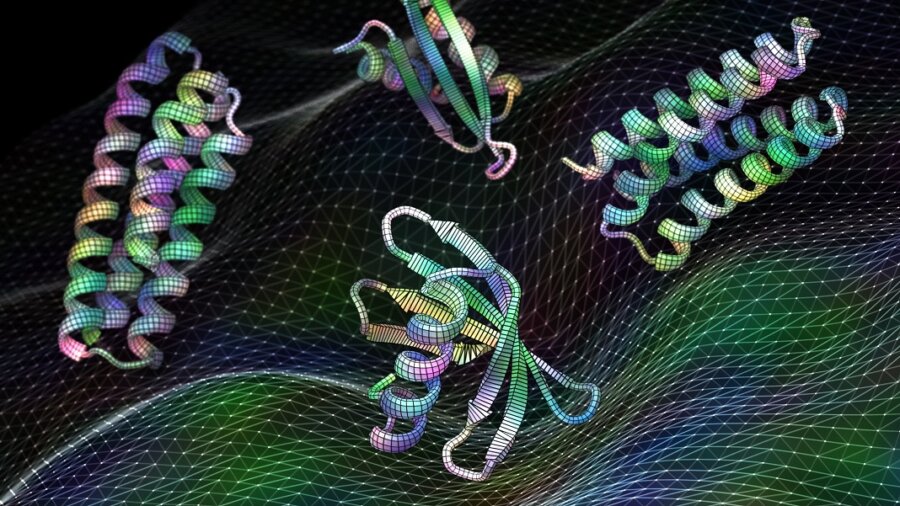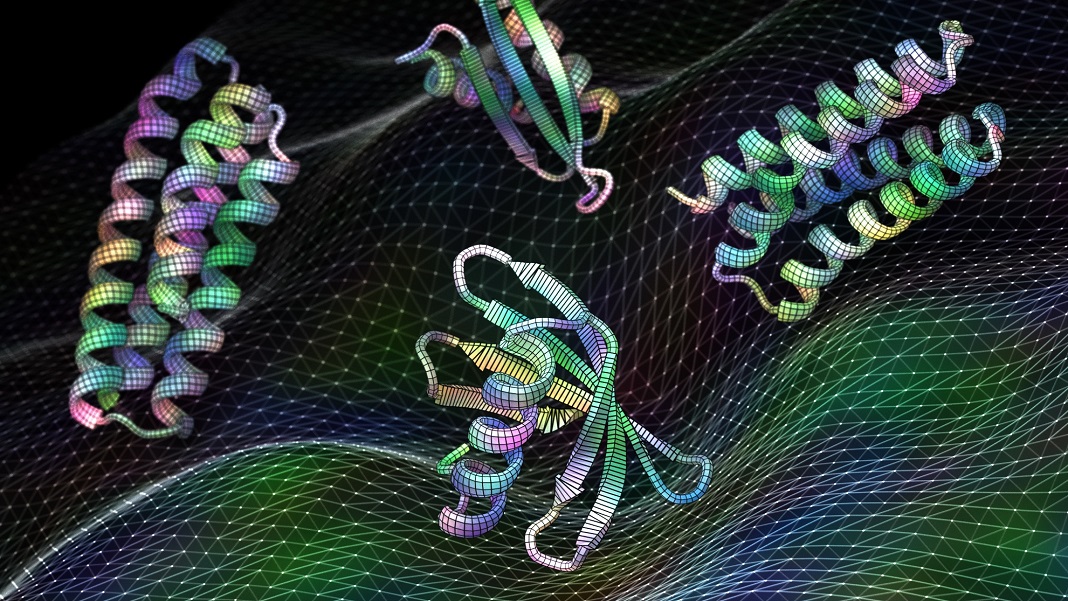
Projetar uma proteína é um pouco como fazer um armário. O primeiro passo é construir a espinha dorsal que mantém a proteína unida. Mas então vem a parte difícil: descobrir onde instalar as dobradiças no andaime – ou seja, encontrar os melhores “pontos quentes” – para colocar em portas, prateleiras e outros acessórios que, em última análise, tornam o gabinete totalmente funcional.
De certa forma, as proteínas também têm hotspots embutidos em suas estruturas. Fiel ao seu nome, “locais funcionais”, esses cantos e recantos intrigantes formam docas intrincadas para outras proteínas ou drogas se agarrarem. Os locais são centrais para realizar a maioria de nossos processos biológicos básicos. Eles também são uma enorme mina de ouro para projetar novos tratamentos e medicamentos.
O problema? Sites funcionais são difíceis de mapear. Os cientistas tradicionalmente tinham que mutar áreas suspeitas de uma proteína uma a uma – trocando um aminoácido por outro – para identificar pontos de ligação precisos. Como um detetive examinando centenas de suspeitos, dos quais pode haver muitos, é extremamente tedioso.
A novo estudo in Ciência derrubou todo o gamebook. Liderada pelo Dr. David Baker da Universidade de Washington, uma equipe aproveitou a “imaginação” de uma IA para criar uma infinidade de sites funcionais a partir do zero. É a “criatividade” de uma mente de máquina no seu melhor – um algoritmo de aprendizado profundo que prevê a área geral do local funcional de uma proteína, mas depois esculpe ainda mais a estrutura.
Como uma verificação da realidade, a equipe usou o novo software para gerar medicamentos que combatem o câncer e projetar vacinas contra vírus comuns, embora às vezes mortais. Em um caso, a mente digital surgiu com uma solução que, quando testada em células isoladas, era uma combinação perfeita para um anticorpo existente contra um vírus comum. Em outras palavras, o algoritmo “imaginou” um hotspot de uma proteína viral, tornando-a vulnerável como alvo para projetar novos tratamentos.
O algoritmo é a primeira incursão do aprendizado profundo na construção de proteínas em torno de suas funções, abrindo uma porta para tratamentos que antes eram inimagináveis. Mas o software não se limita a hotspots de proteínas naturais. “As proteínas que encontramos na natureza são moléculas incríveis, mas as proteínas projetadas podem fazer muito mais”, disse Baker em um comunicado à imprensa. O algoritmo está “fazendo coisas que nenhum de nós pensou que seria capaz”.
O ponto de proteína
A equipe de Baker conhece bem a previsão de proteínas com mentes artificiais. Alguns anos atrás, eles abalaram o campo da biologia estrutural ao lançar o Rosetta, um software que pode prever a estrutura 3D de uma proteína com base apenas em sua sequência de aminoácidos. Eles mapearam ainda mais complexos de proteínas e projetaram “chaves de fenda” de proteínas do zero para separar interações indesejáveis de proteínas. No final do ano passado, eles lançaram um rede de aprendizado profundo apelidado de trRosetta, um “arquiteto” de IA que generaliza como sequências de aminoácidos se organizam em estruturas intrincadas em nanoescala.
Vamos voltar.
É fácil imaginar as proteínas como a asa de frango carnuda e vigorosa que estou mordendo enquanto digito esta frase. Mas no nível molecular, eles são muito mais elegantes. Imagine vários blocos de Lego – aminoácidos – mantidos juntos por uma corda. Agora gire-o ao redor, torcendo a corrente até que alguns blocos se encaixem. Isso forma uma estrutura delicada que muitas vezes se assemelha a uma hélice ou lençóis amarrotados. Em algumas proteínas, esses blocos de construção se reúnem em complexos – por exemplo, criando um canal que atravessa a membrana protetora de uma célula como uma rodovia interestadual patrulhada.
As proteínas alimentam todos os processos biológicos, muitas vezes por meio de uma cascata de interações com outras proteínas ou drogas, que – dependendo do parceiro – podem desencadear consequências completamente diferentes: uma célula deve viver ou morrer? Atacar um invasor em potencial ou desistir? Em outras palavras, as proteínas são os blocos de construção da vida, e analisar sua estrutura é como podemos invadir a vida.
Aqui está a coisa: nem todas as partes de uma proteína são criadas iguais. Se uma proteína é um corpo humano, os locais funcionais são suas “mãos” – onde ela se agarra a outra proteína ou droga, provoca reações enzimáticas ou combate patógenos invasores. Incorporados diretamente na estrutura da proteína, esses locais são difíceis de identificar e ainda mais difíceis de recriar.
O novo estudo abordou o problema com uma versão do Rosetta: com algum conhecimento prévio, é possível um computador sonhar com uma cadeia de aminoácidos que se dobra naturalmente em um local funcional?
O sonhador e o realista
O problema pode parecer exótico, mas há um exemplo anterior — em um campo diferente. Usando uma rede neural, a OpenAI criou uma ampla variedade de imagens apenas com legendas de texto. Um spinoff do gerador de texto Rockstar AI GPT-3, o algoritmo DALL·E gerou imagens fantásticas, mas de aparência realista, com base em prompts de texto simples, detectando padrões de seu treinamento. “Pega os recessos mais profundos e sombrios da sua imaginação e os transforma em algo que é assustadoramente pertinente.” dito Dr. Hany Farid na UC Berkeley após o lançamento inicial da ferramenta.
A construção de um sítio funcional de proteína é semelhante. Aqui, os aminoácidos são as letras e o sítio funcional da proteína é a imagem. “A ideia é a mesma: as redes neurais podem ser treinadas para ver padrões nos dados. Uma vez treinado, você pode avisá-lo e ver se ele pode gerar uma solução elegante”, disse o Dr. Joseph Watson, principal autor do novo trabalho. Exceto que, em vez de escrever um romance, o algoritmo poderia ajudar a reescrever a vida.
A equipe começou com uma criação anterior, trRosetta. É uma rede neural originalmente projetada para inventar novas proteínas com base em sequências de aminoácidos enquanto é capaz de prever sua estrutura – algumas tão estranhas às naturais que a equipe apelidou o funcionamento interno do aprendizado profundo de “alucinação”. O algoritmo parecia perfeito: ele podia prever a sequência de aminoácidos de uma proteína e sua estrutura.
O soluço? Não funcionou. Em contraste, o GO de previsão da estrutura da proteína, RosaTTAFold, atuou como um campeão. O poder do algoritmo vem de seu design: modelar cada aminoácido em nanoescala, fornecendo coordenadas para cada átomo. Como fixar um site geográfico usando o Google Maps, isso fornece um nível de verdade para uma estrutura que uma IA pode explorar ainda mais – uma espécie de “alucinação restrita”.
Tradução? O RoseTTAFold pode prever uma estrutura funcional – específica para o problema em questão – e apresentar um esboço como o projeto final.
Então veio outro truque inteligente, apelidado de “inpainting”. Aqui, a equipe escondeu partes da sequência ou estrutura da proteína. O software teve que aprender a decifrar informações do que é essencialmente uma interceptação de rádio barulhenta, onde você só pode ouvir as primeiras palavras, mas tentar entender seu significado preenchendo os espaços em branco. O RoseTTAFold abordou o “problema de recuperação de informações ausentes” com entusiasmo, completando automaticamente sequências e estruturas de aminoácidos para construir uma determinada região funcional com alta fidelidade.
O RoseTTAFold pode resolver os problemas de construir sequências de aminoácidos e gerar uma espinha dorsal para o site ao mesmo tempo. É como colocar palavras no papel: o escritor garante que cada letra esteja no lugar certo, enquanto verifica se a gramática e o significado fazem sentido.
Questionando a Natureza da Realidade
Colocando sua nova criação à prova, a equipe gerou vários projetos de medicamentos e vacinas que poderiam combater vírus e câncer ou ajudar com problemas de saúde com baixo teor de ferro.
Para o autor principal Dr. Jue Wang, o algoritmo tornou-se inesperadamente pertinente. Enquanto trabalhava no projeto, seu filho de dois anos foi hospitalizado na unidade de emergência devido a uma infecção pulmonar pelo VSR (Vírus Sincicial Respiratório) – um vírus que normalmente apresenta sintomas semelhantes aos do resfriado, mas pode ser mortal em jovens e crianças. idoso.
Na época, Wang estava usando o algoritmo para projetar novos tratamentos, que incluíam locais potenciais no RSV para testar vacinas e medicamentos contra. É uma estrutura relativamente bem mapeada. O software alucinava projetos que recapitularam dois locais para as vacinas potencialmente se ligarem. Testes usando proteínas alucinadas, reconstruídas em bactérias, rapidamente se agarraram aos anticorpos existentes – um sinal de que eles são funcionais e que a abordagem de aprendizado profundo funciona.
O incidente “me fez perceber que mesmo os problemas de 'teste' em que estávamos trabalhando eram realmente bastante significativos”, disse Wang.
Em vários testes adicionais, a equipe projetou locais funcionais para uma enzima, proteínas de ligação a proteínas e proteínas que se agarram a íons metálicos – basicamente, como você absorve ferro e outros metais importantes.
Embora poderoso, há espaço para crescimento. O método abre as portas para desmistificar proteínas naturais, mas também potencialmente projetando novas para a biologia sintética. “Estas são novas abordagens muito poderosas, mas ainda há muito espaço para melhorias”, disse Baker.
Ao todo, é outra vitória para o aprendizado profundo e uma vitrine fascinante de como a IA e a biologia podem sinergizar. “O aprendizado profundo transformou a previsão da estrutura da proteína nos últimos dois anos, agora estamos no meio de uma transformação semelhante do design da proteína”, disse Baker.
Crédito da imagem: Ian C. Haydon/Instituto UW para Design de Proteínas. Novos softwares de inteligência artificial treinados em estruturas de proteínas podem gerar proteínas funcionais, incluindo essas vacinas candidatas para o vírus respiratório RSV, em segundos.