- Juli 7, 2014
- Vasilis Vryniotis
- . 1 kommentar
I de tidigare artiklarna diskuterade vi i detalj Dirichlet processblandningsmodeller och hur de kan användas i klusteranalys. I den här artikeln kommer vi att presentera en Java-implementering av två olika DPMM-modeller: Dirichlet Multivariate Normal Mixture Model som kan användas för att klustera gaussiska data och Dirichlet-Multinomial Mixture Model som används för att gruppera dokument. Java-koden är öppen källkod under GPL v3-licens och kan laddas ner gratis från Github.
Uppdatering: Datumbox Machine Learning Framework är nu öppen källkod och gratis att ladda ner. Kolla in paketet com.datumbox.framework.machinelearning.clustering för att se implementeringen av Dirichlet Process Mixture Models i Java.
Dirichlet Process Mixture Model implementering i Java
Koden implementerar Dirichlet Process Mixture Model med Gibbs Sampler och använder Apache Commons Math 3.3 som ett matrisbibliotek. Den är licensierad under GPLv3 så använd den gärna, modifiera den och distribuera den fritt och du kan ladda ner Java-implementeringen från Github. Observera att du kan hitta alla teoretiska delar av klustringsmetoden i de föregående 5 artiklarna och detaljerade Javadoc-kommentarer för implementering i källkoden.
Nedan listar vi en beskrivning på hög nivå av koden:
1. DPMM-klass
DPMM är en abstrakt klass och fungerar som en bas för de olika modellerna, implementerar Kinesisk restaurangprocess och innehåller Kollapsad Gibbs Sampler. Den har den offentliga metoden cluster() som tar emot datamängden som en lista över poäng och är ansvarig för att utföra klusteranalysen. Andra användbara metoder för klassen är getPointAssignments() som används för att hämta klustertilldelningarna efter att klustringen är klar och getClusterList() som används för att få listan över identifierade kluster. DPMM innehåller det statiska kapslade abstrakta klassen Cluster; den innehåller flera abstrakta metoder rörande hanteringen av poängen och skattningen av den bakre pdf som används för uppskattningen av klustertilldelningarna.
2. Gaussisk DPMM-klass
GaussianDPMM är implementeringen av Dirichlet Multivariate Normal Mixture Model och utökar DPMM-klassen. Den innehåller alla metoder som krävs för att uppskatta sannolikheterna under det Gaussiska antagandet. Dessutom innehåller den den statiska kapslade klassen Cluster som implementerar alla abstrakta metoder i klassen DPMM.Cluster.
3. MultinomialDPMM-klass
MultinomialDPMM implementerar Dirichlet-Multinomial Mixture Model och utökar DPMM-klassen. På samma sätt som GaussianDPMM-klassen innehåller den alla metoder som krävs för att uppskatta sannolikheterna under Multinomial-Dirichlet-antagandet och innehåller den statiska kapslade klassen Cluster som implementerar de abstrakta metoderna för DPMM.Cluster.
4. SRS-klass
SRS-klassen används för att utföra enkel slumpmässig sampling från en frekvenstabell. Den används av Gibbs Sampler för att uppskatta de nya klustertilldelningarna i varje steg i den iterativa processen.
5. Poängklass
Point-klassen fungerar som en tuppel som lagrar postens data tillsammans med dess id.
6. Apache Commons Math Lib
Apache Commons Math 3.3 lib används för Matrix-multiplikationer och det är det enda beroendet av vår implementering.
7. DPMMExempelklass
Den här klassen innehåller exempel på hur man använder Java-implementeringen.
Använder Java-implementeringen
Användaren av koden kan konfigurera alla parametrar för blandningsmodellerna, inklusive modelltyperna och hyperparametrarna. I följande kodavsnitt kan vi se hur algoritmen initieras och exekveras:
List<Point> pointList = new ArrayList<>();
//add records in pointList
//Dirichlet Process parameter
Integer dimensionality = 2;
double alpha = 1.0;
//Hyper parameters of Base Function
int kappa0 = 0;
int nu0 = 1;
RealVector mu0 = new ArrayRealVector(new double[]{0.0, 0.0});
RealMatrix psi0 = new BlockRealMatrix(new double[][]{{1.0,0.0},{0.0,1.0}});
//Create a DPMM object
DPMM dpmm = new GaussianDPMM(dimensionality, alpha, kappa0, nu0, mu0, psi0);
int maxIterations = 100;
int performedIterations = dpmm.cluster(pointList, maxIterations);
//get a list with the point ids and their assignments
Map<Integer, Integer> zi = dpmm.getPointAssignments();
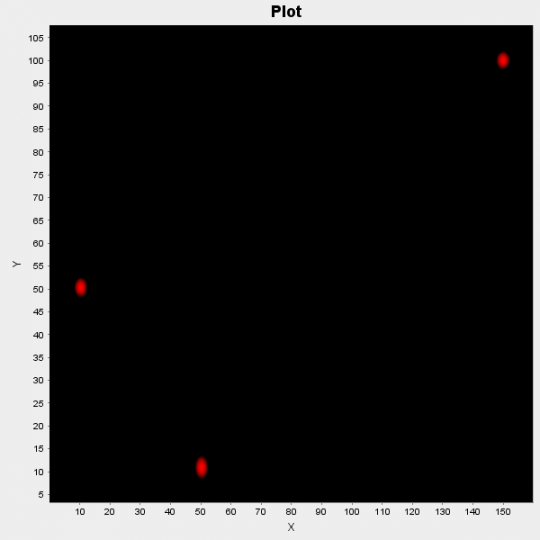
Nedan kan vi se resultatet av att köra algoritmen på en syntetisk datauppsättning som består av 300 datapunkter. Punkterna genererades ursprungligen av 3 olika fördelningar: N([10,50], I), N([50,10], I) och N([150,100], I).
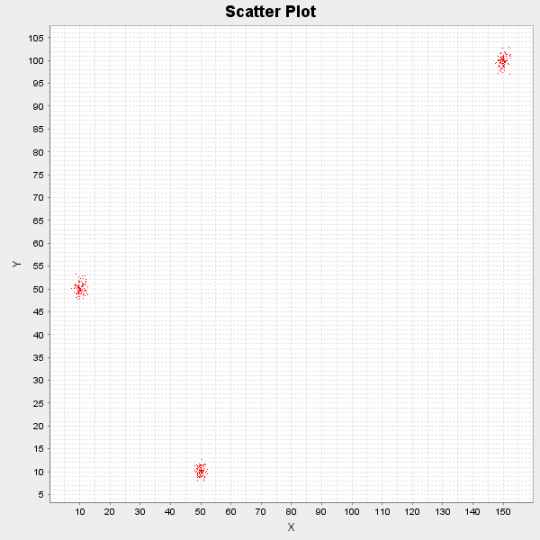
Figur 1: Scatter Plot för demodatauppsättning
Algoritmen efter att ha körts i 10 iterationer identifierade den följande 3 klustercentra: [10.17, 50.11], [49.99, 10.13] och [149.97, 99.81]. Slutligen, eftersom vi behandlar allt på ett Bayesianskt sätt, kan vi inte bara tillhandahålla enpunktsuppskattningar av klustercentra utan även deras sannolikhetsfördelning genom att använda formeln 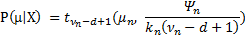 .
.
Figur 2: Spridningsdiagram över sannolikheter för klustercentrum
I figuren ovan plottar vi dessa sannolikheter; de röda områdena indikerar hög sannolikhet att vara mitten av ett kluster och svarta områden indikerar låg sannolikhet.
För att använda Java-implementeringen i verkliga applikationer måste du skriva extern kod som konverterar din ursprungliga datauppsättning till det format som krävs. Dessutom kan ytterligare kod vara nödvändig om du vill visualisera utdata som vi ser ovan. Notera slutligen att Apache Commons Math-biblioteket ingår i projektet och därför krävs ingen ytterligare konfiguration för att köra demos.
Om du använder implementeringen i ett intressant projekt skriv till oss så presenterar vi ditt projekt på vår blogg. Om du gillar artikeln, vänligen ta en stund och dela den på Twitter eller Facebook.
- AI
- ai konst
- ai art generator
- har robot
- artificiell intelligens
- artificiell intelligenscertifiering
- artificiell intelligens robot
- robotar med artificiell intelligens
- programvara för artificiell intelligens
- blockchain
- blockchain konferens ai
- coingenius
- konversationskonstnärlig intelligens
- kryptokonferens ai
- dalls
- Datumbox
- djupt lärande
- du har google
- maskininlärning
- Maskininlärning och statistik
- plato
- plato ai
- Platon Data Intelligence
- Platon spel
- PlatonData
- platogaming
- Programmering
- skala ai
- syntax
- zephyrnet