Amazon SageMaker管道 是为机器学习 (ML) 用例设计的持续集成和持续交付 (CI/CD) 服务。 您可以使用它来创建、自动化和管理端到端 ML 工作流。 它解决了编排 ML 流程的每个步骤的挑战,这需要时间、精力和资源。 为方便其使用,您可以根据需要自定义多个模板。
完全托管的图像和视频分析服务也加速了计算机视觉解决方案的采用。 AWS 提供预先训练和完全托管的 AWS AI 服务,称为 亚马逊重新认识 可以使用 API 调用集成到计算机视觉应用程序中,并且不需要 ML 经验。 您只需向 亚马逊识别 API 它可以根据预定义的标签识别所需的对象。 还可以提供特定于您的用例的自定义标签,并构建自定义的计算机视觉模型,而几乎不需要 ML 专业知识。
在这篇文章中,我们解决了一个特定的计算机视觉问题:皮肤病变分类,并通过自定义现有模板并将其定制为该任务来使用 Pipelines。 准确的皮肤病变分类有助于癌症疾病的早期诊断。 然而,这在医学领域是一项具有挑战性的任务,因为不同类型的皮肤病变之间存在高度相似性。 流水线让我们能够利用现有的各种模型和算法,以最少的努力和时间建立端到端的生产化流水线。
解决方案概述
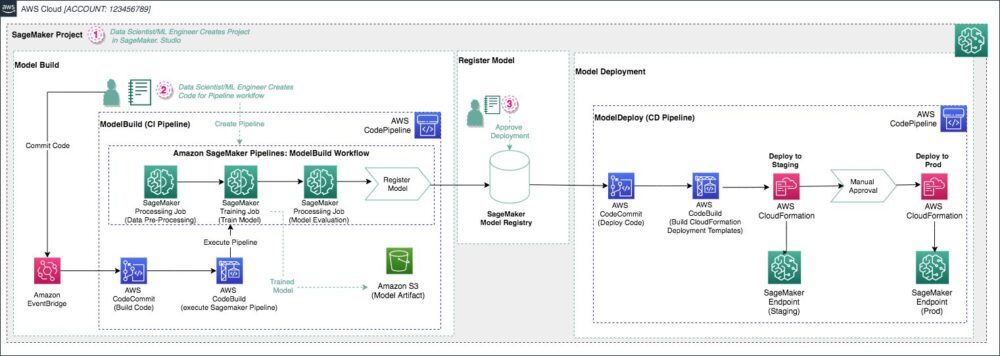
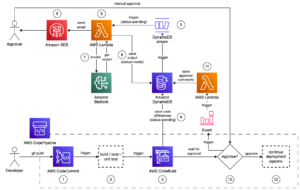
在这篇文章中,我们使用 Pipelines 构建了一个端到端的管道,以对常见色素性皮肤病变的皮肤镜图像进行分类。 我们使用 亚马逊SageMaker Studio 项目模板 用于构建,训练和部署模型的MLOps模板 和下面的代码 GitHub存储库. 生成的架构如下图所示。
对于这个管道,我们使用 HAM10000(“具有 10000 张训练图像的人机对抗机器”)数据集,其中包含 10,015 张皮肤镜图像。 手头的任务是计算机视觉领域的多类分类。 该数据集描述了色素病变领域中六个最重要的诊断类别:光化性角化病和上皮内癌或鲍文氏病(akiec), 基底细胞癌 (bcc),良性角化样病变(日光性痣或脂溢性角化病和扁平苔藓样角化病, bkl), 皮肤纤维瘤 (df), 黑色素瘤 (mel), 黑素细胞痣 (nv)和血管病变(血管瘤、血管角化瘤、化脓性肉芽肿和出血, vasc).
对于模型输入的格式,我们使用 RecordIO 格式。 这是一种紧凑的格式,可以将图像数据存储在一起以供连续阅读,因此可以更快、更有效地进行训练。 此外,使用 HAM10000 数据集的挑战之一是类别不平衡。 下表说明了类分布。
| 增益级 | 阿基克 | BCC | 黑色 | df | 蜂蜜 | nv | 血管 |
| 图片数量 | 327 | 514 | 1099 | 115 | 1113 | 6705 | 142 |
| 合计 | 10015 | ||||||
为了解决这个问题,我们使用随机变换(例如裁剪、翻转、镜像和旋转)来扩充数据集,以使所有类别的图像数量大致相同。
此预处理步骤使用 MXNet 和 OpenCV,因此它使用预构建的 MXNet 容器映像。 其余的依赖项是使用 requirements.txt 文件。 如果要创建和使用自定义图像,请参阅 使用映像构建 CI/CD 管道创建 Amazon SageMaker 项目.
对于训练步骤,我们使用 SageMaker 内置 Scikit Docker 映像中可用的估计器进行图像分类,并设置参数如下:
有关容器映像的更多详细信息,请参阅 图像分类算法.
创建工作室项目
有关如何设置 Studio 的详细说明,请参阅 使用快速设置载入 Amazon SageMaker 域. 要创建您的项目,请完成以下步骤:
- 在 Studio 中,选择 项目 菜单上 SageMaker资源 菜单。
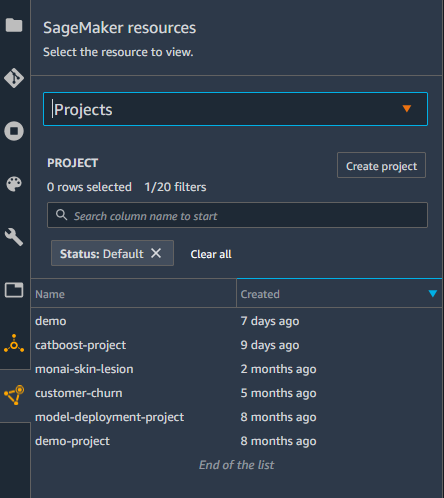
在项目页面上,您可以启动预配置的 SageMaker MLOps 模板。 - 用于模型构建,培训和部署的MLOps模板.
- 选择项目模板.
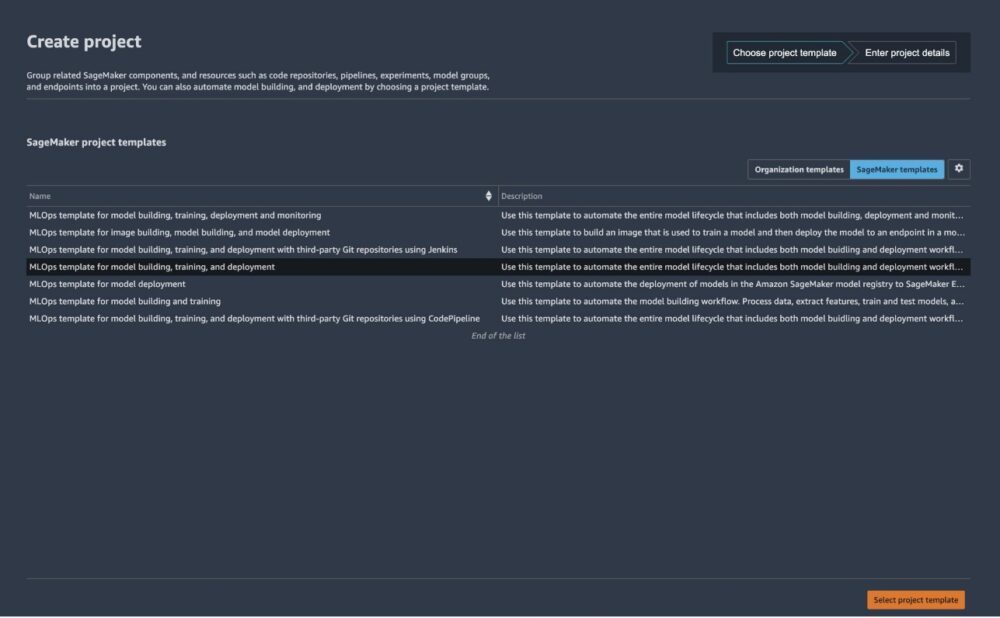
- 输入项目名称和简短描述。
- 创建项目.
创建该项目需要几分钟时间。
准备数据集
要准备数据集,请完成以下步骤:
- 在MyCAD中点击 软件更新 哈佛数据宇宙.
- 访问数据集,并查看许可证 Creative Commons Attribution-NonCommercial 4.0 International Public License。
- 如果您接受许可,请选择 原始格式 Zip 并下载 ZIP 文件。
- 创建 亚马逊简单存储服务 (Amazon S3) 存储桶并选择以
sagemaker(这允许 SageMaker 在没有任何额外权限的情况下访问存储桶)。 - 您可以启用访问日志记录和加密以获得最佳安全实践。
- 上传
dataverse_files.zip到桶。 - 保存 S3 存储桶路径以供以后使用。
- 记下您存储数据的存储桶的名称以及任何后续文件夹的名称,以供以后使用。
准备数据预处理
因为我们在预处理步骤中使用 MXNet 和 OpenCV,所以我们使用预构建的 MXNet Docker 映像并使用 requirements.txt 文件。 为此,您需要将其复制并粘贴到 pipelines/skin ,在 sagemaker--modelbuild 存储库。 此外,添加 MANIFEST.in 文件在同一级别 setup.py, 告诉 Python 包含 requirements.txt 文件。 有关更多信息 清单文件,请参阅 使用 MANIFEST.in 在源代码分发中包含文件. 这两个文件都可以在 GitHub存储库.
更改管道模板
要更新管道模板,请完成以下步骤:
- 在默认存储桶内创建一个文件夹。
- 确保 Studio 执行角色有权访问默认存储桶以及包含数据集的存储桶。
- 从项目列表中,选择您刚刚创建的项目。
- 点击 库 选项卡,选择要在本地克隆的超链接 AWS 代码提交 存储库到您的本地 Studio 实例。
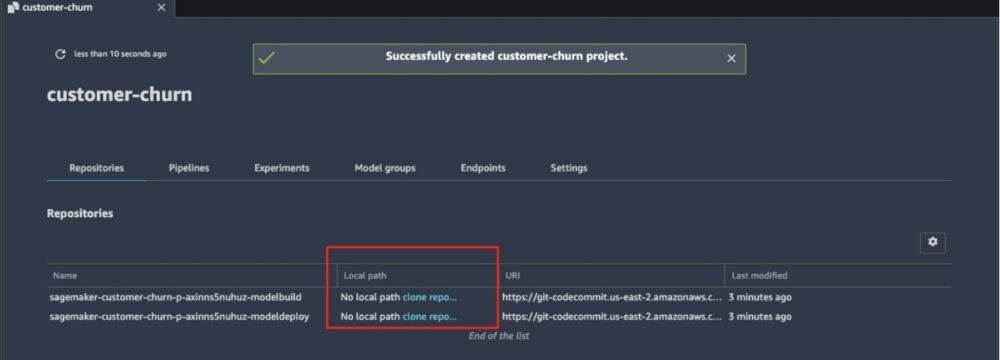
- 导航到
pipelines里面的目录sagemaker--modelbuild目录并重命名abalone目录skin. - 打开
codebuild-buildspec.yml文件中sagemaker--modelbuild目录并修改运行管道路径run-pipeline —module-name pipelines.abalone.pipeline(第 15 行)到以下内容: - 保存该文件。
- 替换文件
pipelines.py,preprocess.py和evaluate.py在管道目录中,其中包含来自 GitHub存储库.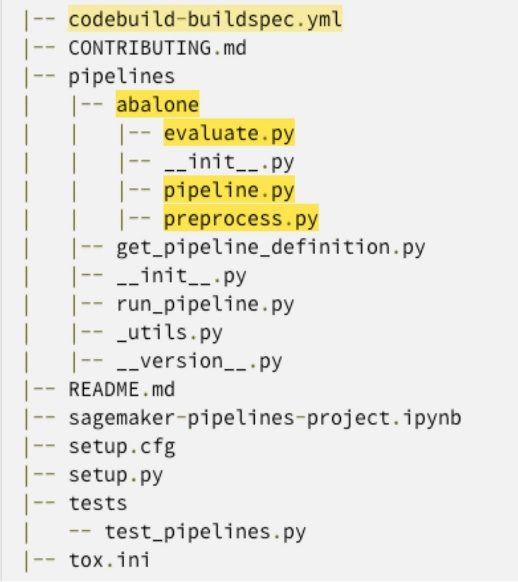
- 更新
preprocess.py文件(第 183-186 行)与 S3 位置(SKIN_CANCER_BUCKET) 和文件夹名称 (SKIN_CANCER_BUCKET_PATH) 您上传的位置dataverse_files.zip档案:skin_cancer_bucket=””skin_cancer_bucket_path=””skin_cancer_files=””skin_cancer_files_ext=””
在前面的示例中,数据集将存储在 s3://monai-bucket-skin-cancer/skin_cancer_bucket_prefix/dataverse_files.zip.
触发管道运行
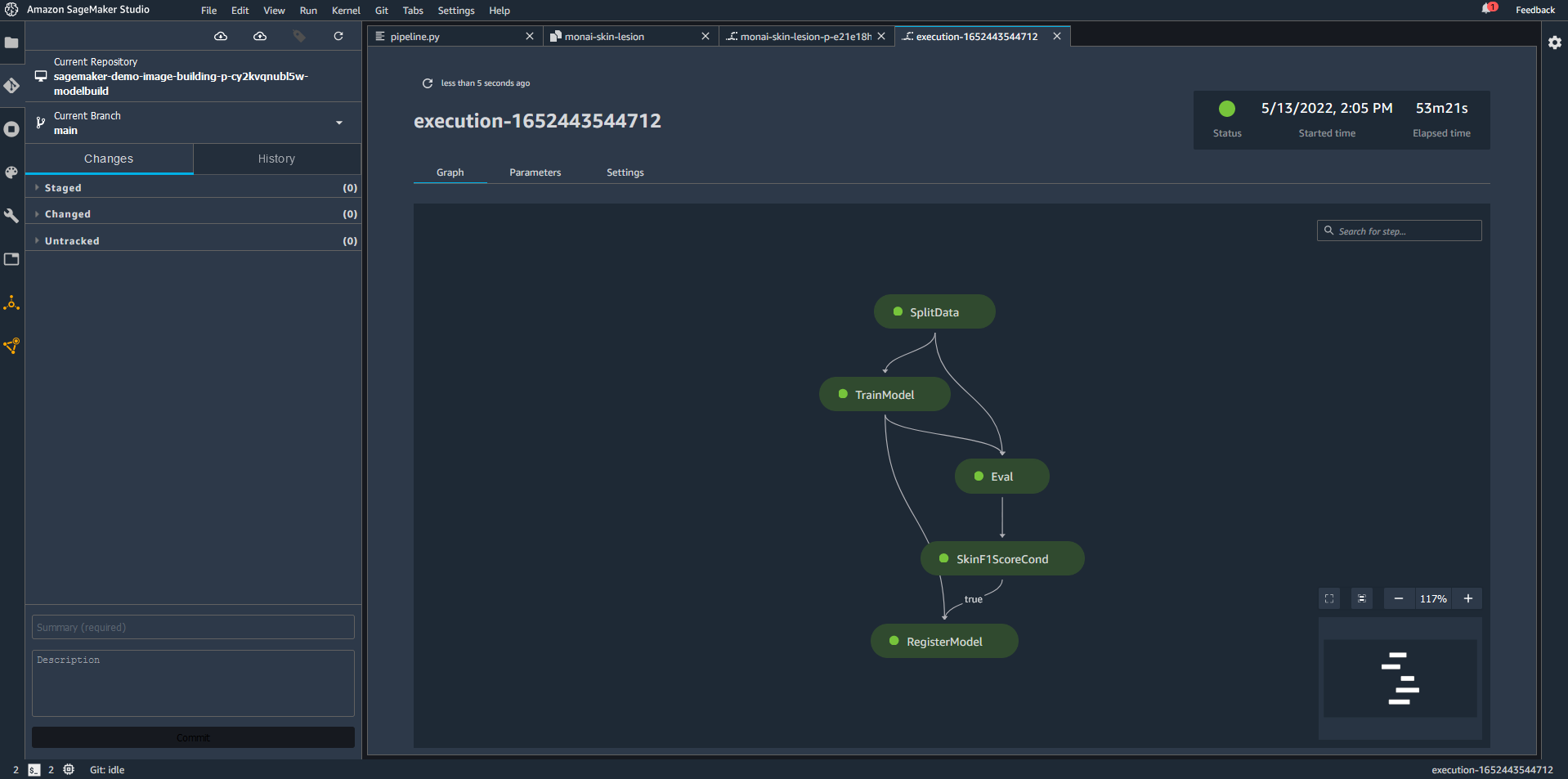
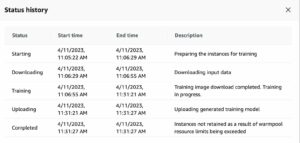
将已提交的更改推送到 CodeCommit 存储库(在 Studio 源代码控制选项卡上完成)会触发新的管道运行,因为 亚马逊EventBridge 提交的事件监视器。 我们可以通过选择 SageMaker 项目中的管道来监控运行。 以下屏幕截图显示了成功运行的管道示例。
- 要提交更改,请导航到左侧窗格中的 Git 部分。
- 暂存所有相关更改。 您无需跟踪
-checkpoint文件。 您可以将条目添加到.gitignore文件*checkpoint.*忽略它们。 - 通过提供摘要以及您的姓名和电子邮件地址来提交更改。
- 推动更改。
- 导航回项目并选择 管道 部分。
- 如果您选择正在进行的管道,则会显示管道的步骤。
这使您可以监视当前正在运行的步骤。管道可能需要几分钟才能出现。 为了使管道开始运行,CI/CD 中定义的步骤codebuild-buildspec.yml必须成功运行。 要检查这些步骤的状态,您可以使用 AWS 代码构建. 有关更多信息,请参阅 AWS CodeBuild(AMS SSPS). - 管道完成后,返回项目页面并选择 模型组 选项卡以检查附加到模型工件的元数据。
- 如果一切看起来都不错,请选择 更新状态 选项卡并手动批准模型。默认
ModelApprovalStatus被设置为PendingManualApproval。 如果我们的模型的准确性超过60%,则会将其添加到模型注册表中,但要等到手动批准完成后才能部署。 - 导航到 端点 SageMaker 控制台上的页面,您可以在其中看到正在创建的暂存端点。几分钟后,端点与
InService状态。 - 要将端点部署到生产中,在
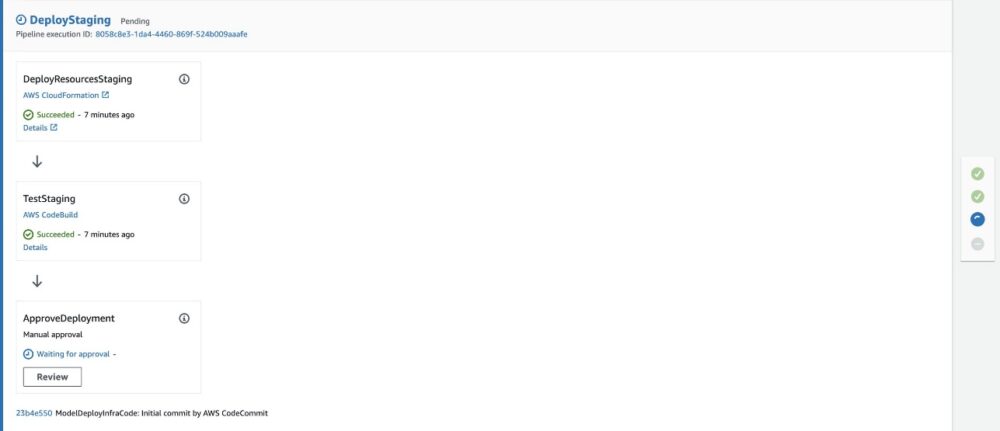
CodePipeline控制台,选择sagemaker--modeldeploy目前正在进行的管道。 - 在结束时
DeployStaging阶段,您需要手动批准部署。
完成此步骤后,您可以看到 SageMaker 上部署的生产端点 端点 页。 一段时间后,端点显示为 InService.
清理
您可以轻松清理 SageMaker 项目创建的所有资源。
- 在 Studio 的导航窗格中,选择 SageMaker资源.
- 项目 从下拉菜单中选择您的项目。
- 点击 行动 菜单中选择 删除 删除所有相关资源。
结果和后续步骤
我们使用 Pipelines 成功地使用 HAM10000 数据集上的内置模型创建了用于皮肤病变分类的端到端 MLOps 框架。 对于存储库中提供的参数,我们在测试集上得到了以下结果。
| 米制 | 平台精度 | 记得 | F1分数 |
| 值 | 0.643 | 0.8 | 0.713 |
您可以通过微调其超参数、为数据增强添加更多转换或使用其他方法(例如合成少数过采样技术 (SMOTE) 或生成对抗网络 (GAN))来进一步提高模型的性能。 此外,您可以使用自己的模型或算法进行训练,方法是使用内置的 SageMaker Docker 映像或调整自己的容器以在 SageMaker 上工作。 有关详细信息,请参阅 将 Docker 容器与 SageMaker 一起使用.
您还可以向管道添加其他功能。 如果要包括监控,可以选择 用于模型构建、训练、部署和监控的 MLOps 模板 创建 SageMaker 项目时的模板。 由此产生的架构有一个额外的监控步骤。 或者,如果您有一个现有的第三方 Git 存储库,您可以通过选择 MLOps 模板,用于使用 Jenkins 使用第三方 Git 存储库进行模型构建、训练和部署 项目并为模型构建和模型部署存储库提供信息。 这允许您利用任何现有代码并节省您在 SageMaker 和 Git 之间集成的任何时间或精力。 然而,对于这个选项,一个 AWS 代码之星 需要连接。
结论
在这篇文章中,我们展示了如何使用 Studio 和自动化流水线创建端到端的 ML 工作流。 工作流程包括获取数据集,将其存储在 ML 模型可访问的位置,配置容器图像以进行预处理,然后修改样板代码以适应此类图像。 然后我们展示了如何触发管道、管道遵循的步骤以及它们是如何工作的。 我们还讨论了如何监控模型性能并将模型部署到端点。
我们在 Studio 中执行了大部分任务,Studio 充当了一个包罗万象的 ML IDE,并加速了此类模型的开发和部署。
此解决方案不受皮肤分类任务的约束。 您可以使用任何 SageMaker 内置算法或预训练模型将其扩展到任何分类或回归任务。
关于作者
 玛丽姆·克西里 是 AWS Professional Services Globals 的 AI/ML 顾问,并且是医疗保健和生命科学 (HCLS) 团队的一员。 她热衷于为各种问题构建 ML 解决方案,并且总是渴望抓住新的机会和举措。 她住在德国慕尼黑,热衷于旅行和探索世界其他地方。
玛丽姆·克西里 是 AWS Professional Services Globals 的 AI/ML 顾问,并且是医疗保健和生命科学 (HCLS) 团队的一员。 她热衷于为各种问题构建 ML 解决方案,并且总是渴望抓住新的机会和举措。 她住在德国慕尼黑,热衷于旅行和探索世界其他地方。
 亚辛·扎福里 是 AWS 专业服务部门的 AI/ML 顾问。 他使全球企业客户能够在云中构建和部署 AI/ML 解决方案,以克服他们的业务挑战。 在业余时间,他喜欢打球、观看体育赛事和环游世界。
亚辛·扎福里 是 AWS 专业服务部门的 AI/ML 顾问。 他使全球企业客户能够在云中构建和部署 AI/ML 解决方案,以克服他们的业务挑战。 在业余时间,他喜欢打球、观看体育赛事和环游世界。
 福蒂诺斯·基里亚基德斯 是 AWS 专业服务部门的 AI/ML 工程师。 他热衷于使用该技术为客户提供价值并实现业务成果。 常驻伦敦,业余时间喜欢跑步和探索。
福蒂诺斯·基里亚基德斯 是 AWS 专业服务部门的 AI/ML 工程师。 他热衷于使用该技术为客户提供价值并实现业务成果。 常驻伦敦,业余时间喜欢跑步和探索。
 安娜·扎帕什奇科娃 曾是 AI/ML 领域的 ProServe 顾问,也是 Amazon Healthcare TFC 的成员。 她对技术及其对医疗保健的影响充满热情。 她的背景是为保险、汽车和医疗保健等多个领域的客户问题构建 MLOps 和人工智能驱动的解决方案。
安娜·扎帕什奇科娃 曾是 AI/ML 领域的 ProServe 顾问,也是 Amazon Healthcare TFC 的成员。 她对技术及其对医疗保健的影响充满热情。 她的背景是为保险、汽车和医疗保健等多个领域的客户问题构建 MLOps 和人工智能驱动的解决方案。