Introduktion
Opdagelsen under Human Genome Project i begyndelsen af 2000'erne, at vi mennesker kun har omkring 20,000 proteinkodende gener - omtrent lige så mange som den lille jordlevende nematodeorm og mindre end halvt så mange som risplanten - kom som et chok . Det slag mod vores stolthed blev dog mildnet af tanken om, at det menneskelige genom er rigt på regulatoriske forbindelser. Vores gener interagerer i et tæt netværk, hvor stykker af DNA og de molekyler, de koder for (RNA og proteiner), styrer "ekspressionen" af andre gener, hvilket påvirker, om de laver deres respektive RNA og proteiner. For at forstå det menneskelige genom var vi nødt til at forstå denne proces med genregulering.
Den opgave har dog vist sig at være meget sværere end at afkode genomets sekvens.
Oprindeligt var det mistanke om, at genregulering var et simpelt spørgsmål om, at et genprodukt fungerede som en tænd/sluk-knap for et andet gen på digital vis. I 1960'erne belyste de franske biologer François Jacob og Jacques Monod første gang en genregulerende proces i mekanistiske detaljer: In Escherichia coli bakterier, når et repressorprotein binder sig til et bestemt DNA-segment, blokerer det transskriptionen og translationen af en tilstødende række af gener, der koder for enzymer til at fordøje sukkerlaktosen. Dette reguleringskredsløb, som Monod og Jacob døbte sø operon, har en pæn, gennemsigtig logik.
Men genregulering i komplekse metazoer - dyr som mennesker med komplekse eukaryote celler - ser generelt ikke ud til at fungere på denne måde. I stedet involverer det en bande af molekyler, inklusive proteiner, RNA'er og stykker af DNA fra hele et kromosom, som på en eller anden måde samarbejder om at kontrollere ekspressionen af et gen.
Det er ikke kun, at denne reguleringsproces i eukaryoter har flere spillere, end det typisk ses i bakterier og andre simple, prokaryote celler; det ser ud til at være en kategorisk anderledes proces, og en mere uklar proces.
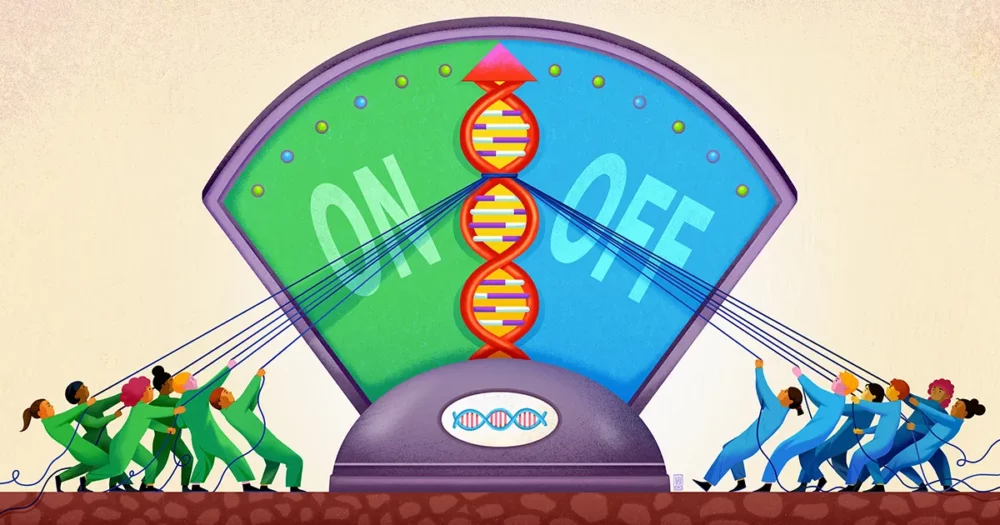
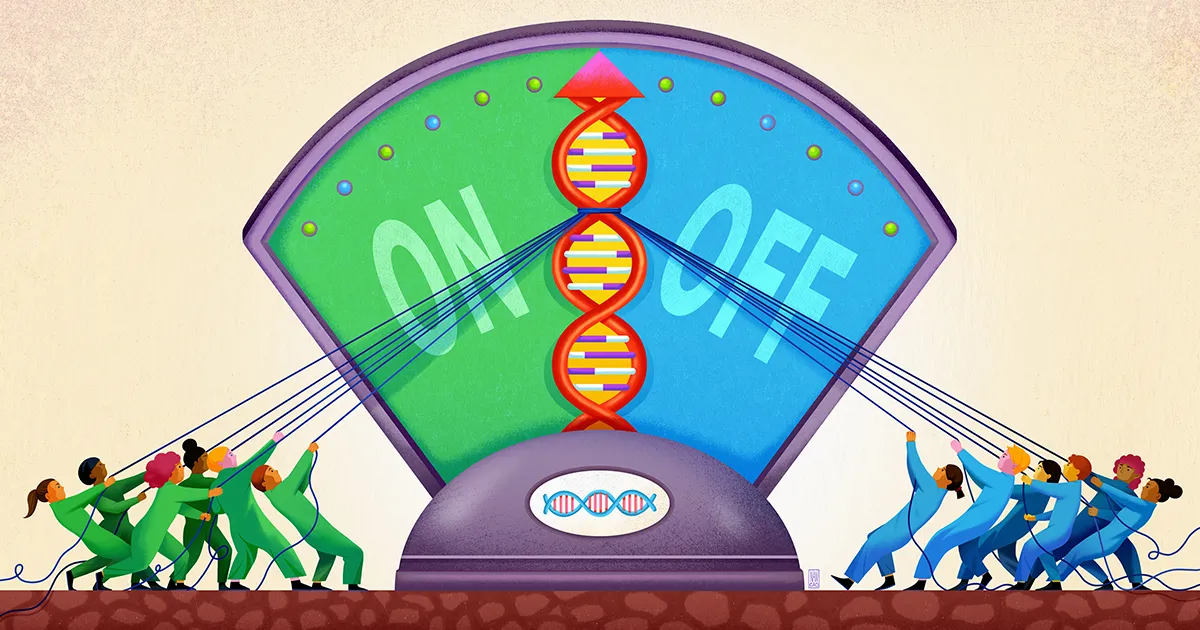
Et team på Stanford University, ledet af biofysikeren og bioingeniøren Polly Fordyce, ser nu ud til at have afsløret en komponent af denne uklare genreguleringsmetode. Deres arbejde, udgivet i september sidste år i Videnskab, tyder på, at DNA'et i nærheden af et gen fungerer som en slags lavvandet brønd til at fange forskellige regulatoriske molekyler og holde dem klar til handling, så de, når det er nødvendigt, kan tilføje deres stemme til beslutningen om, hvorvidt genet skal aktiveres.
Introduktion
Disse regulatoriske brønde er lavet af decideret underlige strækninger af DNA. De består af sekvenser, hvor et kort stykke DNA, fra et til seks basepar langt, gentages mange gange. Tit af kopier af disse "short tandem repeats" (STR'er) kan sættes sammen i disse sekvenser, som det samme lille "ord" skrevet igen og igen.
STR'er er rigelige i det menneskelige genom: De udgør omkring 5% af alt vores DNA. De blev engang anset for at være klassiske eksempler på "junk"-DNA, fordi en gentagen DNA-"tekst", der kun består af STR'er, ikke kan indeholde nær så meget meningsfuld information som f.eks. den uregelmæssige rækkefølge af bogstaver, der udgør en sætning i denne artikel.
Og alligevel er STR'er tydeligvis ikke ubetydelige: De er blevet forbundet med lidelser som Huntingtons sygdom, spinobulbar muskelatrofi, Crohns sygdom og nogle kræftformer. I løbet af de sidste par årtier er der akkumuleret beviser for, at de på en eller anden måde kan forbedre eller hæmme genregulering. Mysteriet var, hvordan de kunne være så kraftfulde med så lidt informationsindhold.
Komplekse kontroller til komplekse celler
For at forstå, hvordan STR'er passer ind i det store billede af genregulering, lad os tage et skridt tilbage. Gener er rutinemæssigt flankeret af stykker af DNA, der ikke koder for RNA eller protein, men har regulerende funktioner. Bakterielle gener har "promotor"-regioner, hvor polymeraseenzymer kan binde for at begynde transkriptionen af tilstødende DNA til RNA. De har også rutinemæssigt "operator"-regioner, hvor repressorproteiner kan binde sig til at blokere transkription, hvilket slår et gen fra, som i sø operon.
Hos mennesker og andre eukaryoter kan de regulerende sekvenser være mere talrige, forskellige - og forvirrende. Regioner kaldet forstærkere, for eksempel, påvirker sandsynligheden for, at et gen vil blive transskriberet. Enhancers er ofte målene for proteiner kaldet transkriptionsfaktorer, som kan binde for at booste eller hæmme genekspression. Mærkeligt nok er nogle forstærkere titusindvis af basepar væk fra de gener, de regulerer, og bringes kun tæt på dem gennem den fysiske omlejring af DNA-løkkerne i et pakket kromosom.
Eukaryotisk genregulering involverer typisk disse mange forskellige regulatoriske blokke af DNA, sammen med en eller flere transkriptionsfaktorer og andre molekyler, der alle samles omkring et gen som en komité, der er indkaldt til at beslutte, hvad det skal gøre. De samles i en løs, tæt klynge.
Ofte ser de molekylære deltagere heller ikke ud til at interagere gennem de meget selektive "lås og nøgle"-parringer, der er almindelige i molekylærbiologi. De er i stedet meget mindre kræsne, interagerer ret svagt og uselektivt, som om de vandrer rundt og indleder korte samtaler med hinanden.
Faktisk har det været noget af et mysterium, hvordan transkriptionsfaktorer binder til DNA i eukaryoter. Det har længe været antaget, at en del af en transkriptionsfaktor skal matche en bindende "motiv"-sekvens i DNA'et, som brikkerne i et puslespil. Men selv om nogle sådanne motiver er blevet identificeret, er deres tilstedeværelse ikke altid korreleret særlig godt med, hvor videnskabsmænd finder transkriptionsfaktorer, der klæber til DNA i celler. Nogle gange hænger transkriptionsfaktorer i områder uden motiver, mens nogle motiver, der virker, som om de skulle binde kraftigt til transskriptionsfaktorer, forbliver tomme.
"Traditionelt i genomik har målet været at klassificere genomiske steder på en [binær] måde som enten 'bundet' eller 'ubundet'" af transkriptionsfaktorer, sagde Fordyce. "Men billedet er meget mere nuanceret end som så." De enkelte medlemmer af disse genregulerende "komitéer" ser ikke ud til altid at være til stede til eller fraværende fra deres møder, men snarere har forskellige sandsynligheder for at være der eller ej.
Tendensen til genregulering hos eukaryoter til at stole på så mange forskellige svage interaktioner mellem store molekylære komplekser "er en af de ting, der gør det notorisk vanskeligt at få styr på teoretisk," sagde biofysikeren Thomas Kuhlman fra University of California, Riverside, som skrev en kommentar på Fordyce-laboratoriets papir for Videnskab. Det er et dybt puslespil, hvordan der ud af denne tilsyneladende kaotiske proces opstår præcise beslutninger om at tænde og slukke for gener.
Ud over den mystiske fuzzy logik i den beslutningsproces, er der også spørgsmålet om, hvordan alle udvalgsmedlemmer overhovedet finder vej til det rigtige rum - og så bliver der. Molekyler bevæger sig generelt rundt i cellen ved diffusion, stødt af alle de andre omgivende molekyler, såsom vand, og vandrer i tilfældige retninger. Vi kan forvente, at disse løse udvalg glider fra hinanden for hurtigt til at udføre deres reguleringsarbejde.
Det, mener Fordyce og hendes kolleger, er, hvor STR'erne kommer ind. STR'er er slående almindelige inden for forstærkersteder på DNA. I deres papir hævder forskerne, at STR'erne fungerer som klæbrige pletter, der fremkalder transkriptionsfaktorer og forhindrer dem i at forvilde sig.
Finjustering af klæbrigheden
Fordyces gruppe undersøgte systematisk, hvordan forskelle i STR-sekvens påvirker vedhæftningen af transkriptionsfaktorer til et bindingsmotiv. De så på to faktorer - en fra gær, en fra mennesker - der holder sig til et bestemt seks-baseret motiv. Forskerne målte både styrken (eller affiniteten) af den binding og den hastighed, hvormed transkriptionsfaktorerne sætter sig fast og ikke hænger fast (kinetik), når motivet er flankeret af en STR i stedet for en tilfældig sekvens. Til sammenligning så de på, hvor let faktorerne binder til STR alene og til en helt tilfældig DNA-sekvens.
"En af de største udfordringer på dette felt er at skille de utallige variabler, der påvirker [transkriptionsfaktor]-binding ved en specifik position af genomet," sagde David Suter, en molekylærbiolog ved Swiss Federal Institute of Technology Lausanne i Schweiz. DNA-form, nærhed til andre DNA-segmenter og fysisk spænding i DNA-molekylerne kan alle spille en rolle i transkriptionsfaktorbinding. Værdierne af disse parametre adskiller sig sandsynligvis på hver position i genomet, og måske også mellem celletyper og inden for en enkelt celle over tid på en given position. "Dette er et stort rum af ukendte variabler, som er meget svære at kvantificere," sagde Suter.
Introduktion
Det er derfor, velkontrollerede eksperimenter som dem fra Stanford-holdet er så nyttige, tilføjede Kuhlman. Normalt, når forskere skal måle svage interaktioner som disse, har de to valg: De kan foretage nogle få meget detaljerede, ekstremt præcise målinger og generalisere ud fra dem, eller de kan tage rigtig mange hurtige og beskidte målinger og bruge matematisk komplekse statistiske metoder til at udlede resultater. Men Fordyce og hendes kolleger, sagde Kuhlman, brugte en automatiseret, mikrofluidisk chip-baseret procedure til at tage præcise målinger under high-throughput eksperimenter "for at få det bedste fra begge verdener."
Stanford-holdet fandt ud af, at forskellige STR-sekvenser kan ændre bindingsaffiniteterne af transkriptionsfaktorer til DNA med så meget som en faktor på 70; de har nogle gange mere indflydelse på transkriptionsfaktorbinding end at ændre sekvensen af selve bindingsmotivet. Og effekterne var forskellige for de to forskellige transskriptionsfaktorer, de så på.
Så STR'er ser ud til at være i stand til at finjustere transskriptionsfaktorers evne til at docke på et DNA-sted og dermed regulere et gen. Men hvordan præcist?
Et venteværelse nær et gen
Forskerne regnede med, at den del af en transkriptionsfaktor, der binder DNA, kunne interagere svagt med en STR, med den nøjagtige styrke af denne affinitet afhængig af STR-sekvensen. Fordi en sådan binding er svag, vil den ikke have meget specificitet. Men hvis en transkriptionsfaktor er løst grebet og frigivet af en STR igen og igen, er den kumulative effekt at holde transkriptionsfaktoren i nærheden af genet, så det er mere sandsynligt, at den binder sikkert til motivregionen, hvis det er nødvendigt.
Fordyce og hendes kolleger forudsagde, at STR'er således fungerer som en "lobby" eller brønd, hvor transkriptionsfaktorer kan samle sig, dog forbigående, nær et regulatorisk bindingssted. "Den gentagne karakter af en STR forstærker den svage effekt af ethvert enkelt bindingssted, som det er lavet af," sagde Connor Horton, den første forfatter på undersøgelsen, som nu er ph.d.-studerende ved University of California, Berkeley.
Omvendt tilføjede han, at nogle STR'er også kan virke til at trække transkriptionsfaktorer væk fra regulatoriske sekvenser og opsuge transkriptionsfaktorer andre steder som en svamp. På denne måde kan de hæmme genekspression.
Arbejdet, sagde Suter, "viser overbevisende, at STR'er direkte påvirker binding af transkriptionsfaktorer in vitro." Hvad mere er, brugte Stanford-teamet en maskinlæringsalgoritme til at vise, at virkningerne set i deres in vitro-eksperimenter også ser ud til at forekomme i levende celler (det vil sige in vivo).
Men Robert Tjian, en biokemiker ved Berkeley og en efterforsker ved Howard Hughes Medical Institute, mener, at det kan være for tidligt at være sikker på, hvilken indflydelse en given STR-transkriptionsfaktorkombination har på genekspression i rigtige celler.
Tjian, Xavier Darzacq og deres kolleger i laboratoriet, de kører sammen i Berkeley, er enige om, at STR'er synes at tilbyde en måde at koncentrere transkriptionsfaktorer i nærheden af genregulerende steder. Men uden at vide, hvor tæt faktorerne skal være for at aktivere transskription, er det svært at forstå den funktionelle betydning af dette resultat. Tjian sagde, at han gerne ville se, om introduktion af en STR i en levende celle forudsigeligt påvirker ekspressionen af et målgen. På nuværende tidspunkt, sagde han, er han "ikke overbevist om, at STR'er nødvendigvis vil være et vigtigt aspekt af [regulatoriske] mekanismer in vivo."
En kombinatorisk grammatik
Et dvælende puslespil er, hvordan en sådan mekanisme pålideligt giver den type præcis genregulering, som celler har brug for, da både styrken og selektiviteten af transkriptionsfaktorbinding i STR-brøndene er svag. Fordyce mener, at en sådan specificitet af indflydelse kan komme fra mange kilder - ikke kun fra forskelle i STR-sekvenserne, men også fra samarbejdsinteraktioner mellem transkriptionsfaktorer og andre proteiner involveret i regulering.
I betragtning af alt det, sagde Horton, er det ikke klart, at det vil være ligetil at forudsige effekten af en given STR-transkriptionsfaktorkombination på ekspressionen af et gen. Logikken i processen er faktisk uklar. Og "grammatikken" af indflydelsen er sandsynligvis kombinatorisk, tilføjede Horton: Resultatet afhænger af forskellige kombinationer af transkriptionsfaktorer og andre molekyler.
Stanford-teamet mener, at måske 90% af transkriptionsfaktorerne er følsomme over for STR'er, men at der er mange flere typer af transkriptionsfaktorer i det menneskelige genom, end der er typer af STR'er. "At mutere en STR-sekvens kan påvirke bindingen af 20 forskellige transkriptionsfaktorer i den celletype, hvilket fører til et samlet fald i transkriptionen af det nærliggende gen uden at implicere nogen specifik transkriptionsfaktor," sagde Horton.
Så i virkeligheden er Stanford-holdet enige med Tjian i, at genregulering i levende celler ikke vil blive drevet af en enkelt enkel mekanisme. Tværtimod kan transkriptionsfaktorer, deres DNA-bindingssteder og andre regulatoriske molekyler samle sig til tætte samlinger, der udøver deres indflydelse kollektivt.
"Der er nu flere eksempler, der understøtter ideen om, at DNA-elementer kan fortrænge transkriptionsfaktorer til det punkt, hvor de danner kondensater med cofaktorer," sagde Richard Young, en cellebiolog ved Whitehead Institute of the Massachusetts Institute of Technology. Enhancers binder mange transkriptionsfaktorer for at producere den trængsel. STR'er kan være en ingrediens, der hjælper med at samle transkriptionsfaktorer til at klynge sig tæt på et gen, men de vil ikke være hele historien.
Hvorfor regulere gener på denne komplicerede måde i stedet for at stole på den slags stærke og specifikke interaktioner mellem regulatoriske proteiner og DNA-steder, der dominerer i prokaryoter? Det er muligt, at sådan uklarhed overhovedet har gjort store komplekse metazoer mulige.
For at være levedygtige arter skal organismer være i stand til at udvikle sig og tilpasse sig skiftende omstændigheder. Hvis vores celler stolede på et enormt, men tæt foreskrevet netværk af genregulerende interaktioner, ville det være svært at foretage ændringer i det uden at forstyrre hele genstanden, ligesom et schweizisk ur vil gribe fat, hvis vi fjerner (eller endda lidt forskyder) af dens utallige tandhjul. Hvis de regulatoriske molekylære interaktioner er løse og ret uspecifikke, er der dog et nyttigt slapt i systemet - ligesom et udvalg generelt kan træffe en god beslutning, selvom et af dets medlemmer er sygemeldt.
Fordyce bemærker, at i prokaryoter som bakterier kan det være relativt nemt for transkriptionsfaktorer at finde deres bindingssteder, fordi genomet, der skal søges, er mindre. Men det bliver sværere, efterhånden som genomet bliver større. I de store genomer af eukaryoter, "kan du ikke længere tolerere risikoen for, at du forbigående sidder fast på et 'forkert' bindingssted," sagde Fordyce, fordi det ville kompromittere evnen til at reagere hurtigt på skiftende miljøforhold.
Desuden er STR'er selv meget evolverbare. En forlængelse eller forkortelse af deres sekvens eller en ændring af størrelsen og dybden af "transkriptionsfaktorbrønden" kan let forekomme gennem uheld i DNA-replikation eller reparation eller gennem seksuel rekombination af kromosomerne. For Fordyce antyder det, at STR'er "derfor kan tjene som råmateriale til at udvikle nye regulatoriske elementer og finjustere eksisterende regulatoriske moduler til følsomme transskriptionsprogrammer," såsom dem, der styrer udviklingen af dyr og planter.
Kraften i svage interaktioner
Sådanne overvejelser får molekylærbiologer til at være meget mere opmærksomme på svage og relativt uselektive interaktioner i genomet. Mange af disse involverer proteiner, der i stedet for at have en fast og præcis struktur, er løse og floppy - "iboende uordnede", som biokemikere udtrykker det. Hvis proteiner kun virkede gennem stive strukturelle domæner, forklarede Young, ville det ikke kun begrænse, hvor godt reguleringssystemer kunne udvikle sig, men også den slags dynamiske regulering, der ses i livet. "Du vil ikke finde en levende organisme - eller endda en virus - der kun fungerer med stabile strukturelle elementer som dem i et schweizisk ur," sagde Young.
Måske snublede evolutionen bare over STR'er som en del af en så kompleks, men i sidste ende mere effektiv løsning på genregulering i eukaryoter. STR'er selv kan opstå på flere måder - for eksempel gennem fejl i DNA-replikation eller aktiviteten af DNA-segmenter kaldet transponerbare elementer, der laver kopier af sig selv i hele genomet.
"Det skete bare sådan, at de resulterende svage interaktioner mellem proteiner og de gentagne sekvenser var noget, der kunne ... give selektiv fordel til cellerne, hvor det forekom," sagde Kuhlman. Hans gæt er, at denne uklarhed sandsynligvis blev påtvunget eukaryoter, men at "de efterfølgende var i stand til at udnytte [det] til deres egen fordel." Bakterier og andre prokaryoter kan stole på veldefineret "digital" regulatorisk logik, fordi deres celler har tendens til kun at eksistere i nogle få enkle, forskellige tilstande, såsom at bevæge sig rundt og replikere.
Men de forskellige celletilstande for metazoer er "meget mere komplekse og nogle gange tæt på et kontinuum," sagde Suter, så de er bedre tjent med mere fuzzier "analog" regulering.
"De genregulerende systemer i bakterier og eukaryoter ser ud til at have divergeret ganske væsentligt," sagde Tjian enig. Mens Monod siges at have engang bemærket, at "hvad er sandt for E. coli er sandt for elefanten,” ser det ud til, at det ikke altid er tilfældet.
- SEO Powered Content & PR Distribution. Bliv forstærket i dag.
- PlatoData.Network Vertical Generative Ai. Styrk dig selv. Adgang her.
- PlatoAiStream. Web3 intelligens. Viden forstærket. Adgang her.
- PlatoESG. Kulstof, CleanTech, Energi, Miljø, Solenergi, Affaldshåndtering. Adgang her.
- PlatoHealth. Bioteknologiske og kliniske forsøgs intelligens. Adgang her.
- Kilde: https://www.quantamagazine.org/a-lobby-where-a-molecule-mob-tells-genes-what-to-do-20240214/
- :har
- :er
- :ikke
- :hvor
- ][s
- $OP
- 000
- 20
- 70
- a
- evne
- I stand
- Om
- fraværende
- rigelig
- Akkumuleret
- Lov
- handler
- Handling
- aktivere
- aktivitet
- handlinger
- tilpasse
- tilføje
- tilføjet
- tilstødende
- Fordel
- påvirke
- tilhørsforhold
- igen
- aftalt
- er enig
- algoritme
- Alle
- alene
- sammen
- også
- Skønt
- altid
- blandt
- forstærker
- an
- ,
- dyr
- En anden
- enhver
- fra hinanden
- ER
- argumentere
- opstå
- omkring
- artikel
- AS
- udseende
- antaget
- At
- opmærksomhed
- forfatter
- Automatiseret
- væk
- tilbage
- Bakterier
- bund
- BE
- fordi
- bliver
- været
- begynde
- være
- gavner det dig
- Berkeley
- BEDSTE
- Bedre
- mellem
- Big
- Store billede
- større
- Største
- binde
- binding
- biologi
- Bloker
- Blocks
- blæse
- boost
- både
- bragte
- men
- by
- california
- kaldet
- kom
- CAN
- celle
- Celler
- vis
- udfordringer
- Ændringer
- skiftende
- valg
- Kromosomer
- omstændigheder
- Classic
- Klassificere
- klar
- tydeligt
- Luk
- nøje
- Cluster
- samarbejde
- kolleger
- kollektivt
- kombination
- kombinationer
- Kom
- udvalg
- Fælles
- sammenligning
- komplekse
- kompliceret
- komponent
- kompromis
- betingelser
- Tilslutninger
- overvejelser
- indhold
- Continuum
- kontrol
- kontrol
- samtaler
- kooperativ
- kunne
- Par
- crowd
- årtier
- beslutte
- beslutning
- afgørelser
- Dekodning
- falde
- Afhængigt
- afhænger
- dybde
- detail
- detaljeret
- Udvikling
- afvige
- forskelle
- forskellige
- svært
- Broadcasting
- digital
- digital mode
- direkte
- opdagelse
- Sygdom
- distinkt
- forskelligartede
- dna
- do
- Dock
- Er ikke
- Domæner
- Dominere
- Dont
- drevet
- døbt
- i løbet af
- dynamisk
- Tidligt
- nemt
- let
- effekt
- Effektiv
- effekter
- enten
- elementer
- elefant
- andetsteds
- emerge
- forbedre
- miljømæssige
- fejl
- Endog
- Hver
- bevismateriale
- evolution
- udvikle sig
- udviklende
- præcist nok
- eksempel
- eksempler
- eksisterer
- eksisterende
- forvente
- eksperimenter
- forklarede
- Exploit
- udtryk
- ekstremt
- Faktisk
- faktor
- faktorer
- Mode
- Federal
- få
- felt
- regnede
- Finde
- Fornavn
- passer
- fast
- Til
- tvunget
- formular
- fundet
- Fransk
- fra
- funktionel
- fungerer
- funktioner
- Bande
- samle
- indsamling
- sammenkomster
- generelt
- genomforskning
- få
- får
- given
- mål
- gå
- godt
- styrende
- stor
- gruppe
- gætte
- Halvdelen
- håndtere
- skete
- Hård Ost
- hårdere
- Have
- have
- he
- hjælper
- hende
- stærkt
- hans
- hold
- Hvordan
- Men
- HTML
- HTTPS
- kæmpe
- menneskelig
- Mennesker
- idé
- identificeret
- if
- KIMOs Succeshistorier
- in
- Herunder
- faktisk
- individuel
- indflydelse
- påvirke
- oplysninger
- ubetydelig
- i stedet
- Institut
- interagere
- interaktion
- interaktioner
- ind
- indføre
- uvægerligt
- involvere
- involverede
- involverer
- IT
- ITS
- selv
- jacob
- puslespil
- Job
- lige
- Holde
- holde
- Venlig
- slags
- Kendskab til
- lab
- stor
- Efternavn
- førende
- læring
- Led
- mindre
- Livet
- ligesom
- Sandsynlig
- forbundet
- lidt
- levende
- logik
- Lang
- længere
- kiggede
- maskine
- machine learning
- lavet
- magasin
- større
- lave
- maerker
- måde
- mange
- Massachusetts
- Massachusetts Tekniske Institut
- Match
- materiale
- matematisk
- Matter
- Kan..
- kan være
- meningsfuld
- måle
- målinger
- mekanisme
- mekanismer
- medicinsk
- møder
- Medlemmer
- metoder
- måske
- MIT
- tilstand
- Moduler
- molekylær
- molekyle
- mere
- bevæge sig
- flytning
- meget
- flere
- skal
- utal
- mystisk
- Mystery
- Natur
- I nærheden af
- næsten
- nødvendigvis
- Behov
- behov
- netværk
- ingen
- Noter
- nu
- nuanceret
- talrige
- forekomme
- forekom
- forekommende
- of
- off
- tilbyde
- tit
- on
- engang
- ONE
- kun
- or
- Andet
- vores
- ud
- Resultat
- i løbet af
- samlet
- egen
- pakket
- parringer
- par
- Papir
- parametre
- del
- deltagere
- særlig
- forbi
- Patches
- Betal
- måske
- overtalt
- fysisk
- billede
- stykker
- Planter
- plato
- Platon Data Intelligence
- PlatoData
- Leg
- spillere
- Punkt
- position
- mulig
- magt
- vigtigste
- brug
- forudsige
- forudsagde
- tilstedeværelse
- præsentere
- stolthed
- sandsynligvis
- procedure
- behandle
- producere
- Produkt
- dyb
- Programmer
- projekt
- Protein
- Proteiner
- give
- giver
- bevise
- offentliggjort
- sætte
- puslespil
- spørgsmål
- hurtigt
- helt
- tilfældig
- Sats
- hellere
- Raw
- let
- klar
- ægte
- region
- regioner
- Regulere
- Regulering
- lovgivningsmæssige
- relativt
- frigivet
- stole
- stole
- forblive
- bemærkede
- Fjern
- reparere
- repetitiv
- replikation
- forskere
- dem
- Svar
- resultere
- resulterer
- Resultater
- Ris
- Rich
- højre
- stiv
- Risiko
- Riverside
- RNA
- roller
- Værelse
- rutinemæssigt
- Kør
- Said
- samme
- siger
- forskere
- sikkert
- se
- synes
- tilsyneladende
- synes
- set
- segment
- segmenter
- Gribe
- selektiv
- følsom
- dømme
- september
- Sequence
- tjener
- serveret
- flere
- Seksuel
- lavvandet
- Shape
- Kort
- bør
- Vis
- betydning
- Simpelt
- siden
- enkelt
- websted
- Websteder
- SIX
- Størrelse
- slæk
- mindre
- So
- løsninger
- nogle
- en eller anden måde
- noget
- sommetider
- Kilder
- Space
- specifikke
- specificitet
- stabil
- Stanford
- Stanford University
- Stater
- statistiske
- forblive
- Trin
- stikning
- klæbrig
- Stands
- Historie
- ligetil
- styrke
- stærk
- kraftigt
- strukturel
- struktur
- studerende
- Studere
- Efterfølgende
- væsentligt
- sådan
- sukker
- foreslår
- suite
- support
- sikker
- Omkringliggende
- Schweizisk
- Kontakt
- Schweiz
- systemet
- Systemer
- Tag
- Tandem
- mål
- mål
- Opgaver
- hold
- Teknologier
- fortæller
- tendens
- tiere
- end
- at
- deres
- Them
- selv
- derefter
- Der.
- derfor
- Disse
- de
- ting
- tror
- tænker
- denne
- dem
- selvom?
- tænkte
- tusinder
- Gennem
- hele
- Dermed
- stramt
- tid
- gange
- til
- sammen
- også
- Oversættelse
- gennemsigtig
- fældefangst
- sand
- Drejning
- to
- typen
- typer
- typisk
- UCR
- Ultimativt
- afdækket
- forstå
- universitet
- University of California
- ukendt
- på
- brug
- anvendte
- nyttigt
- sædvanligvis
- Værdier
- forskellige
- Vast
- meget
- levedygtig
- virus
- Voice
- Venter
- var
- Ur
- Vand
- Vej..
- måder
- we
- svag
- WebP
- GODT
- veldefinerede
- Wells
- var
- Hvad
- hvornår
- hvorvidt
- som
- mens
- WHO
- Hele
- helt
- hvorfor
- vilje
- med
- inden for
- uden
- Arbejde
- arbejdede
- Verdens
- orm
- ville
- skriftlig
- skrev
- endnu
- Du
- unge
- zephyrnet