La transcriptómica espacial y la proteómica proporcionan información complementaria que transformó nuestra comprensión de procesos biológicos complejos. Sin embargo, la integración experimental de estas modalidades es limitada.
Un nuevo método llamado secuenciación espacial de proteínas y transcriptomas (SPOTS) puede iluminar las identidades y actividades de las células de un órgano o tumor con una resolución sin precedentes. Desarrollado por investigadores de Weill Cornell Medicine, NewYork-Presbyterian y el New York Genome Center, SPOTS puede iluminar las identidades y actividades de las células de un órgano o un tumor con una resolución sin precedentes.
La técnica conserva información sobre la ubicación precisa de las células mientras registra patrones de actividad genética y la presencia de elementos esenciales. proteínas en células a través de muestras de tejido. Esto hace posible crear “mapas” de órganos intrincados y ricos en datos, incluidos órganos enfermos y tumores, que pueden ser extremadamente útiles tanto en la investigación básica como en la clínica.
El coautor principal del estudio, el Dr. Dan Landau, profesor asociado de medicina en la División de Hematología y Oncología Médica y miembro del Centro Oncológico Sandra y Edward Meyer de Weill Cornell Medicine y miembro principal de la facultad del Centro del Genoma de Nueva York. dicho, "Esta tecnología es apasionante porque nos permite mapear la organización espacial de los tejidos, incluidos los tipos de células, las actividades celulares y las interacciones entre células, como nunca antes".
El nuevo enfoque es parte de una iniciativa más amplia de científicos e ingenieros para crear medios más eficaces de "ver" a nivel microscópico cómo funcionan los órganos y tejidos. En los últimos años se han visto avances significativos en la investigación, en particular en métodos para perfilar la actividad genética y otras capas de datos en células individuales o pequeños grupos de células. Sin embargo, es necesario recuperar la información sobre las ubicaciones originales de las células perfiladas dentro de los tejidos, porque estos enfoques frecuentemente requieren la descomposición de los tejidos y la separación de las células de sus vecinas. La nueva técnica también registra esos datos espaciales y lo hace con una resolución excelente.
El método se basa en parte en la tecnología 10x Genomics existente. Utiliza portaobjetos de vidrio adecuados para obtener imágenes de muestras de tejido con métodos de patología basados en microscopios ordinarios, pero también está recubierto con miles de moléculas sonda particulares.
El “código de barras” químico de cada molécula sonda identifica su ubicación bidimensional en el portaobjetos. Las moléculas sonda en el portaobjetos agarran el ARN mensajeros (ARNm), esencialmente las transcripciones de genes activados, de células vecinas, cuando se coloca una muestra de tejido en rodajas finas en el portaobjetos, sus células se vuelven permeables. En el procedimiento se utilizan anticuerpos de diseño que se unen a las moléculas sonda únicas y a las proteínas de interés en el tejido.
Los investigadores pueden identificar rápida y automáticamente los ARNm recolectados y las proteínas elegidas y mapearlos con precisión en sus ubicaciones originales en toda la muestra de tejido. Los mapas producidos pueden considerarse de forma independiente o en comparación con las imágenes patológicas de rutina de la muestra.
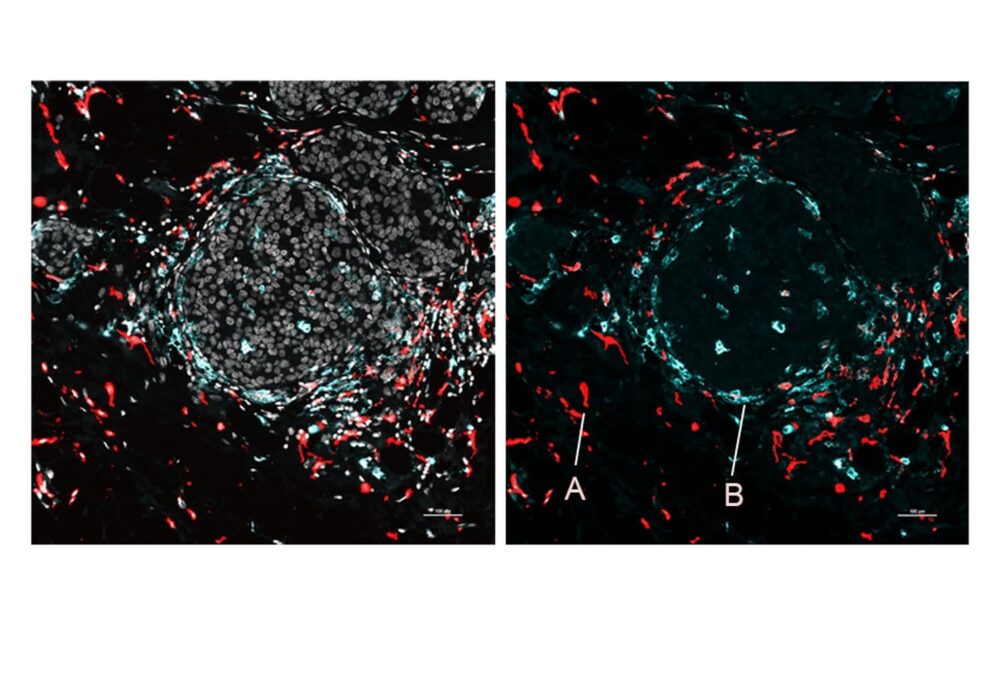
En tejido de un bazo de ratón sano, el equipo utilizó SPOTS para mostrar la intrincada arquitectura funcional de este órgano, incluidos grupos de varios tipos de células, sus estados funcionales y cómo esos estados se alteraban según la ubicación de las células.
Los investigadores también utilizaron SPOTS para mapear la estructura celular de un tumor de mama de ratón, destacando su potencial para su uso en la investigación del cáncer. El mapa generado mostró macrófagos y células inmunes en dos estados diferentes, cada uno indicado por un marcador proteico diferente: una condición estaba activa y luchaba contra los tumores, mientras que la otra era inmunosupresora y establecía una barrera para proteger el tumor.
El Dr. Landau, oncólogo del Centro Médico NewYork-Presbyterian/Weill Cornell, dijo, "Pudimos ver que estos dos subconjuntos de macrófagos se encuentran en diferentes áreas del tumor e interactúan con diferentes células, y esa diferencia en el microambiente probablemente impulse sus distintos estados de actividad".
"Estos detalles del entorno inmunológico del tumor (detalles que a menudo no pueden resolverse debido a la escasez de células inmunes dentro de los tumores) podrían ayudar a explicar por qué algunos pacientes responden a terapia de refuerzo inmunológico y otros no, y por lo tanto podrían contribuir al diseño de futuras inmunoterapias”.
“Esta versión inicial de SPOTS tiene una resolución espacial tal que cada “píxel” del conjunto de datos resultante suma información de actividad genética para al menos varias células. Sin embargo, los investigadores esperan pronto reducir esta resolución a células individuales y al mismo tiempo agregar otras capas de información celular clave”.
Referencia de la revista:
- Ben-Chetrit, N., Niu, X., Swett, AD et al. Integración del perfil espacial del transcriptoma completo con marcadores de proteínas. Nat biotecnología (2023). DUELE: 10.1038/s41587-022-01536-3