A fehérjék számos biológiai folyamatot irányítanak, például enzimaktivitást, molekuláris transzportot és sejttámogatást. A fehérje háromdimenziós szerkezete betekintést nyújt a működésébe és más biomolekulákkal való kölcsönhatásba. A fehérjeszerkezet meghatározására szolgáló kísérleti módszerek, mint például a röntgenkrisztallográfia és az NMR-spektroszkópia, drágák és időigényesek.
Ezzel szemben a közelmúltban kifejlesztett számítási módszerek gyorsan és pontosan megjósolhatják egy fehérje szerkezetét annak aminosavszekvenciájából. Ezek a módszerek kritikusak olyan fehérjék esetében, amelyeket nehéz kísérletileg tanulmányozni, mint például a membránfehérjék, amelyek számos gyógyszer célpontjai. Ennek egyik jól ismert példája az AlphaFold, egy mély tanuláson alapuló algoritmus, amelyet a pontos előrejelzéseiért ünnepeltek.
ESMFold egy másik nagyon pontos, mély tanuláson alapuló módszer, amelyet a fehérje szerkezetének aminosavszekvenciájából történő előrejelzésére fejlesztettek ki. Az ESMFold egy nagy fehérjenyelvi modellt (pLM) használ gerincként, és végpontokig működik. Az AlphaFold2-vel ellentétben nem igényel keresést ill Több szekvencia igazítás (MSA) lépésben, és nem támaszkodik külső adatbázisokra az előrejelzések generálásához. Ehelyett a fejlesztőcsapat az UniRef fehérjeszekvenciáinak millióira tanította a modellt. A képzés során a modell figyelmi mintákat alakított ki, amelyek elegánsan reprezentálják a szekvenciában szereplő aminosavak közötti evolúciós kölcsönhatásokat. A pLM használata MSA helyett akár 60-szor gyorsabb előrejelzési időt tesz lehetővé, mint a többi korszerű modell.
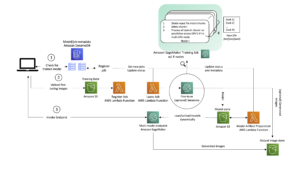
Ebben a bejegyzésben a Hugging Face előre betanított ESMFold modelljét használjuk Amazon SageMaker hogy megjósoljuk a nehézlánc szerkezetét trastuzumabEgy monoklonális antitest először fejlesztette ki Genentech betegségek kezelésére HER2-pozitív emlőrák. A fehérje szerkezetének gyors előrejelzése hasznos lehet, ha a kutatók tesztelni akarják a szekvencia-módosítások hatását. Ez potenciálisan a betegek túlélésének javulásához vagy kevesebb mellékhatáshoz vezethet.
Ez a bejegyzés egy példa a Jupyter-jegyzetfüzetre és a kapcsolódó szkriptekre a következőkben GitHub tárház.
Előfeltételek
Javasoljuk, hogy ezt a példát egy Amazon SageMaker Studio jegyzetfüzet a PyTorch 1.13 Python 3.9 CPU-optimalizált képfájl futtatása ml.r5.xlarge példánytípuson.
Vizualizálja a trastuzumab kísérleti szerkezetét
Kezdésként használjuk a biopython könyvtárat és egy segédszkriptet a trastuzumab szerkezet letöltéséhez a RCSB Protein Data Bank:
Ezután használjuk a py3Dmol könyvtár a szerkezet interaktív 3D vizualizációként való megjelenítéséhez:
A következő ábra a Protein Data Bank (PDB) 3N1Z 8D fehérjeszerkezetét mutatja be. Ezen a képen a trastuzumab könnyű lánca narancssárga, a nehéz lánc kék (a változó régióval világoskék), a HER2 antigén pedig zöld.
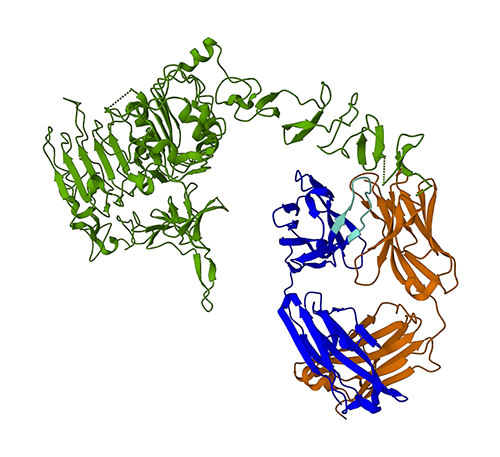
Először az ESMFold segítségével megjósoljuk a nehéz lánc (B lánc) szerkezetét annak aminosavszekvenciájából. Ezután összehasonlítjuk az előrejelzést a fent bemutatott kísérletileg meghatározott szerkezettel.
Jósolja meg a trastuzumab nehézlánc szerkezetét annak szekvenciájából az ESMFold segítségével
Használjuk az ESMFold modellt a nehéz lánc szerkezetének előrejelzésére, és hasonlítsuk össze a kísérleti eredménnyel. Kezdésként egy előre beépített notebook-környezetet fogunk használni a Studioban, amely számos fontos könyvtárat tartalmaz, például PyTorch, előre telepítve. Bár használhatunk gyorsított példánytípust a notebook elemzés teljesítményének javítására, ehelyett nem gyorsított példányt használunk, és az ESMFold előrejelzést futtatjuk egy CPU-n.
Először betöltjük az előre betanított ESMFold modellt és tokenizátort innen Átölelő Arc Hub:
Ezután átmásoljuk a modellt az eszközünkre (ebben az esetben a CPU-ra), és beállítunk néhány modellparamétert:
A fehérjeszekvencia elemzésre való előkészítéséhez tokenizálnunk kell. Ez lefordítja az aminosav-szimbólumokat (EVQLV…) az ESMFold modell által is értelmezhető numerikus formátumba (6,19,5,10,19,…):
Ezután a tokenizált bemenetet a módba másoljuk, előrejelzést készítünk, és az eredményt fájlba mentjük:
Ez körülbelül 3 percet vesz igénybe egy nem gyorsított példánytípuson, például egy r5 esetén.
Az ESMFold előrejelzés pontosságát a kísérleti szerkezettel való összehasonlítással ellenőrizhetjük. Ezt a US-Align eszköz, amelyet a Michigani Egyetem Zhang Labja fejlesztett ki:
| PDBchain1 | PDBchain2 | TM-Score |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
A sablon modellezési pontszám (TM-score) a fehérjeszerkezetek hasonlóságának felmérésére szolgáló mérőszám. Az 1.0 pont tökéletes egyezést jelez. A 0.7 feletti pontszámok azt jelzik, hogy a fehérjéknek ugyanaz a gerincszerkezete. A 0.9 feletti pontszámok azt jelzik, hogy a fehérjék funkcionálisan cserélhető későbbi használatra. A TM-Score 0.802 elérése esetén az ESMFold előrejelzés valószínűleg megfelelő lenne olyan alkalmazásokhoz, mint a szerkezetpontozás vagy a ligandumkötési kísérletek, de előfordulhat, hogy nem megfelelő olyan felhasználási esetekben, mint pl. molekuláris helyettesítés amelyek rendkívül nagy pontosságot igényelnek.
Ezt az eredményt az igazított struktúrák megjelenítésével tudjuk érvényesíteni. A két szerkezet nagy, de nem tökéletes átfedést mutat. A fehérjeszerkezet előrejelzése egy gyorsan fejlődő terület, és sok kutatócsoport egyre pontosabb algoritmusokat fejleszt!
Telepítse az ESMFoldot SageMaker következtetési végpontként
A modellkövetkeztetés notebookban való futtatása megfelelő a kísérletezéshez, de mi van, ha integrálnia kell a modellt egy alkalmazással? Vagy egy MLOps csővezeték? Ebben az esetben jobb megoldás, ha a modellt következtetési végpontként telepíti. A következő példában az ESMFoldot SageMaker valós idejű következtetési végpontként telepítjük egy gyorsított példányon. A SageMaker valós idejű végpontjai méretezhető, költséghatékony és biztonságos módot kínálnak a gépi tanulási (ML) modellek üzembe helyezésére és tárolására. Az automatikus skálázással beállíthatja a végpontot futtató példányok számát, hogy megfeleljen az alkalmazás igényeinek, optimalizálva a költségeket és biztosítva a magas rendelkezésre állást.
Az előre épített SageMaker konténer átölelő archoz megkönnyíti a mély tanulási modellek telepítését a gyakori feladatokhoz. Azonban az olyan újszerű felhasználási esetekben, mint a fehérjeszerkezet előrejelzése, meg kell határoznunk egy szokást inference.py parancsfájlt a modell betöltéséhez, az előrejelzés futtatásához és a kimenet formázásához. Ez a szkript nagyjából ugyanazt a kódot tartalmazza, amelyet a notebookunkban is használtunk. Létrehozunk továbbá a requirements.txt fájlt, hogy meghatározzon néhány Python-függőséget a végpontunk számára. Az általunk létrehozott fájlokat a GitHub tárház.
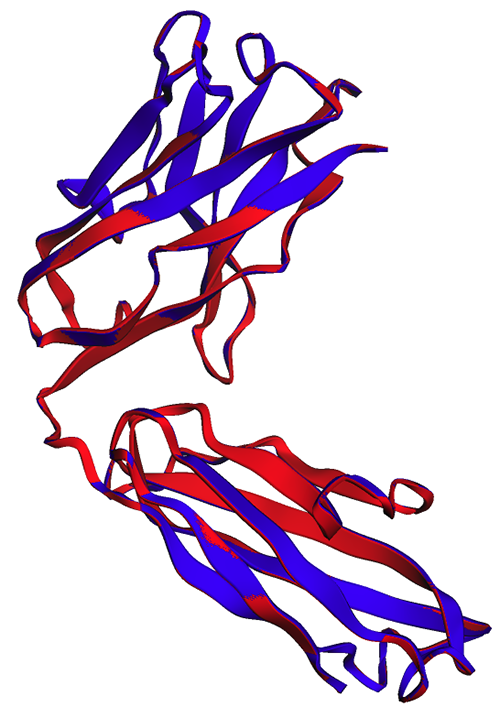
A következő ábrán a trastuzumab nehézláncának kísérleti (kék) és előre jelzett (piros) szerkezete nagyon hasonló, de nem azonos.

Miután létrehoztuk a szükséges fájlokat a code könyvtárat, a SageMaker segítségével telepítjük a modellünket HuggingFaceModel osztály. Ez egy előre beépített tárolót használ, hogy leegyszerűsítse a Hugging Face modellek SageMaker rendszerbe való telepítésének folyamatát. Vegye figyelembe, hogy a végpont létrehozása akár 10 percet is igénybe vehet, a rendelkezésre állástól függően ml.g4dn példánytípusok régiónkban.
Amikor a végpont telepítése befejeződött, újra elküldhetjük a fehérjeszekvenciát, és megjeleníthetjük az előrejelzés első néhány sorát:
Mivel a végpontunkat egy gyorsított példányra telepítettük, az előrejelzés csak néhány másodpercet vesz igénybe. Az eredmény minden sora egyetlen atomnak felel meg, és tartalmazza az aminosav azonosságot, három térbeli koordinátát és egy pLDDT pontszám az adott helyen az előrejelzési megbízhatóságot reprezentálja.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | FOGLALKOZÁS | PLDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Ugyanazt a módszert használva, mint korábban, azt látjuk, hogy a notebook és a végpont előrejelzése azonos.
| PDBchain1 | PDBchain2 | TM-Score |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
Amint az a következő ábrán látható, a jegyzetfüzetben (piros) és a végpont által (kék) generált ESMFold előrejelzések tökéletes igazodást mutatnak.
Tisztítsuk meg
A további költségek elkerülése érdekében töröljük a következtetési végpontunkat és a tesztadatokat:
Összegzésként
A számítási fehérjeszerkezet-előrejelzés kritikus eszköz a fehérjék működésének megértéséhez. Az alapkutatáson kívül az olyan algoritmusok, mint az AlphaFold és az ESMFold, számos alkalmazási területtel rendelkeznek az orvostudományban és a biotechnológiában. Az e modellek által generált szerkezeti betekintések segítenek jobban megérteni a biomolekulák kölcsönhatását. Ez jobb diagnosztikai eszközökhöz és terápiákhoz vezethet a betegek számára.
Ebben a bejegyzésben bemutatjuk, hogyan telepítheti az ESMFold fehérjenyelvi modellt a Hugging Face Hubból méretezhető következtetési végpontként a SageMaker segítségével. A Hugging Face modellek SageMakeren történő telepítésével kapcsolatos további információkért lásd: Használja Hugging Face-t az Amazon SageMakerrel. További fehérjetudományi példákat is találhat a Félelmetes fehérjeelemzés az AWS-en GitHub repó. Kérjük, írjon nekünk megjegyzést, ha van más példa, amelyet szeretne látni!
A szerzőkről
 Brian hűséges az Amazon Web Services globális egészségügyi és élettudományi csapatának vezető AI/ML megoldások építésze. Több mint 17 éves tapasztalattal rendelkezik a biotechnológia és a gépi tanulás terén, és szenvedélyesen segíti az ügyfeleket a genomikai és proteomikai kihívások megoldásában. Szabadidejében szívesen főz és étkezik barátaival és családjával.
Brian hűséges az Amazon Web Services globális egészségügyi és élettudományi csapatának vezető AI/ML megoldások építésze. Több mint 17 éves tapasztalattal rendelkezik a biotechnológia és a gépi tanulás terén, és szenvedélyesen segíti az ügyfeleket a genomikai és proteomikai kihívások megoldásában. Szabadidejében szívesen főz és étkezik barátaival és családjával.
 Shamika Ariyawansa AI/ML Specialist Solutions Architect az Amazon Web Services globális egészségügyi és élettudományi csapatában. Szenvedélyesen dolgozik az ügyfelekkel, hogy felgyorsítsa a mesterséges intelligencia és az ML elterjedését azáltal, hogy technikai útmutatást ad, és segít nekik az innovációban és biztonságos felhőmegoldások kiépítésében az AWS-en. A munkán kívül szeret síelni és terepezni.
Shamika Ariyawansa AI/ML Specialist Solutions Architect az Amazon Web Services globális egészségügyi és élettudományi csapatában. Szenvedélyesen dolgozik az ügyfelekkel, hogy felgyorsítsa a mesterséges intelligencia és az ML elterjedését azáltal, hogy technikai útmutatást ad, és segít nekik az innovációban és biztonságos felhőmegoldások kiépítésében az AWS-en. A munkán kívül szeret síelni és terepezni.
 Yanjun Qi az AWS Machine Learning Solution Lab vezető alkalmazott tudományos menedzsere. Megújítja és alkalmazza a gépi tanulást, hogy segítse az AWS-ügyfeleket az AI és a felhő alkalmazásának felgyorsításában.
Yanjun Qi az AWS Machine Learning Solution Lab vezető alkalmazott tudományos menedzsere. Megújítja és alkalmazza a gépi tanulást, hogy segítse az AWS-ügyfeleket az AI és a felhő alkalmazásának felgyorsításában.
- SEO által támogatott tartalom és PR terjesztés. Erősödjön még ma.
- PlatoAiStream. Web3 adatintelligencia. Felerősített tudás. Hozzáférés itt.
- A jövő pénzverése – Adryenn Ashley. Hozzáférés itt.
- Részvények vásárlása és eladása PRE-IPO társaságokban a PREIPO® segítségével. Hozzáférés itt.
- Forrás: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :van
- :is
- :nem
- $ UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Rólunk
- felett
- AC
- gyorsul
- felgyorsult
- pontosság
- pontos
- pontosan
- elérése
- tevékenység
- mellett
- Örökbefogadás
- AI
- AI / ML
- algoritmus
- algoritmusok
- igazított
- igazítás
- Is
- Bár
- amazon
- Amazon SageMaker
- Az Amazon Web Services
- an
- elemzés
- és a
- Másik
- bármilyen
- Alkalmazás
- alkalmazások
- alkalmazott
- megfelelő
- VANNAK
- AS
- értékelése
- At
- atom
- figyelem
- Automatikus
- elérhetőség
- elkerülése érdekében
- AWS
- AWS gépi tanulás
- Hátgerinc
- Bank
- alapvető
- BE
- előtt
- kezdődik
- Jobb
- között
- kötés
- biotechnológia
- Kék
- épít
- de
- by
- TUD
- Rák
- rajzfilm
- eset
- esetek
- ünnepelt
- lánc
- kihívások
- díjak
- ellenőrizze
- osztály
- felhő
- felhő elfogadása
- kód
- szín
- jön
- megjegyzés
- Közös
- összehasonlítani
- összehasonlítva
- teljes
- bizalom
- Konténer
- kontraszt
- megfelel
- költséghatékony
- kiadások
- tudott
- teremt
- készítette
- kritikai
- Ügyfelek
- dátum
- adatbázisok
- dátum idő
- mély
- mély tanulás
- DeepMind
- Fok
- igények
- attól
- telepíteni
- telepített
- bevezetéséhez
- bevetés
- Határozzuk meg
- eltökélt
- fejlett
- fejlesztése
- Fejlesztés
- eszköz
- nehéz
- kijelző
- do
- nem
- Nem
- letöltés
- hajtás
- Kábítószer
- alatt
- minden
- könnyű
- hatás
- hatások
- lehetővé teszi
- végén
- Endpoint
- biztosítása
- Környezet
- példa
- példák
- drága
- tapasztalat
- kísérletek
- külső
- rendkívüli módon
- Arc
- család
- gyorsabb
- kevés
- kevesebb
- mező
- Ábra
- filé
- Fájlok
- Találjon
- végén
- vezetéknév
- következő
- A
- formátum
- barátok
- ból ből
- funkció
- további
- generál
- generált
- GitHub
- Globális
- Zöld
- útmutatást
- Legyen
- he
- egészségügyi
- nehéz
- segít
- segít
- Magas
- övé
- vendéglátó
- Hogyan
- How To
- azonban
- HTML
- HTTPS
- Kerékagy
- HuggingFace
- identiques
- Identitás
- if
- kép
- importál
- fontos
- javul
- javított
- in
- magában foglalja a
- jelez
- jelzi
- információ
- újít
- bemenet
- Insight
- meglátások
- példa
- helyette
- integrálni
- kölcsönhatásba
- kölcsönhatások
- interaktív
- kölcsönhatásba lép
- bele
- IT
- ITS
- labor
- nyelv
- nagy
- vezet
- tanulás
- Szabadság
- könyvtárak
- könyvtár
- élet
- Life Sciences
- fény
- mint
- Valószínű
- kiszámításának
- elhelyezkedés
- lookup
- gép
- gépi tanulás
- csinál
- KÉSZÍT
- menedzser
- sok
- Mérkőzés
- Lehet..
- orvostudomány
- Találkozik
- módszer
- mód
- metrikus
- Michigan
- Több millió
- Perc
- ML
- MLOps
- Mód
- modell
- modellek
- Módosítások
- molekuláris
- több
- sok
- név
- elengedhetetlen
- Szükség
- NIH
- jegyzetfüzet
- regény
- szám
- objektumok
- elavult
- of
- on
- ONE
- csak
- működik
- optimalizálása
- opció
- or
- narancs
- OS
- Más
- mi
- teljesítmény
- kívül
- paraméterek
- szenvedélyes
- beteg
- betegek
- minták
- tökéletes
- teljesítmény
- csővezeték
- Plató
- Platón adatintelligencia
- PlatoData
- kérem
- állás
- potenciálisan
- előre
- jósolt
- előrejelzésére
- előrejelzés
- Tippek
- Készít
- korábban
- folyamat
- Folyamatok
- Fehérje
- Fehérjék
- ad
- biztosít
- amely
- Piton
- pytorch
- Qi
- gyorsan
- gyorsan
- gyorsan fejlődő terület
- real-time
- ajánl
- Piros
- vidék
- összefüggő
- támaszkodnak
- képvisel
- képviselő
- jelentése
- szükség
- kutatás
- kutatók
- eredményez
- SOR
- futás
- futás
- sagemaker
- SageMaker következtetés
- azonos
- Megtakarítás
- skálázható
- skálázás
- Tudomány
- TUDOMÁNYOK
- pontszám
- pontozás
- szkriptek
- másodperc
- biztonság
- lát
- idősebb
- Sorozat
- Szolgáltatások
- készlet
- számos
- Megosztás
- ő
- kellene
- előadás
- mutatott
- oldal
- hasonló
- egyszerűsítése
- egyetlen
- megoldások
- Megoldások
- SOLVE
- néhány
- térbeli
- szakember
- spektroszkópia
- sebesség
- kezdet
- csúcs-
- Lépés
- szerkezeti
- struktúra
- stúdió
- Tanulmány
- ilyen
- megfelelő
- támogatás
- túlélés
- Vesz
- tart
- célok
- feladatok
- csapat
- csapat
- Műszaki
- teszt
- mint
- hogy
- A
- azok
- Őket
- akkor
- Ott.
- Ezek
- ezt
- három
- háromdimenziós
- idő
- időigényes
- alkalommal
- nak nek
- tokenizálni
- Vezérjeles
- szerszám
- szerszámok
- fáklya
- kiképzett
- Képzések
- transzformerek
- szállítható
- kezelés
- kettő
- típus
- típusok
- megért
- megértés
- egyetemi
- nem úgy mint
- us
- használ
- használt
- segítségével
- ÉRVÉNYESÍT
- nagyon
- Megnézem
- megjelenítés
- W
- kívánatos
- Út..
- we
- háló
- webes szolgáltatások
- jól ismert
- Mit
- Wikipedia
- lesz
- val vel
- WordPress
- Munka
- művek
- lenne
- röntgen
- te
- A te
- zephyrnet