Protein menggerakkan banyak proses biologis, seperti aktivitas enzim, transportasi molekuler, dan dukungan seluler. Struktur tiga dimensi protein memberikan wawasan tentang fungsinya dan bagaimana ia berinteraksi dengan biomolekul lain. Metode eksperimental untuk menentukan struktur protein, seperti kristalografi sinar-X dan spektroskopi NMR, mahal dan memakan waktu.
Sebaliknya, metode komputasi yang dikembangkan baru-baru ini dapat dengan cepat dan akurat memprediksi struktur protein dari urutan asam aminonya. Metode ini sangat penting untuk protein yang sulit dipelajari secara eksperimental, seperti protein membran, target banyak obat. Salah satu contoh terkenal dari ini adalah Lipatan Alfa, algoritme berbasis pembelajaran mendalam yang terkenal karena prediksi akuratnya.
Lipatan ES adalah metode berbasis pembelajaran mendalam yang sangat akurat yang dikembangkan untuk memprediksi struktur protein dari urutan asam aminonya. ESMFold menggunakan model bahasa protein besar (pLM) sebagai tulang punggung dan beroperasi dari ujung ke ujung. Tidak seperti AlphaFold2, itu tidak memerlukan pencarian atau Penjajaran Urutan Berganda (MSA), juga tidak bergantung pada database eksternal untuk menghasilkan prediksi. Sebaliknya, tim pengembangan melatih model pada jutaan urutan protein dari UniRef. Selama pelatihan, model tersebut mengembangkan pola perhatian yang secara elegan mewakili interaksi evolusioner antara asam amino dalam urutannya. Penggunaan pLM ini sebagai pengganti MSA memungkinkan waktu prediksi hingga 60 kali lebih cepat daripada model canggih lainnya.
Dalam postingan ini, kami menggunakan model ESMFold pra-pelatihan dari Hugging Face with Amazon SageMaker untuk memprediksi struktur rantai berat trastuzumab, Sebuah antibodi monoklonal pertama kali dikembangkan oleh Genentech untuk perawatan Kanker payudara HER2-positif. Memprediksi dengan cepat struktur protein ini dapat bermanfaat jika peneliti ingin menguji efek modifikasi urutan. Ini berpotensi meningkatkan kelangsungan hidup pasien atau lebih sedikit efek samping.
Posting ini memberikan contoh notebook Jupyter dan skrip terkait berikut ini Repositori GitHub.
Prasyarat
Kami merekomendasikan untuk menjalankan contoh ini dalam file Studio Amazon SageMaker buku catatan menjalankan gambar PyTorch 1.13 Python 3.9 yang dioptimalkan CPU pada jenis instans ml.r5.xlarge.
Visualisasikan struktur eksperimental trastuzumab
Untuk memulai, kami menggunakan biopython perpustakaan dan skrip pembantu untuk mengunduh struktur trastuzumab dari Bank Data Protein RCSB:
Selanjutnya, kami menggunakan py3Dmol perpustakaan untuk memvisualisasikan struktur sebagai visualisasi 3D interaktif:
Gambar berikut mewakili struktur protein 3D 1N8Z dari Protein Data Bank (PDB). Pada gambar ini, rantai ringan trastuzumab ditampilkan dalam warna oranye, rantai berat berwarna biru (dengan wilayah variabel berwarna biru muda), dan antigen HER2 berwarna hijau.
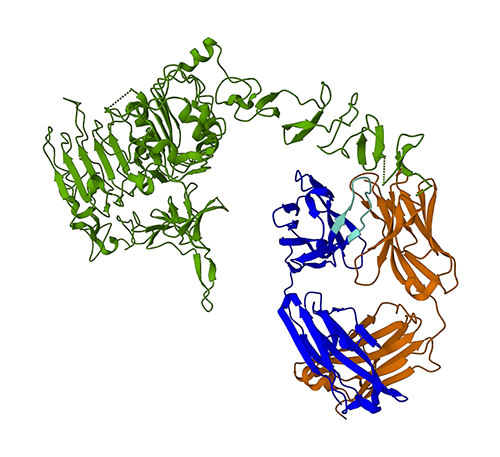
Kami pertama-tama akan menggunakan ESMFold untuk memprediksi struktur rantai berat (Rantai B) dari urutan asam aminonya. Kemudian, kami akan membandingkan prediksi dengan struktur yang ditentukan secara eksperimental yang ditunjukkan di atas.
Prediksi struktur rantai berat trastuzumab dari urutannya menggunakan ESMFold
Mari gunakan model ESMFold untuk memprediksi struktur rantai berat dan membandingkannya dengan hasil eksperimen. Untuk memulai, kita akan menggunakan lingkungan notebook bawaan di Studio yang dilengkapi dengan beberapa pustaka penting, seperti PyTorch, pra-instal. Meskipun kami dapat menggunakan jenis instans yang dipercepat untuk meningkatkan kinerja analisis notebook kami, sebagai gantinya kami akan menggunakan instans non-akselerasi dan menjalankan prediksi ESMFold pada CPU.
Pertama, kami memuat model dan tokenizer ESMFold yang telah dilatih sebelumnya Memeluk Wajah Hub:
Selanjutnya, kami menyalin model ke perangkat kami (CPU dalam hal ini) dan mengatur beberapa parameter model:
Untuk menyiapkan sekuen protein untuk dianalisis, kita perlu menandainya. Ini menerjemahkan simbol asam amino (EVQLV…) ke dalam format numerik yang dapat dipahami oleh model ESMFold (6,19,5,10,19,…):
Selanjutnya, kami menyalin input token ke mode, membuat prediksi, dan menyimpan hasilnya ke file:
Ini membutuhkan waktu sekitar 3 menit pada jenis instans non-akselerasi, seperti r5.
Kami dapat memeriksa keakuratan prediksi ESMFold dengan membandingkannya dengan struktur eksperimental. Kami melakukan ini menggunakan AS-Selaraskan alat yang dikembangkan oleh Zhang Lab di University of Michigan:
| rantai PDB1 | rantai PDB2 | Skor TM |
| data/prediksi.pdb:A | data/eksperimental.pdb:B | 0.802 |
Grafik skor pemodelan template (skor TM) adalah metrik untuk menilai kesamaan struktur protein. Skor 1.0 menunjukkan kecocokan yang sempurna. Skor di atas 0.7 menunjukkan bahwa protein memiliki struktur tulang punggung yang sama. Skor di atas 0.9 menunjukkan bahwa protein tersebut fungsional dapat dipertukarkan untuk penggunaan hilir. Dalam kasus kami untuk mencapai TM-Score 0.802, prediksi ESMFold kemungkinan akan sesuai untuk aplikasi seperti skoring struktur atau eksperimen pengikatan ligan, tetapi mungkin tidak cocok untuk kasus penggunaan seperti penggantian molekul yang membutuhkan ketelitian yang sangat tinggi.
Kami dapat memvalidasi hasil ini dengan memvisualisasikan struktur yang selaras. Kedua struktur menunjukkan tingkat tumpang tindih yang tinggi, tetapi tidak sempurna. Prediksi struktur protein adalah bidang yang berkembang pesat dan banyak tim peneliti sedang mengembangkan algoritme yang semakin akurat!
Terapkan ESMFold sebagai titik akhir inferensi SageMaker
Menjalankan inferensi model di notebook bagus untuk eksperimen, tetapi bagaimana jika Anda perlu mengintegrasikan model Anda dengan aplikasi? Atau pipa MLOps? Dalam hal ini, opsi yang lebih baik adalah menerapkan model Anda sebagai titik akhir inferensi. Dalam contoh berikut, kami akan menerapkan ESMFold sebagai titik akhir inferensi real-time SageMaker pada instans yang dipercepat. Titik akhir real-time SageMaker menyediakan cara yang dapat diskalakan, hemat biaya, dan aman untuk menerapkan dan menghosting model machine learning (ML). Dengan penskalaan otomatis, Anda dapat menyesuaikan jumlah instans yang menjalankan titik akhir untuk memenuhi permintaan aplikasi Anda, mengoptimalkan biaya, dan memastikan ketersediaan yang tinggi.
Yang dibuat sebelumnya Wadah SageMaker untuk Memeluk Wajah memudahkan penerapan model deep learning untuk tugas-tugas umum. Namun, untuk kasus penggunaan baru seperti prediksi struktur protein, kami perlu menentukan kebiasaan inference.py skrip untuk memuat model, menjalankan prediksi, dan memformat output. Skrip ini mencakup banyak kode yang sama yang kami gunakan di notebook kami. Kami juga membuat requirements.txt file untuk menentukan beberapa dependensi Python untuk titik akhir kami untuk digunakan. Anda dapat melihat file yang kami buat di Repositori GitHub.
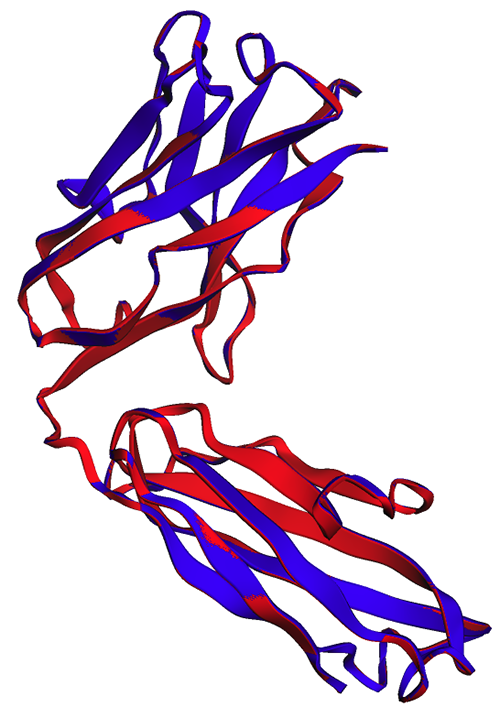
Pada gambar berikut, struktur eksperimental (biru) dan prediksi (merah) dari rantai berat trastuzumab sangat mirip, tetapi tidak identik.

Setelah kami membuat file yang diperlukan di code direktori, kami menerapkan model kami menggunakan SageMaker HuggingFaceModel kelas. Ini menggunakan wadah bawaan untuk menyederhanakan proses penerapan model Hugging Face ke SageMaker. Perhatikan bahwa mungkin diperlukan waktu 10 menit atau lebih untuk membuat titik akhir, bergantung pada ketersediaan ml.g4dn jenis instans di Wilayah kami.
Saat penyebaran titik akhir selesai, kami dapat mengirimkan kembali urutan protein dan menampilkan beberapa baris pertama dari prediksi:
Karena kami menerapkan titik akhir kami ke instans yang dipercepat, prediksi hanya akan memakan waktu beberapa detik. Setiap baris dalam hasil sesuai dengan satu atom dan mencakup identitas asam amino, tiga koordinat spasial, dan a skor pLDDT mewakili kepercayaan prediksi di lokasi itu.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | RANTAI_ID | SEQ_ID | KARTN_X | KARTN_Y | KARTN_Z | OKUPANSI | PLDDT | ATOM_ID |
| ATOM | 1 | N | Glu | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | Glu | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | Glu | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | Glu | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | Glu | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | Glu | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | Glu | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | Glu | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | Glu | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Dengan menggunakan metode yang sama seperti sebelumnya, kami melihat bahwa prediksi notebook dan titik akhir identik.
| rantai PDB1 | rantai PDB2 | Skor TM |
| data/prediksi_titik akhir.pdb:A | data/prediksi.pdb:A | 1.0 |
Seperti yang diamati pada gambar berikut, prediksi ESMFold yang dihasilkan di dalam notebook (merah) dan pada titik akhir (biru) menunjukkan keselarasan yang sempurna.
Membersihkan
Untuk menghindari biaya lebih lanjut, kami menghapus titik akhir inferensi dan data pengujian kami:
Kesimpulan
Prediksi struktur protein komputasi adalah alat penting untuk memahami fungsi protein. Selain penelitian dasar, algoritme seperti AlphaFold dan ESMFold memiliki banyak aplikasi dalam bidang kedokteran dan bioteknologi. Wawasan struktural yang dihasilkan oleh model ini membantu kita lebih memahami bagaimana biomolekul berinteraksi. Ini kemudian dapat mengarah pada alat diagnostik dan terapi yang lebih baik untuk pasien.
Dalam postingan ini, kami menunjukkan cara menerapkan model bahasa protein ESMFold dari Hugging Face Hub sebagai titik akhir inferensi yang dapat diskalakan menggunakan SageMaker. Untuk informasi selengkapnya tentang penerapan model Hugging Face di SageMaker, lihat Gunakan Memeluk Wajah dengan Amazon SageMaker. Anda juga dapat menemukan lebih banyak contoh ilmu protein di Analisis Protein Luar Biasa di AWS repo GitHub. Silakan tinggalkan kami komentar jika ada contoh lain yang ingin Anda lihat!
Tentang Penulis
 Brian Setia adalah Arsitek Solusi AI/ML Senior di tim Kesehatan Global dan Ilmu Hayati di Amazon Web Services. Dia memiliki pengalaman lebih dari 17 tahun dalam bioteknologi dan pembelajaran mesin, dan bersemangat membantu pelanggan memecahkan tantangan genomik dan proteomik. Di waktu luangnya, ia menikmati memasak dan makan bersama teman dan keluarganya.
Brian Setia adalah Arsitek Solusi AI/ML Senior di tim Kesehatan Global dan Ilmu Hayati di Amazon Web Services. Dia memiliki pengalaman lebih dari 17 tahun dalam bioteknologi dan pembelajaran mesin, dan bersemangat membantu pelanggan memecahkan tantangan genomik dan proteomik. Di waktu luangnya, ia menikmati memasak dan makan bersama teman dan keluarganya.
 Shamika Ariyawansa adalah Arsitek Solusi Spesialis AI/ML di tim Kesehatan Global dan Ilmu Hayati di Amazon Web Services. Dia dengan penuh semangat bekerja dengan pelanggan untuk mempercepat adopsi AI dan ML mereka dengan memberikan panduan teknis dan membantu mereka berinovasi dan membangun solusi cloud yang aman di AWS. Di luar pekerjaan, dia suka bermain ski dan off-road.
Shamika Ariyawansa adalah Arsitek Solusi Spesialis AI/ML di tim Kesehatan Global dan Ilmu Hayati di Amazon Web Services. Dia dengan penuh semangat bekerja dengan pelanggan untuk mempercepat adopsi AI dan ML mereka dengan memberikan panduan teknis dan membantu mereka berinovasi dan membangun solusi cloud yang aman di AWS. Di luar pekerjaan, dia suka bermain ski dan off-road.
 Yanjunqi adalah Manajer Sains Terapan Senior di AWS Machine Learning Solution Lab. Dia berinovasi dan menerapkan pembelajaran mesin untuk membantu pelanggan AWS mempercepat penerapan AI dan cloud mereka.
Yanjunqi adalah Manajer Sains Terapan Senior di AWS Machine Learning Solution Lab. Dia berinovasi dan menerapkan pembelajaran mesin untuk membantu pelanggan AWS mempercepat penerapan AI dan cloud mereka.
- Konten Bertenaga SEO & Distribusi PR. Dapatkan Amplifikasi Hari Ini.
- PlatoAiStream. Kecerdasan Data Web3. Pengetahuan Diperkuat. Akses Di Sini.
- Mencetak Masa Depan bersama Adryenn Ashley. Akses Di Sini.
- Beli dan Jual Saham di Perusahaan PRE-IPO dengan PREIPO®. Akses Di Sini.
- Sumber: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :memiliki
- :adalah
- :bukan
- $NAIK
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Tentang Kami
- atas
- AC
- mempercepat
- dipercepat
- ketepatan
- tepat
- akurat
- mencapai
- kegiatan
- tambahan
- Adopsi
- AI
- AI / ML
- algoritma
- algoritma
- selaras
- penjajaran
- juga
- Meskipun
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- analisis
- dan
- Lain
- Apa pun
- Aplikasi
- aplikasi
- terapan
- sesuai
- ADALAH
- AS
- Menilai
- At
- atom
- perhatian
- secara otomatis
- tersedianya
- menghindari
- AWS
- Pembelajaran Mesin AWS
- Tulang punggung
- Bank
- dasar
- BE
- sebelum
- mulai
- Lebih baik
- antara
- mengikat
- bioteknologi
- Biru
- membangun
- tapi
- by
- CAN
- Kanker
- gambar kartun
- kasus
- kasus
- kenamaan
- rantai
- tantangan
- beban
- memeriksa
- kelas
- awan
- adopsi cloud
- kode
- warna
- datang
- komentar
- Umum
- membandingkan
- pembandingan
- lengkap
- kepercayaan
- Wadah
- kontras
- berkorespondensi
- hemat biaya
- Biaya
- bisa
- membuat
- dibuat
- kritis
- pelanggan
- data
- database
- tanggal Waktu
- mendalam
- belajar mendalam
- DeepMind
- Derajat
- tuntutan
- Tergantung
- menyebarkan
- dikerahkan
- penggelaran
- penyebaran
- Menentukan
- ditentukan
- dikembangkan
- berkembang
- Pengembangan
- alat
- sulit
- Display
- do
- tidak
- Tidak
- Download
- mendorong
- Obat-obatan
- selama
- setiap
- Mudah
- efek
- efek
- memungkinkan
- akhir
- Titik akhir
- memastikan
- Lingkungan Hidup
- contoh
- contoh
- mahal
- pengalaman
- eksperimen
- luar
- sangat
- Menghadapi
- keluarga
- lebih cepat
- beberapa
- sedikit
- bidang
- Angka
- File
- File
- Menemukan
- akhir
- Pertama
- berikut
- Untuk
- format
- teman
- dari
- fungsi
- lebih lanjut
- menghasilkan
- dihasilkan
- GitHub
- Aksi
- Hijau
- bimbingan
- Memiliki
- he
- kesehatan
- berat
- membantu
- membantu
- High
- -nya
- tuan rumah
- Seterpercayaapakah Olymp Trade? Kesimpulan
- How To
- Namun
- HTML
- HTTPS
- Pusat
- MemelukWajah
- identik
- identitas
- if
- gambar
- mengimpor
- penting
- memperbaiki
- ditingkatkan
- in
- termasuk
- menunjukkan
- menunjukkan
- informasi
- berinovasi
- memasukkan
- wawasan
- wawasan
- contoh
- sebagai gantinya
- mengintegrasikan
- berinteraksi
- interaksi
- interaktif
- interaktif
- ke
- IT
- NYA
- laboratorium
- bahasa
- besar
- memimpin
- pengetahuan
- Meninggalkan
- perpustakaan
- Perpustakaan
- Hidup
- Biologi
- cahaya
- 'like'
- Mungkin
- memuat
- tempat
- lookup
- mesin
- Mesin belajar
- membuat
- MEMBUAT
- manajer
- banyak
- Cocok
- Mungkin..
- obat
- Pelajari
- metode
- metode
- metrik
- Michigan
- jutaan
- menit
- ML
- MLOps
- mode
- model
- model
- Modifikasi
- molekuler
- lebih
- banyak
- nama
- perlu
- Perlu
- NIH
- buku catatan
- novel
- jumlah
- objek
- usang
- of
- on
- ONE
- hanya
- beroperasi
- mengoptimalkan
- pilihan
- or
- Jeruk
- OS
- Lainnya
- kami
- keluaran
- di luar
- parameter
- bergairah
- pasien
- pasien
- pola
- sempurna
- prestasi
- pipa saluran
- plato
- Kecerdasan Data Plato
- Data Plato
- silahkan
- Pos
- berpotensi
- meramalkan
- diprediksi
- memprediksi
- ramalan
- Prediksi
- Mempersiapkan
- sebelumnya
- proses
- proses
- Protein
- Protein
- memberikan
- menyediakan
- menyediakan
- Ular sanca
- pytorch
- Qi
- segera
- cepat
- bidang yang berkembang pesat
- real-time
- sarankan
- Merah
- wilayah
- terkait
- mengandalkan
- mewakili
- mewakili
- merupakan
- membutuhkan
- penelitian
- peneliti
- mengakibatkan
- BARIS
- Run
- berjalan
- pembuat bijak
- Inferensi SageMaker
- sama
- Save
- terukur
- skala
- Ilmu
- ILMU PENGETAHUAN
- skor
- mencetak gol
- script
- detik
- aman
- melihat
- senior
- Urutan
- Layanan
- set
- beberapa
- Share
- dia
- harus
- Menunjukkan
- ditunjukkan
- sisi
- mirip
- menyederhanakan
- tunggal
- larutan
- Solusi
- MEMECAHKAN
- beberapa
- spasial
- spesialis
- Spektroskopi
- kecepatan
- awal
- state-of-the-art
- Langkah
- struktural
- struktur
- studio
- Belajar
- seperti itu
- cocok
- mendukung
- kelangsungan hidup
- Mengambil
- Dibutuhkan
- target
- tugas
- tim
- tim
- Teknis
- uji
- dari
- bahwa
- Grafik
- mereka
- Mereka
- kemudian
- Sana.
- Ini
- ini
- tiga
- tiga dimensi
- waktu
- membuang-buang waktu
- kali
- untuk
- tokenisasi
- dipatok
- alat
- alat
- obor
- terlatih
- Pelatihan
- transformer
- mengangkut
- pengobatan
- dua
- mengetik
- jenis
- memahami
- pemahaman
- universitas
- tidak seperti
- us
- menggunakan
- bekas
- menggunakan
- MENGESAHKAN
- sangat
- View
- visualisasi
- W
- ingin
- Cara..
- we
- jaringan
- layanan web
- terkenal
- Apa
- Wikipedia
- akan
- dengan
- WordPress
- Kerja
- bekerja
- akan
- x-ray
- Kamu
- Anda
- zephyrnet.dll