Le proteine guidano molti processi biologici, come l'attività enzimatica, il trasporto molecolare e il supporto cellulare. La struttura tridimensionale di una proteina fornisce informazioni sulla sua funzione e su come interagisce con altre biomolecole. I metodi sperimentali per determinare la struttura delle proteine, come la cristallografia a raggi X e la spettroscopia NMR, sono costosi e richiedono tempo.
Al contrario, i metodi computazionali sviluppati di recente possono prevedere in modo rapido e accurato la struttura di una proteina dalla sua sequenza di amminoacidi. Questi metodi sono fondamentali per le proteine che sono difficili da studiare sperimentalmente, come le proteine di membrana, i bersagli di molti farmaci. Un esempio ben noto di questo è AlphaFold, un algoritmo basato sul deep learning celebrato per le sue previsioni accurate.
ESMFold è un altro metodo altamente accurato e basato sull'apprendimento profondo sviluppato per prevedere la struttura delle proteine dalla sua sequenza di amminoacidi. ESMFold utilizza un modello di linguaggio proteico di grandi dimensioni (pLM) come spina dorsale e opera end-to-end. A differenza di AlphaFold2, non ha bisogno di una ricerca o Allineamento di sequenze multiple (MSA), né si basa su database esterni per generare previsioni. Invece, il team di sviluppo ha addestrato il modello su milioni di sequenze proteiche di UniRef. Durante l'allenamento, il modello ha sviluppato schemi di attenzione che rappresentano elegantemente le interazioni evolutive tra gli amminoacidi nella sequenza. Questo utilizzo di un pLM invece di un MSA consente tempi di previsione fino a 60 volte più rapidi rispetto ad altri modelli all'avanguardia.
In questo post, utilizziamo il modello ESMFold pre-addestrato di Hugging Face with Amazon Sage Maker per prevedere la struttura della catena pesante di trastuzumab, un anticorpo monoclonale sviluppato per la prima volta da Genentech per il trattamento di Cancro al seno HER2-positivo. Prevedere rapidamente la struttura di questa proteina potrebbe essere utile se i ricercatori volessero testare l'effetto delle modifiche di sequenza. Ciò potrebbe potenzialmente portare a una migliore sopravvivenza del paziente o a minori effetti collaterali.
Questo post fornisce un notebook Jupyter di esempio e gli script correlati di seguito Repository GitHub.
Prerequisiti
Si consiglia di eseguire questo esempio in un file Amazon Sage Maker Studio taccuino eseguire l'immagine ottimizzata per CPU PyTorch 1.13 Python 3.9 su un tipo di istanza ml.r5.xlarge.
Visualizza la struttura sperimentale di trastuzumab
Per iniziare, usiamo il biopython library e uno script helper per scaricare la struttura di trastuzumab dal file Banca dati delle proteine RCSB:
Successivamente, usiamo il py3Dmol libreria per visualizzare la struttura come una visualizzazione 3D interattiva:
La figura seguente rappresenta la struttura proteica 3D 1N8Z dal Protein Data Bank (PDB). In questa immagine, la catena leggera del trastuzumab è visualizzata in arancione, la catena pesante è blu (con la regione variabile in azzurro) e l'antigene HER2 è verde.
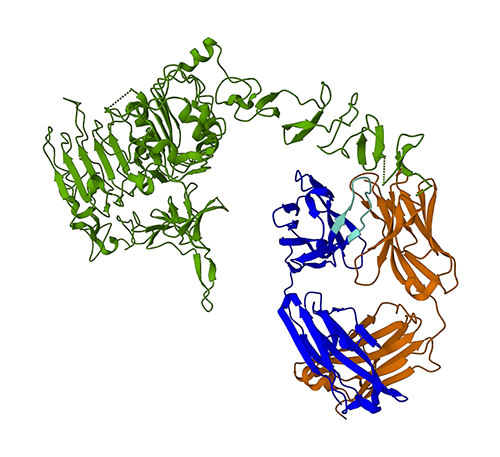
Per prima cosa utilizzeremo ESMFold per predire la struttura della catena pesante (Catena B) dalla sua sequenza amminoacidica. Quindi, confronteremo la previsione con la struttura determinata sperimentalmente mostrata sopra.
Prevedi la struttura della catena pesante del trastuzumab dalla sua sequenza utilizzando ESMFold
Usiamo il modello ESMFold per prevedere la struttura della catena pesante e confrontarla con il risultato sperimentale. Per iniziare, utilizzeremo un ambiente notebook predefinito in Studio che viene fornito con diverse librerie importanti, come PyTorch, pre installato. Anche se potremmo utilizzare un tipo di istanza accelerata per migliorare le prestazioni della nostra analisi del notebook, utilizzeremo invece un'istanza non accelerata ed eseguiremo la previsione ESMFold su una CPU.
Per prima cosa, carichiamo il modello e il tokenizer ESMFold pre-addestrati da Hub per il viso che abbraccia:
Successivamente, copiamo il modello sul nostro dispositivo (CPU in questo caso) e impostiamo alcuni parametri del modello:
Per preparare la sequenza proteica per l'analisi, dobbiamo tokenizzarla. Questo traduce i simboli degli amminoacidi (EVQLV...) in un formato numerico che il modello ESMFold può comprendere (6,19,5,10,19,...):
Successivamente, copiamo l'input tokenizzato nella modalità, facciamo una previsione e salviamo il risultato in un file:
Questo richiede circa 3 minuti su un tipo di istanza non accelerata, come una r5.
Possiamo verificare l'accuratezza della previsione ESMFold confrontandola con la struttura sperimentale. Lo facciamo usando il Allineamento USA strumento sviluppato dallo Zhang Lab dell'Università del Michigan:
| PDBchain1 | PDBchain2 | Punteggio TM |
| dati/previsione.pdb:A | dati/sperimentale.pdb:B | 0.802 |
Il punteggio di modellazione del modello (TM-score) è una metrica per valutare la somiglianza delle strutture proteiche. Un punteggio di 1.0 indica una corrispondenza perfetta. Punteggi superiori a 0.7 indicano che le proteine condividono la stessa struttura della spina dorsale. I punteggi superiori a 0.9 indicano che le proteine lo sono funzionalmente intercambiabili per uso a valle. Nel nostro caso di raggiungimento del punteggio TM 0.802, la previsione ESMFold sarebbe probabilmente appropriata per applicazioni come il punteggio della struttura o gli esperimenti di associazione di ligandi, ma potrebbe non essere adatta per casi d'uso come sostituzione molecolare che richiedono una precisione estremamente elevata.
Possiamo convalidare questo risultato visualizzando le strutture allineate. Le due strutture presentano un elevato, ma non perfetto, grado di sovrapposizione. Le previsioni sulla struttura delle proteine sono un campo in rapida evoluzione e molti team di ricerca stanno sviluppando algoritmi sempre più accurati!
Distribuisci ESMFold come endpoint di inferenza SageMaker
L'esecuzione dell'inferenza del modello in un notebook va bene per la sperimentazione, ma cosa succede se è necessario integrare il modello con un'applicazione? O una pipeline MLOps? In questo caso, un'opzione migliore consiste nel distribuire il modello come endpoint di inferenza. Nell'esempio seguente, distribuiremo ESMFold come endpoint di inferenza in tempo reale SageMaker su un'istanza accelerata. Gli endpoint in tempo reale di SageMaker forniscono un modo scalabile, economico e sicuro per distribuire e ospitare modelli di machine learning (ML). Con il ridimensionamento automatico, puoi regolare il numero di istanze che eseguono l'endpoint per soddisfare le esigenze della tua applicazione, ottimizzando i costi e garantendo un'elevata disponibilità.
Il prefabbricato Contenitore SageMaker per Hugging Face semplifica l'implementazione di modelli di deep learning per attività comuni. Tuttavia, per nuovi casi d'uso come la previsione della struttura delle proteine, dobbiamo definire un'usanza inference.py script per caricare il modello, eseguire la stima e formattare l'output. Questo script include gran parte dello stesso codice che abbiamo usato nel nostro notebook. Creiamo anche un requirements.txt file per definire alcune dipendenze Python per il nostro endpoint da utilizzare. Puoi vedere i file che abbiamo creato nel formato Repository GitHub.
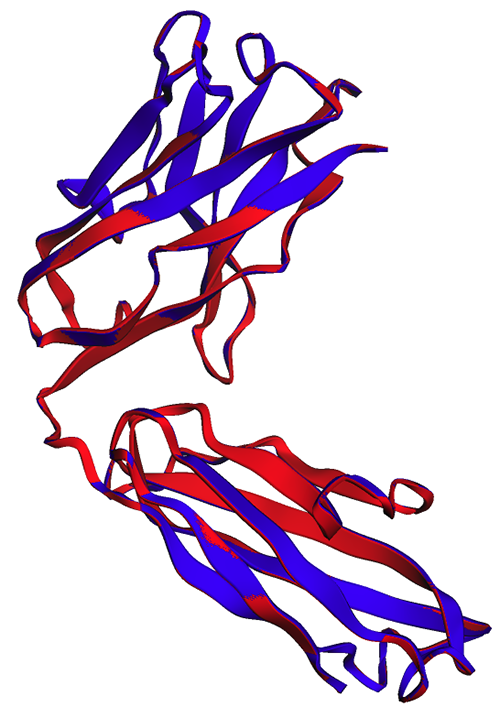
Nella figura seguente, le strutture sperimentali (blu) e previste (rosse) della catena pesante del trastuzumab sono molto simili, ma non identiche.

Dopo aver creato i file necessari nel file code directory, distribuiamo il nostro modello utilizzando SageMaker HuggingFaceModel classe. Questo utilizza un contenitore predefinito per semplificare il processo di distribuzione dei modelli Hugging Face in SageMaker. Si noti che potrebbero essere necessari 10 minuti o più per creare l'endpoint, a seconda della disponibilità di ml.g4dn tipi di istanza nella nostra regione.
Quando la distribuzione dell'endpoint è completa, possiamo inviare nuovamente la sequenza proteica e visualizzare le prime righe della previsione:
Poiché abbiamo distribuito il nostro endpoint a un'istanza accelerata, la previsione dovrebbe richiedere solo pochi secondi. Ogni riga nel risultato corrisponde a un singolo atomo e include l'identità dell'amminoacido, tre coordinate spaziali e a Punteggio pLDDT che rappresenta la confidenza della previsione in quella posizione.
| GRUPPO_PDB | ID | ETICHETTA_ATOMO | RES_ID | CATENA_ID | ID_SEQ | CART_X | CARTON_Y | CART_Z | OCCUPAZIONE | PLDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | all'19.953 ottobre | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | all'19.595 ottobre | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | all'18.693 ottobre | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | all'18.906 ottobre | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | all'17.715 ottobre | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | all'18.694 ottobre | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | all'18.042 ottobre | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | all'17.805 ottobre | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | all'17.767 ottobre | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | all'19.063 ottobre | all'0.258 ottobre | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | all'18.309 ottobre | all'1.415 ottobre | 1 | 0.87 | C |
Utilizzando lo stesso metodo di prima, vediamo che le previsioni del notebook e dell'endpoint sono identiche.
| PDBchain1 | PDBchain2 | Punteggio TM |
| data/endpoint_prediction.pdb:A | dati/previsione.pdb:A | 1.0 |
Come osservato nella figura seguente, le previsioni ESMFold generate nel notebook (rosso) e dall'endpoint (blu) mostrano un allineamento perfetto.
ripulire
Per evitare ulteriori addebiti, eliminiamo il nostro endpoint di inferenza e i dati di test:
Sommario
La previsione computazionale della struttura proteica è uno strumento fondamentale per comprendere la funzione delle proteine. Oltre alla ricerca di base, algoritmi come AlphaFold ed ESMFold hanno molte applicazioni in medicina e biotecnologia. Le intuizioni strutturali generate da questi modelli ci aiutano a capire meglio come interagiscono le biomolecole. Ciò può quindi portare a migliori strumenti diagnostici e terapie per i pazienti.
In questo post, mostriamo come distribuire il modello di linguaggio proteico ESMFold da Hugging Face Hub come endpoint di inferenza scalabile utilizzando SageMaker. Per ulteriori informazioni sulla distribuzione dei modelli Hugging Face su SageMaker, fare riferimento a Usa Hugging Face con Amazon SageMaker. Puoi anche trovare altri esempi di scienza delle proteine nel Impressionante analisi delle proteine su AWS Repository GitHub. Per favore lasciaci un commento se ci sono altri esempi che vorresti vedere!
Informazioni sugli autori
 Brian leale è Senior AI/ML Solutions Architect nel team Global Healthcare and Life Sciences di Amazon Web Services. Ha più di 17 anni di esperienza nella biotecnologia e nell'apprendimento automatico ed è appassionato di aiutare i clienti a risolvere le sfide genomiche e proteomiche. Nel tempo libero ama cucinare e mangiare con i suoi amici e la sua famiglia.
Brian leale è Senior AI/ML Solutions Architect nel team Global Healthcare and Life Sciences di Amazon Web Services. Ha più di 17 anni di esperienza nella biotecnologia e nell'apprendimento automatico ed è appassionato di aiutare i clienti a risolvere le sfide genomiche e proteomiche. Nel tempo libero ama cucinare e mangiare con i suoi amici e la sua famiglia.
 Shamika Ariyawansa è un AI/ML Specialist Solutions Architect nel team Global Healthcare and Life Sciences di Amazon Web Services. Lavora con passione con i clienti per accelerare la loro adozione di IA e ML fornendo indicazioni tecniche e aiutandoli a innovare e creare soluzioni cloud sicure su AWS. Al di fuori del lavoro, ama lo sci e il fuoristrada.
Shamika Ariyawansa è un AI/ML Specialist Solutions Architect nel team Global Healthcare and Life Sciences di Amazon Web Services. Lavora con passione con i clienti per accelerare la loro adozione di IA e ML fornendo indicazioni tecniche e aiutandoli a innovare e creare soluzioni cloud sicure su AWS. Al di fuori del lavoro, ama lo sci e il fuoristrada.
 Yanjun Qi è Senior Applied Science Manager presso l'AWS Machine Learning Solution Lab. Innova e applica l'apprendimento automatico per aiutare i clienti AWS ad accelerare l'adozione dell'intelligenza artificiale e del cloud.
Yanjun Qi è Senior Applied Science Manager presso l'AWS Machine Learning Solution Lab. Innova e applica l'apprendimento automatico per aiutare i clienti AWS ad accelerare l'adozione dell'intelligenza artificiale e del cloud.
- Distribuzione di contenuti basati su SEO e PR. Ricevi amplificazione oggi.
- PlatoAiStream. Intelligenza dei dati Web3. Conoscenza amplificata. Accedi qui.
- Coniare il futuro con Adryenn Ashley. Accedi qui.
- Acquista e vendi azioni in società PRE-IPO con PREIPO®. Accedi qui.
- Fonte: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :ha
- :È
- :non
- $ SU
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- WRI
- sopra
- AC
- accelerare
- accelerata
- precisione
- preciso
- con precisione
- il raggiungimento
- attività
- aggiunta
- Adozione
- AI
- AI / ML
- algoritmo
- Algoritmi
- Allineati
- allineamento
- anche
- Sebbene il
- Amazon
- Amazon Sage Maker
- Amazon Web Services
- an
- .
- ed
- Un altro
- in qualsiasi
- Applicazioni
- applicazioni
- applicato
- opportuno
- SONO
- AS
- valutare
- At
- atomo
- attenzione
- Automatico
- disponibilità
- evitare
- AWS
- Apprendimento automatico di AWS
- Spina dorsale
- Banca
- basic
- BE
- prima
- iniziare
- Meglio
- fra
- rilegatura
- biotecnologie
- Blu
- costruire
- ma
- by
- Materiale
- Cancro
- cartone animato
- Custodie
- casi
- celebre
- catena
- sfide
- oneri
- dai un'occhiata
- classe
- Cloud
- adozione del cloud
- codice
- colore
- viene
- commento
- Uncommon
- confrontare
- confronto
- completamento di una
- fiducia
- Contenitore
- contrasto
- corrisponde
- costo effettivo
- Costi
- potuto
- creare
- creato
- critico
- Clienti
- dati
- banche dati
- datetime
- deep
- apprendimento profondo
- DeepMind
- Laurea
- richieste
- Dipendente
- schierare
- schierato
- distribuzione
- deployment
- Determinare
- determinato
- sviluppato
- in via di sviluppo
- Mercato
- dispositivo
- difficile
- Dsiplay
- do
- effettua
- non
- scaricare
- guidare
- farmaci
- durante
- ogni
- facile
- effetto
- effetti
- Abilita
- fine
- endpoint
- assicurando
- Ambiente
- esempio
- Esempi
- costoso
- esperienza
- esperimenti
- esterno
- estremamente
- Faccia
- famiglia
- più veloce
- pochi
- meno
- campo
- figura
- Compila il
- File
- Trovate
- sottile
- Nome
- i seguenti
- Nel
- formato
- amici
- da
- function
- ulteriormente
- generare
- generato
- GitHub
- globali
- Green
- guida
- Avere
- he
- assistenza sanitaria
- pesante
- Aiuto
- aiutare
- Alta
- il suo
- host
- Come
- Tutorial
- Tuttavia
- HTML
- HTTPS
- Hub
- abbracciare il viso
- identico
- Identità
- if
- Immagine
- importare
- importante
- competenze
- migliorata
- in
- inclusi
- indicare
- indica
- informazioni
- innovare
- ingresso
- intuizione
- intuizioni
- esempio
- invece
- integrare
- interagire
- interazioni
- interattivo
- interagisce
- ai miglioramenti
- IT
- SUO
- laboratorio
- Lingua
- grandi
- portare
- apprendimento
- Lasciare
- biblioteche
- Biblioteca
- Vita
- Life Sciences
- leggera
- piace
- probabile
- caricare
- località
- ricerca
- macchina
- machine learning
- make
- FA
- direttore
- molti
- partita
- Maggio..
- medicina
- Soddisfare
- metodo
- metodi
- metrico
- Michigan
- milioni
- Minuti
- ML
- MLOp
- Moda
- modello
- modelli
- modifiche
- molecolare
- Scopri di più
- molti
- Nome
- necessaria
- Bisogno
- NIH
- taccuino
- romanzo
- numero
- oggetti
- obsoleto
- of
- on
- ONE
- esclusivamente
- opera
- ottimizzazione
- Opzione
- or
- Arancio
- OS
- Altro
- nostro
- produzione
- al di fuori
- parametri
- appassionato
- paziente
- pazienti
- modelli
- perfetta
- performance
- conduttura
- Platone
- Platone Data Intelligence
- PlatoneDati
- per favore
- Post
- potenzialmente
- predire
- previsto
- previsione
- predizione
- Previsioni
- Preparare
- in precedenza
- processi
- i processi
- Proteine
- Proteine
- fornire
- fornisce
- fornitura
- Python
- pytorch
- Qi
- rapidamente
- rapidamente
- campo in rapida evoluzione
- tempo reale
- raccomandare
- Rosso
- regione
- relazionato
- fare affidamento
- rappresentare
- che rappresenta
- rappresenta
- richiedere
- riparazioni
- ricercatori
- colpevole
- RIGA
- Correre
- running
- sagemaker
- Inferenza di SageMaker
- stesso
- Risparmi
- scalabile
- scala
- Scienze
- SCIENZE
- Punto
- punteggio
- script
- secondo
- sicuro
- vedere
- anziano
- Sequenza
- Servizi
- set
- alcuni
- Condividi
- lei
- dovrebbero
- mostrare attraverso le sue creazioni
- mostrato
- lato
- simile
- semplificare
- singolo
- soluzione
- Soluzioni
- RISOLVERE
- alcuni
- Spaziale
- specialista
- Spettroscopia
- velocità
- inizia a
- state-of-the-art
- step
- strutturale
- La struttura
- studio
- Studio
- tale
- adatto
- supporto
- sopravvivenza
- Fai
- prende
- obiettivi
- task
- team
- le squadre
- Consulenza
- test
- di
- che
- Il
- loro
- Li
- poi
- Là.
- Strumenti Bowman per analizzare le seguenti finiture:
- questo
- tre
- tridimensionale
- tempo
- richiede tempo
- volte
- a
- tokenize
- token
- strumenti
- torcia
- allenato
- Training
- trasformatori
- trasporto
- trattamento
- seconda
- Digitare
- Tipi di
- capire
- e una comprensione reciproca
- Università
- a differenza di
- us
- uso
- utilizzato
- utilizzando
- CONVALIDARE
- molto
- Visualizza
- visualizzazione
- W
- ricercato
- Modo..
- we
- sito web
- servizi web
- noto
- Che
- wikipedia
- volere
- con
- WordPress
- Lavora
- lavori
- sarebbe
- raggi X
- Tu
- Trasferimento da aeroporto a Sharm
- zefiro