La trascrittomica spaziale e la proteomica forniscono informazioni complementari che hanno trasformato la nostra comprensione di processi biologici complessi. Tuttavia, l’integrazione sperimentale di queste modalità è limitata.
Un nuovo metodo chiamato Spatial PrOtein and Transcriptome Sequencing (SPOTS) può illuminare le identità e le attività delle cellule in un organo o in un tumore con una risoluzione senza precedenti. Sviluppato dai ricercatori di Weill Cornell Medicine, NewYork-Presbyterian e il New York Genome Center, SPOTS può illuminare le identità e le attività delle cellule in un organo o in un tumore con una risoluzione senza precedenti.
La tecnica conserva le informazioni sulle posizioni precise delle cellule mentre registra i modelli di attività genetica e la presenza di elementi essenziali proteine nelle cellule attraverso campioni di tessuto. Ciò rende possibile creare “mappe” di organi complesse e ricche di dati, compresi organi malati e tumori, che possono essere estremamente utili sia nella ricerca di base che in quella clinica.
Il co-autore senior dello studio, il dottor Dan Landau, professore associato di medicina presso la Divisione di Ematologia e Oncologia medica e membro del Sandra and Edward Meyer Cancer Center presso Weill Cornell Medicine e membro principale della facoltà presso il New York Genome Center, disse, “Questa tecnologia è entusiasmante perché ci consente di mappare l’organizzazione spaziale dei tessuti, compresi i tipi di cellule, le attività cellulari e le interazioni cellula-cellula, come mai prima d’ora”.
Il nuovo approccio fa parte di un’iniziativa più ampia portata avanti da scienziati e ingegneri per creare mezzi più efficaci per “vedere” a livello microscopico come funzionano organi e tessuti. Gli ultimi anni hanno visto progressi significativi nella ricerca, in particolare nei metodi per delineare l’attività genetica e altri strati di dati in singole cellule o piccoli gruppi di cellule. cellule. Tuttavia, è necessario recuperare le informazioni riguardanti il posizionamento originale delle cellule profilate all'interno dei tessuti, poiché questi approcci richiedono spesso la rottura dei tessuti e la separazione delle cellule dalle cellule vicine. La nuova tecnica registra anche i dati spaziali e lo fa con una risoluzione eccellente.
Il metodo si basa in parte sulla tecnologia 10x Genomics esistente. Utilizza vetrini adatti per l'imaging di campioni di tessuto con i normali metodi patologici basati sul microscopio, ma è anche rivestito con migliaia di particolari molecole sonda.
Il “codice a barre” chimico di ciascuna molecola della sonda ne identifica la posizione bidimensionale sul vetrino. Le molecole della sonda sul vetrino afferrano il RNA messaggeri (mRNA), essenzialmente le trascrizioni dei geni attivati, dalle cellule vicine: quando un campione di tessuto tagliato a fettine sottili viene posizionato sul vetrino, le sue cellule vengono rese permeabili. Nella procedura vengono utilizzati anticorpi designer che si attaccano alle molecole sonda e alle proteine uniche di interesse nel tessuto.
I ricercatori possono identificare in modo rapido e automatico gli mRNA raccolti e le proteine scelte e mapparli con precisione nelle loro posizioni originali nel campione di tessuto. Le mappe prodotte possono essere considerate indipendentemente o in confronto all'imaging patologico di routine del campione.
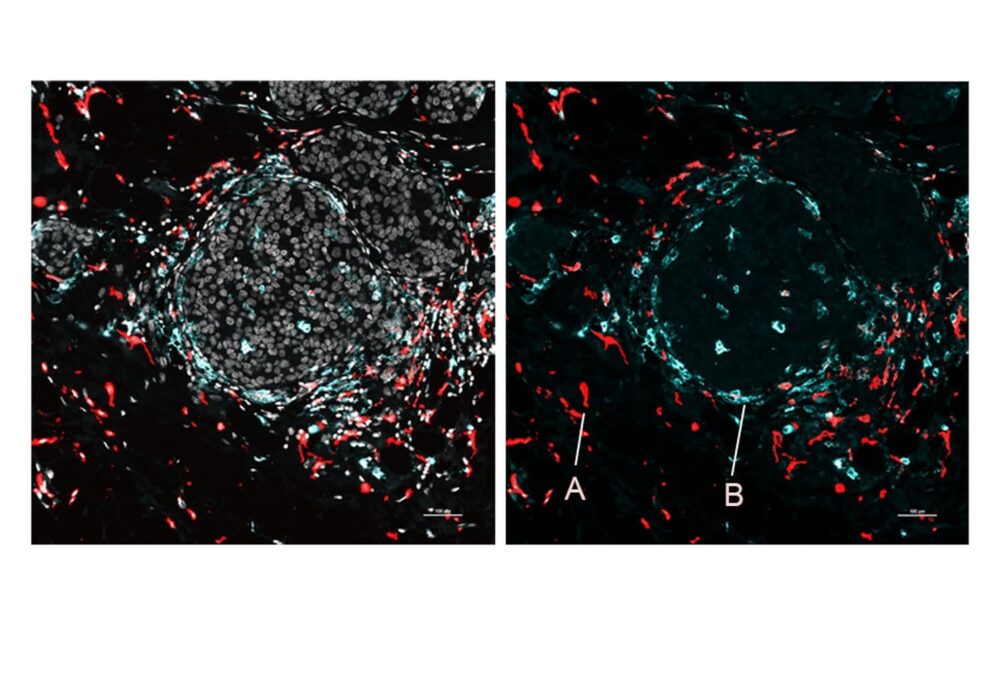
Sul tessuto di una milza di topo sana, il team ha utilizzato SPOTS per mostrare l'intricata architettura funzionale di questo organo, inclusi gruppi di vari tipi di cellule, i loro stati funzionali e come tali stati si alteravano a seconda del posizionamento delle cellule.
I ricercatori hanno anche utilizzato SPOTS per mappare la struttura cellulare di un tumore al seno di topo, evidenziandone il potenziale di utilizzo ricerca sul cancro. La mappa generata mostrava macrofagi e cellule immunitarie in due stati diversi, ciascuno indicato da un diverso marcatore proteico: una condizione era attiva e combatteva i tumori, mentre l’altra era immunosoppressiva e stabiliva una barriera per proteggere il tumore.
Il dottor Landau, oncologo del NewYork-Presbyterian/Weill Cornell Medical Center, disse, "Abbiamo potuto vedere che questi due sottoinsiemi di macrofagi si trovano in aree diverse del tumore e interagiscono con cellule diverse, e che la differenza nel microambiente probabilmente guida i loro distinti stati di attività."
“Tali dettagli dell’ambiente immunitario del tumore – dettagli che spesso non possono essere risolti a causa della scarsità di cellule immunitarie all’interno dei tumori – potrebbero aiutare a spiegare perché alcuni pazienti rispondono terapia di potenziamento immunitario e altri no, e quindi potrebbero informare la progettazione di future immunoterapie”.
“Questa versione iniziale di SPOTS ha una risoluzione spaziale tale che ogni “pixel” del set di dati risultante somma le informazioni sull’attività genetica per almeno diverse cellule. Tuttavia, i ricercatori sperano presto di restringere questa risoluzione alle singole cellule aggiungendo altri strati di informazioni cellulari chiave”.
Riferimento della Gazzetta:
- Ben-Chetrit, N., Niu, X., Swett, AD et al. Integrazione del profilo spaziale dell'intero trascrittoma con marcatori proteici. Nat Biotecnologie (2023). DOI: 10.1038/s41587-022-01536-3