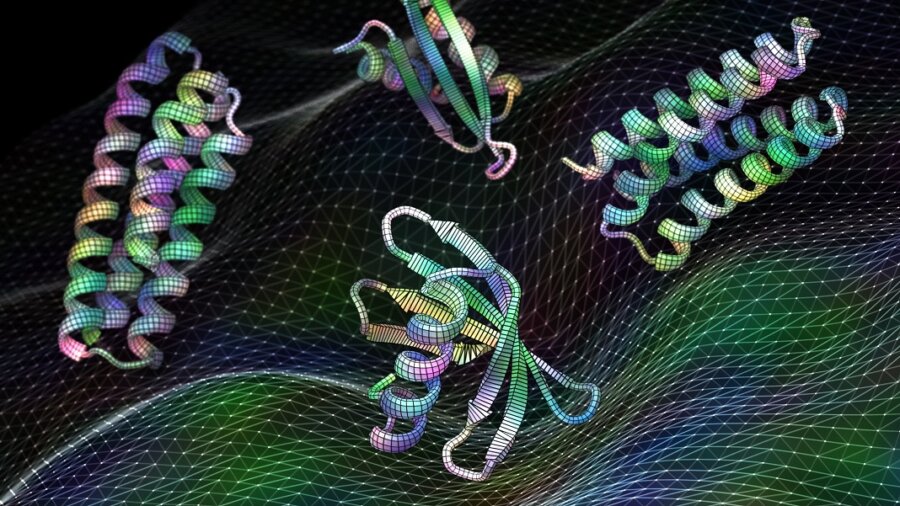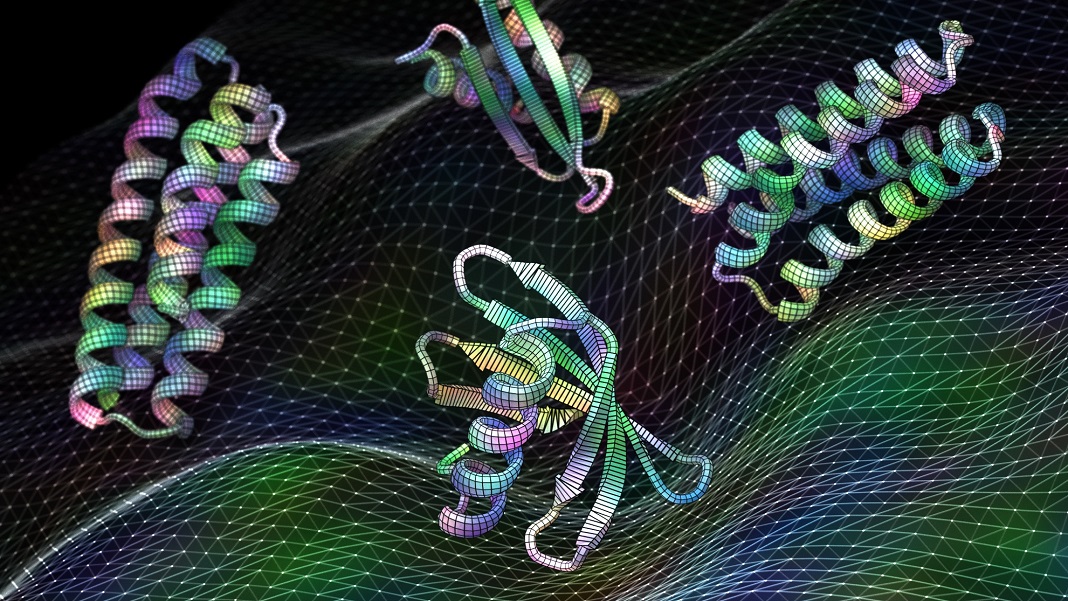
Å designe et protein er litt som å lage et skap. Det første trinnet er å bygge ryggraden som holder proteinet sammen. Men så kommer den vanskelige delen: å finne ut hvor du skal installere hengsler på stillaset – det vil si å finne de beste "hotspots" – for å sette på dører, hyller og andre tilbehør som til slutt gjør skapet fullt funksjonelt.
På en måte har proteiner også hotspots innebygd i strukturene deres. Tro mot navnet sitt, «funksjonelle nettsteder», danner disse spennende kriker og kroker intrikate dokker for andre proteiner eller medisiner å gripe tak i. Lokalitetene er sentrale for å utføre de fleste av våre grunnleggende biologiske prosesser. De er også en enorm gullgruve for å designe nye behandlinger og medisinske medisiner.
Problemet? Funksjonelle nettsteder er vanskelige å kartlegge. Forskere måtte tradisjonelt mutere mistenkte områder på et protein én etter én – bytte en aminosyre til en annen – for å finne nøyaktige bindingspunkter. Som en detektiv som screener hundrevis av mistenkte, som det kan være mange av, er det ekstremt kjedelig.
A ny studie in Vitenskap styrtet hele spillboken. Ledet av Dr. David Baker ved University of Washington, tok et team inn i en AIs "fantasi" for å finne opp en myriade av funksjonelle nettsteder fra bunnen av. Det er et maskinsinns "kreativitet" på sitt beste - en dyp læringsalgoritme som forutsier det generelle området til et proteins funksjonelle sted, men som deretter former strukturen ytterligere.
Som en realitetssjekk brukte teamet den nye programvaren til å generere medisiner som bekjemper kreft og designer vaksiner mot vanlige, men noen ganger dødelige, virus. I ett tilfelle kom det digitale sinnet opp med en løsning som, når den ble testet i isolerte celler, passet perfekt til et eksisterende antistoff mot et vanlig virus. Algoritmen "forestilte seg" med andre ord et hotspot fra et viralt protein, noe som gjorde det sårbart som et mål for å designe nye behandlinger.
Algoritmen er dyplærings første forsøk på å bygge proteiner rundt funksjonene deres, og åpner en dør til behandlinger som tidligere var utenkelige. Men programvaren er ikke begrenset til naturlige protein-hotspots. "Proteinene vi finner i naturen er fantastiske molekyler, men designet proteiner kan gjøre så mye mer," sa Baker i en pressemelding. Algoritmen er "å gjøre ting som ingen av oss trodde den ville være i stand til."
Protein Hotspot
Bakers team er ikke fremmede for å forutsi proteiner med kunstige sinn. For noen år tilbake rystet de det strukturelle biologifeltet ved å gi ut Rosetta, en programvare som kan forutsi et proteins 3D-struktur basert på dets aminosyresekvens alene. De kartla ytterligere proteinkomplekser og designet protein-"skrutrekkere" fra bunnen av for å lirke fra hverandre uønskede proteininteraksjoner. Sent i fjor ga de ut en dypt læringsnettverk kalt trRosetta, en AI "arkitekt" som generaliserer hvordan strenger av aminosyrer ordner seg i intrikate strukturer på nanoskala.
La oss sikkerhetskopiere.
Det er lett å forestille seg proteiner som den kjøttfulle, senete kyllingvingen jeg biter i mens jeg skriver denne setningen. Men på det molekylære nivået er de langt mer elegante. Se for deg flere legoklosser – aminosyrer – holdt sammen av en snor. Snurr nå den rundt, vri kjeden til noen blokker klikker på hverandre. Dette danner en delikat struktur som ofte minner om en helix eller krøllete sengetøy. I noen proteiner samles disse byggesteinene videre til komplekser - for eksempel ved å lage en kanal som går gjennom en celles beskyttende membran som en patruljert mellomstatlig motorvei.
Proteiner driver hver eneste biologiske prosess, ofte gjennom en kaskade av interaksjoner med andre proteiner eller medikamenter, som – avhengig av partneren – kan utløse helt andre konsekvenser: skal en celle leve eller dø? Angripe en potensiell inntrenger eller stå ned? Proteiner er med andre ord livets byggesteiner, og å analysere strukturen deres er hvordan vi kan hacke oss inn i livet.
Her er tingen: ikke alle deler av et protein er skapt like. Hvis et protein er en menneskekropp, er funksjonelle steder dets "hender" - der det griper tak i et annet protein eller medikament, vekker enzymatiske reaksjoner eller bekjemper invaderende patogener. Innebygd direkte i proteinets struktur, er disse nettstedene vanskelige å feste og enda vanskeligere å gjenskape.
Den nye studien taklet problemet med en versjon av Rosetta: med litt tidligere kunnskap, er det mulig for en datamaskin å drømme opp en kjede av aminosyrer som naturlig foldes til et funksjonelt sted?
Drømmeren og realisten
Problemet kan virke eksotisk, men det er et tidligere eksempel - i et annet felt. Ved å bruke et nevralt nettverk skapte OpenAI et bredt spekter av bilder fra teksttekster alene. En spinoff av rockstar AI-tekstgeneratoren GPT-3, genererte DALL·E-algoritmen fantastiske, men realistiske bilder basert på enkle tekstmeldinger ved å oppdage mønstre fra treningen. "Den tar de dypeste, mørkeste fordypningene i fantasien din og gjør den til noe som er uhyggelig relevant," sa Dr. Hany Farid ved UC Berkeley etter verktøyets første utgivelse.
Å bygge et proteinfunksjonelt nettsted er lignende. Her er aminosyrer bokstavene og proteinfunksjonsstedet er bildet. "Ideen er den samme: nevrale nettverk kan trenes til å se mønstre i data. Når du er trent, kan du gi den en oppfordring og se om den kan generere en elegant løsning,” sa Dr. Joseph Watson, hovedforfatter av det nye verket. Bortsett fra i stedet for å skrive en roman, kan algoritmen bidra til å omskrive livet.
Teamet startet med en tidligere kreasjon, trRosetta. Det er et nevralt nettverk som opprinnelig er designet for å tenke opp nye proteiner basert på aminosyresekvenser, samtidig som de er i stand til å forutsi strukturen deres – noen er så fremmede fra naturlige at teamet kalte dyplæringens indre virkemåte for «hallusinasjon». Algoritmen virket perfekt: den kunne både forutsi et proteins aminosyresekvens og dets struktur.
Hikken? Det fungerte egentlig ikke. I motsetning, OG for prediksjon av proteinstruktur, RoseTTAFold, presterte som en mester. Algoritmens kraft kommer fra designen: modellering av hver aminosyre på nanoskala, og gir koordinater til hvert atom. Som å feste et geografisk nettsted ved hjelp av Google Maps, gir dette et nivå av sannhet for en struktur som en AI kan riffe videre på – en slags «begrenset hallusinasjon».
Oversettelse? RoseTTAFold kan forutsi en funksjonell struktur – spesifikk for det aktuelle problemet – og komme opp med en grov skisse som det endelige designet.
Så kom et annet smart triks, kalt «inpainting». Her gjemte teamet deler av proteinsekvensen eller strukturen. Programvaren måtte lære å tyde informasjon fra det som egentlig er en støyende radioavlytting, der du bare kan høre de første par ordene, men prøve å forstå betydningen ved å fylle ut de tomme feltene. RoseTTAFold taklet "problemet med gjenoppretting av manglende informasjon" med velbehag, og autofullførte både aminosyresekvenser og strukturer for å konstruere en gitt funksjonell region med høy kvalitet.
RoseTTAFold kan takle problemene med å bygge aminosyresekvenser og samtidig generere en ryggrad for stedet. Det er som å sette ord på papir: forfatteren sørger for at hver bokstav er på rett plass, samtidig som han sjekker at grammatikken og meningen gir mening.
Stille spørsmål ved virkelighetens natur
Etter å ha satt sin nye kreasjon på prøve, genererte teamet flere medikament- og vaksinedesign som potensielt kunne bekjempe virus og kreft eller hjelpe med helseproblemer med lavt jernnivå.
For hovedforfatteren Dr. Jue Wang ble algoritmen uventet relevant. Mens han jobbet med prosjektet, ble hans to år gamle sønn innlagt på akuttmottaket på grunn av en lungeinfeksjon av RSV (Respiratory Syncytial Virus) – et virus som normalt viser forkjølelseslignende symptomer, men som kan være dødelig hos unge og barn. eldre.
På det tidspunktet brukte Wang algoritmen til å designe nye behandlinger, som inkluderte potensielle nettsteder på RSV for ytterligere å teste vaksiner og medisiner mot. Det er en relativt godt kartlagt struktur. Programvaren hallusinerte design som rekapitulerte to steder for vaksiner å potensielt binde seg til. Tester med hallusinerte proteiner, rekonstruert i bakterier, fanget raskt på eksisterende antistoffer – et tegn på at de er funksjonelle og at dyplæringstilnærmingen fungerer.
Hendelsen "fikk meg til å innse at til og med "testproblemene" vi jobbet med faktisk var ganske meningsfulle, sa Wang.
I flere tilleggstester designet teamet funksjonelle steder for et enzym, proteinbindende proteiner og proteiner som griper inn i metallioner - i utgangspunktet hvordan du absorberer jern og andre viktige metaller.
Selv om det er kraftig, er det rom for vekst. Metoden åpner for å avmystifisere naturlige proteiner, men også potensielt designe nye for syntetisk biologi. "Dette er veldig kraftige nye tilnærminger, men det er fortsatt mye rom for forbedring," sa Baker.
Alt i alt er det nok en seier for dyp læring og et spennende utstillingsvindu for hvordan AI og biologi kan synergi. "Dyp læring transformert proteinstrukturprediksjon de siste to årene, vi er nå midt i en lignende transformasjon av proteindesign," sa Baker.
Bildekreditt: Ian C. Haydon/UW Institute for Protein Design. Ny kunstig intelligens-programvare trent på proteinstrukturer kan generere funksjonelle proteiner, inkludert disse kandidatvaksinene for respiratorisk virus RSV, på sekunder.