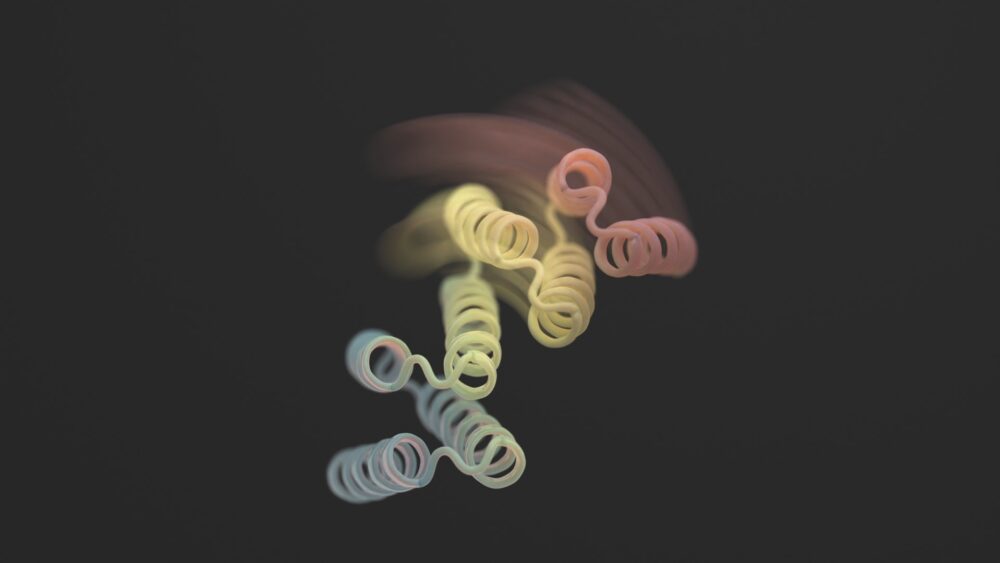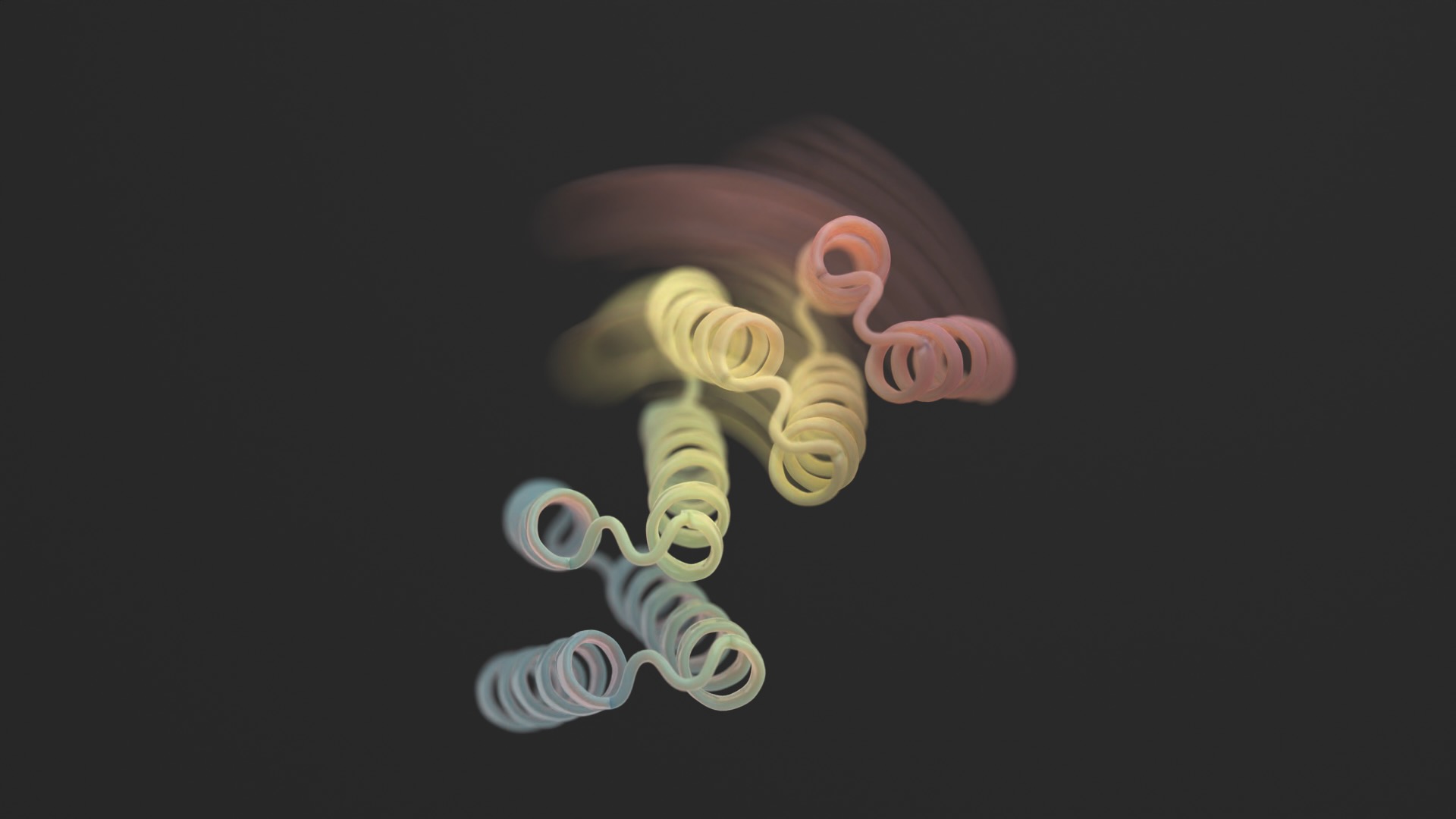
Vi tenker ofte på proteiner som uforanderlige 3D-skulpturer.
Det er ikke helt riktig. Mange proteiner er transformatorer som vrir og endrer form avhengig av biologiske behov. Én konfigurasjon kan forplante skadelige signaler fra hjerneslag eller hjerteinfarkt. En annen kan blokkere den resulterende molekylære kaskaden og begrense skade.
På en måte fungerer proteiner som biologiske transistorer - på-av-brytere ved roten av kroppens molekylære "datamaskin" som bestemmer hvordan den reagerer på eksterne og indre krefter og tilbakemeldinger. Forskere har lenge studert disse formskiftende proteinene for å tyde hvordan kroppen vår fungerer.
Men hvorfor stole på naturen alene? Kan vi lage biologiske "transistorer", ukjente for det biologiske universet, fra bunnen av?
Skriv inn AI. Flere dyplæringsmetoder kan allerede nøyaktig forutsi proteinstrukturer—et gjennombrudd et halvt århundre underveis. Etterfølgende studier som bruker stadig kraftigere algoritmer, har hallusinert proteinstrukturer som ikke er bundet av evolusjonskreftene.
Likevel har disse AI-genererte strukturene en undergang: selv om de er svært intrikate, er de fleste helt statiske – i hovedsak en slags digital proteinskulptur frosset i tid.
En ny studie in Vitenskap denne måneden brøt formen ved å legge til fleksibilitet til designerproteiner. De nye strukturene er ikke kontortionister uten grenser. Imidlertid kan designerproteinene stabilisere seg i to forskjellige former - tenk et hengsel i enten en åpen eller lukket konfigurasjon - avhengig av en ekstern biologisk "lås". Hver tilstand er analog med en datamaskins "0" eller "1", som deretter kontrollerer cellens utgang.
"Før kunne vi bare lage proteiner som hadde en stabil konfigurasjon," sa studieforfatter Dr. Florian Praetorius ved University of Washington. "Nå kan vi endelig lage proteiner som beveger seg, noe som bør åpne for et ekstraordinært utvalg av bruksområder."
Hovedforfatter Dr. David Baker har ideer: "Fra å danne nanostrukturer som reagerer på kjemikalier i miljøet til applikasjoner innen medikamentlevering, begynner vi akkurat å utnytte potensialet deres."
Et protein-ekteskap laget i AI
En rask bit av biologi 101.
Proteiner bygger og driver kroppen vår. Disse makromolekylene begynner sin reise fra DNA. Genetisk informasjon blir oversatt til aminosyrer, byggesteinene til et protein – bildeperler på en snor. Hver streng brettes deretter til intrikate 3D-former, med noen deler som fester seg til andre. Kalt sekundære strukturer, noen konfigurasjoner ser ut som Twizzlers. Andre vever til teppelignende ark. Disse formene bygger videre på hverandre og danner svært sofistikerte proteinarkitekturer.
Ved å forstå hvordan proteiner får sine former, kan vi potensielt konstruere nye fra bunnen av, utvide det biologiske universet og skape nye våpen mot virusinfeksjoner og andre sykdommer.
Tilbake i 2020 brøt DeepMinds AlphaFold og David Baker labs RoseTTAFold det strukturelle biologiske internett ved nøyaktig å forutsi proteinstrukturer basert på deres aminosyresekvenser alene.
Siden den gang har AI-modellene spådd formen til nesten alle proteiner kjent – og ukjent – for vitenskapen. Disse kraftige verktøyene omformer allerede biologisk forskning, og hjelper forskere raskt å finne potensielle mål bekjempe antibiotikaresistens, studere "hus" av vårt DNA, utvikle nye vaksiner eller til og med kaste lys over sykdommer som herjer hjernen, som Parkinsons sykdom.
Så kom en bombe: generative AI-modeller, som DALL-E og ChatGPT, ga et fristende prospekt. I stedet for bare å forutsi proteinstrukturer, hvorfor ikke har AI drømt opp helt ny proteinstrukturer i stedet? Fra et protein som binder hormoner for å regulere kalsiumnivået til kunstige enzymer som katalyserer bioluminescens, utløste de første resultatene entusiasme og potensialet for AI-designede proteiner virket uendelig.
Ved roret for disse oppdagelsene er Bakers laboratorium. Kort tid etter utgivelsen av RoseTTAFold videreutviklet de algoritmen for å finne funksjonelle steder på et protein – der det interagerer med andre proteiner, medikamenter eller antistoffer – og banet vei for forskere å drømme opp nye medisiner de ennå ikke har sett for seg.
Men én ting manglet: fleksibilitet. Et stort antall proteiner "kodeskifter" i form for å endre deres biologiske budskap. Resultatet kan bokstavelig talt være liv eller død: et protein kalt Bax, for eksempel, endrer sin form til en konformasjon som utløser celledød. Amyloid beta, et protein involvert i Alzheimers sykdom, tar notorisk en annen form ettersom det skader hjerneceller.
En kunstig intelligens som hallusinerer lignende flip-flop-proteiner kan bringe oss nærmere å forstå og rekapitulere disse biologiske gåtene – noe som fører til nye medisinske løsninger.
Hinge, Line og Sinker
Å designe ett protein på atomnivå - og håpe det fungerer i en levende celle - er vanskelig. Å designe en med to konfigurasjoner er et mareritt.
Som en løs analogi, tenk på iskrystaller i en sky som til slutt formes til snøflak, hver av dem forskjellige i struktur. AIs jobb er å lage proteiner som kan skifte mellom to forskjellige "snøfnugg" ved å bruke de samme aminosyre-"iskrystallene", med hver tilstand som tilsvarer en "på" eller "av"-bryter. I tillegg må proteinet spille fint inne i levende celler.
Laget begynte med flere regler. For det første bør hver struktur se veldig forskjellig ut mellom de to statene - som en menneskelig profil som står eller sitter. De kunne sjekke dette ved å måle avstander mellom atomer, forklarte teamet. For det andre må endringen skje raskt. Dette betyr at proteinet ikke kan utfolde seg helt før det settes sammen igjen til en annen form, noe som tar tid.
Så er det noen retningslinjer for begrunnelse for et funksjonelt protein: det må leke godt med kroppsvæsker i begge stater. Til slutt må den fungere som en bryter, og endre formen avhengig av innganger og utganger.
Å møte alle "disse egenskapene i ett proteinsystem er utfordrende," sa teamet.
Ved å bruke en blanding av AlphaFold, Rosetta og proteinMPNN, ser det endelige designet ut som et hengsel. Den har to stive deler som kan bevege seg i forhold til hverandre, mens en annen del forblir brettet. Normalt er proteinet lukket. Utløseren er et lite peptid - en kort kjede av aminosyrer - som binder seg til hengslene og utløser formendringen. Disse såkalte "effektorpeptidene" ble nøye designet for spesifisitet, og reduserte sjansene deres for å gripe tak i deler utenfor målet.
Teamet la først glød-i-mørke-utløserpeptider til flere hengseldesigner. Etterfølgende analyse fant at avtrekkeren lett tok tak i hengslet. Proteinets konfigurasjon endret seg. Som en fornuftssjekk var formen en tidligere forutsagt ved hjelp av AI-analyse.
Ytterligere studier ved bruk av krystalliserte strukturer av proteindesignene, enten med eller uten effektoren, validerte resultatene ytterligere. Disse testene jaktet også på designprinsipper som fikk hengslene til å fungere, og parametere som tipper den ene tilstanden til den andre.
Take away? AI kan nå designe proteiner med to forskjellige tilstander – i hovedsak bygge biologiske transistorer for syntetisk biologi. Foreløpig bruker systemet kun spesialdesignede effektorpeptider i sine studier, noe som kan begrense forskning og klinisk potensial. Men ifølge teamet kan strategien også strekke seg til naturlige peptider, for eksempel de som binder proteiner som er involvert i regulering av blodsukker, regulerer vann i vev eller påvirker hjerneaktivitet.
"Som transistorer i elektroniske kretser, kan vi koble bryterne til eksterne utganger og innganger for å lage sanseenheter og inkorporere dem i større proteinsystemer," sa teamet.
Studieforfatter Dr. Philip Leung legger til: "Dette kan revolusjonere bioteknologi på samme måte som transistorer transformerte elektronikk."
Bildekreditt: Ian C Haydon/UW Institute for Protein Design
- SEO-drevet innhold og PR-distribusjon. Bli forsterket i dag.
- PlatoData.Network Vertical Generative Ai. Styrk deg selv. Tilgang her.
- PlatoAiStream. Web3 Intelligence. Kunnskap forsterket. Tilgang her.
- PlatoESG. Bil / elbiler, Karbon, CleanTech, Energi, Miljø, Solenergi, Avfallshåndtering. Tilgang her.
- PlatoHelse. Bioteknologisk og klinisk etterretning. Tilgang her.
- ChartPrime. Hev handelsspillet ditt med ChartPrime. Tilgang her.
- BlockOffsets. Modernisering av eierskap for miljøkompensasjon. Tilgang her.
- kilde: https://singularityhub.com/2023/08/22/ai-can-now-design-proteins-that-behave-like-biological-transistors/
- : har
- :er
- :ikke
- $OPP
- 2020
- 3d
- a
- AC
- Ifølge
- nøyaktig
- Handling
- aktivitet
- la til
- legge
- I tillegg
- Legger
- Etter
- mot
- AI
- AI-modeller
- algoritme
- algoritmer
- Alle
- alene
- allerede
- også
- Selv
- Alzheimers
- an
- analyse
- og
- En annen
- søknader
- ER
- AS
- At
- angripe
- forfatter
- borte
- tilbake
- baker
- basert
- BE
- før du
- begynte
- begynne
- beta
- mellom
- binde
- biologi
- bioteknologi
- Bit
- Blokker
- Blocks
- blod
- organer
- både
- Brain
- Hjerneaktivitet
- Hjerneceller
- gjennombrudd
- Broke
- bygge
- Bygning
- men
- by
- Kalsium
- som heter
- kom
- CAN
- nøye
- cascade
- katalyserer
- Celler
- Århundre
- kjede
- utfordrende
- sjansene
- endring
- endret
- endring
- ChatGPT
- sjekk
- Klinisk
- stengt
- nærmere
- Cloud
- helt
- Konfigurasjon
- kontroller
- Tilsvarende
- kunne
- Par
- skape
- Opprette
- kreditt
- dall sin
- skade
- David
- Død
- dechiffrere
- dyp
- dyp læring
- DeepMind
- levering
- avhengig
- utforming
- designprinsipper
- designet
- designer
- utforme
- design
- bestemme
- utviklet
- Enheter
- forskjellig
- digitalt
- sykdom
- sykdommer
- dna
- ned
- fall
- dr
- drøm
- medikament
- Narkotika
- hver enkelt
- lett
- Edge
- enten
- elektronisk
- Elektronikk
- Endless
- ingeniør
- entusiasme
- Miljø
- Selv
- etter hvert
- Hver
- evolusjon
- eksempel
- ekspanderende
- forklarte
- utvide
- utvendig
- ekstraordinær
- FAST
- tilbakemelding
- slutt~~POS=TRUNC
- Endelig
- Først
- fleksibilitet
- Til
- Krefter
- skjema
- funnet
- fra
- frossen
- funksjon
- funksjonelle
- videre
- Gevinst
- generative
- Generativ AI
- retningslinjer
- HAD
- Halvparten
- skje
- Hard
- skade
- skader
- Ha
- Hjerte
- Heart Attack
- hjelpe
- svært
- Hengsel
- håper
- Hvordan
- Men
- HTTPS
- menneskelig
- ICE
- Ideer
- trodd
- uforanderlige
- in
- innlemme
- stadig
- infeksjoner
- påvirke
- informasjon
- innledende
- innganger
- innsiden
- i stedet
- Institute
- interaktiv
- intern
- Internet
- inn
- involvert
- IT
- DET ER
- selv
- Jobb
- reise
- bare
- lab
- stor
- større
- læring
- nivåer
- Life
- lett
- i likhet med
- BEGRENSE
- grenser
- linje
- levende
- Lang
- Se
- ser ut som
- UTSEENDE
- senking
- laget
- gjøre
- mange
- Kan..
- midler
- måling
- medisinsk
- melding
- metoder
- mangler
- bland
- modeller
- molekyl~~POS=TRUNC
- Måned
- mest
- flytte
- flere
- Naturlig
- Natur
- behov
- Ny
- fint
- NIH
- normalt
- nå
- Antall
- of
- tilbudt
- ofte
- on
- ONE
- seg
- bare
- videre til
- åpen
- or
- Annen
- andre
- vår
- produksjon
- parametere
- deler
- brikke
- plato
- Platon Data Intelligence
- PlatonData
- Spille
- potensiell
- potensielt
- kraftig
- forutsi
- spådd
- forutsi
- tidligere
- prinsipper
- Profil
- egenskaper
- prospektet
- Protein
- Proteiner
- Rask
- raskt
- område
- heller
- reagerer
- Regulere
- regulerende
- slektning
- frigjør
- avhengige
- forblir
- forskning
- Svare
- resultere
- resulterende
- Resultater
- Revolusjonere
- ikke sant
- rigid
- root
- regler
- Kjør
- Sa
- samme
- Vitenskap
- forskere
- skraper
- Sekund
- sekundær
- syntes
- flere
- Form
- figurer
- skur
- skift
- Kort
- Om kort tid
- bør
- signaler
- lignende
- ganske enkelt
- Nettsteder
- Sittende
- liten
- snøflak
- Solutions
- noen
- sofistikert
- gnist
- spesifisitet
- stabilisere
- stabil
- Start
- Tilstand
- Stater
- stikker
- Strategi
- String
- strukturell
- struktur
- studert
- studier
- Studer
- senere
- I ettertid
- slik
- sukker
- Bytte om
- syntetisk
- system
- Systemer
- Ta
- tar
- Pek
- mål
- lag
- tester
- enn
- Det
- De
- deres
- Dem
- deretter
- Der.
- Disse
- de
- ting
- tror
- denne
- De
- tid
- typen
- til
- sammen
- verktøy
- forvandlet
- transformers
- utløse
- vri
- to
- forståelse
- Universe
- universitet
- ukjent
- us
- bruker
- ved hjelp av
- validert
- vesentlig
- viral
- var
- washington
- Vann
- Vei..
- we
- Våpen
- Weave
- web
- var
- hvilken
- mens
- hvorfor
- med
- uten
- Arbeid
- virker
- ennå
- zephyrnet