Proteiner driver mange biologiske prosesser, som enzymaktivitet, molekylær transport og cellulær støtte. Den tredimensjonale strukturen til et protein gir innsikt i dets funksjon og hvordan det interagerer med andre biomolekyler. Eksperimentelle metoder for å bestemme proteinstruktur, som røntgenkrystallografi og NMR-spektroskopi, er kostbare og tidkrevende.
I motsetning til dette kan nylig utviklede beregningsmetoder raskt og nøyaktig forutsi strukturen til et protein fra dets aminosyresekvens. Disse metodene er kritiske for proteiner som er vanskelige å studere eksperimentelt, for eksempel membranproteiner, målene for mange medikamenter. Et kjent eksempel på dette er AlphaFold, en dyp læringsbasert algoritme kjent for sine nøyaktige spådommer.
ESMFold er en annen svært nøyaktig, dyp læringsbasert metode utviklet for å forutsi proteinstruktur fra aminosyresekvensen. ESMFold bruker en stor proteinspråkmodell (pLM) som ryggrad og opererer ende til ende. I motsetning til AlphaFold2, trenger den ikke et oppslag eller Multiple Sequence Alignment (MSA) trinn, og er heller ikke avhengig av eksterne databaser for å generere spådommer. I stedet trente utviklingsteamet modellen på millioner av proteinsekvenser fra UniRef. Under trening utviklet modellen oppmerksomhetsmønstre som elegant representerer de evolusjonære interaksjonene mellom aminosyrer i sekvensen. Denne bruken av en pLM i stedet for en MSA muliggjør opptil 60 ganger raskere prediksjonstider enn andre toppmoderne modeller.
I dette innlegget bruker vi den ferdigtrente ESMFold-modellen fra Hugging Face with Amazon SageMaker å forutsi tungkjedestrukturen til trastuzumaben monoklonalt antistoff først utviklet av Genentech for behandling av HER2-positiv brystkreft. Å raskt forutsi strukturen til dette proteinet kan være nyttig hvis forskere ønsket å teste effekten av sekvensmodifikasjoner. Dette kan potensielt føre til forbedret pasientoverlevelse eller færre bivirkninger.
Dette innlegget gir et eksempel på en Jupyter-notatbok og relaterte skript i det følgende GitHub repository.
Forutsetninger
Vi anbefaler å kjøre dette eksemplet i en Amazon SageMaker Studio bærbare kjører PyTorch 1.13 Python 3.9 CPU-optimalisert bilde på en ml.r5.xlarge-forekomsttype.
Visualiser den eksperimentelle strukturen til trastuzumab
Til å begynne med bruker vi biopython bibliotek og et hjelpeskript for å laste ned trastuzumab-strukturen fra RCSB Protein Data Bank:
Deretter bruker vi py3Dmol bibliotek for å visualisere strukturen som en interaktiv 3D-visualisering:
Følgende figur representerer 3D-proteinstrukturen 1N8Z fra Protein Data Bank (PDB). I dette bildet vises den lette kjeden av trastuzumab i oransje, den tunge kjeden er blå (med den variable regionen i lyseblått), og HER2-antigenet er grønt.
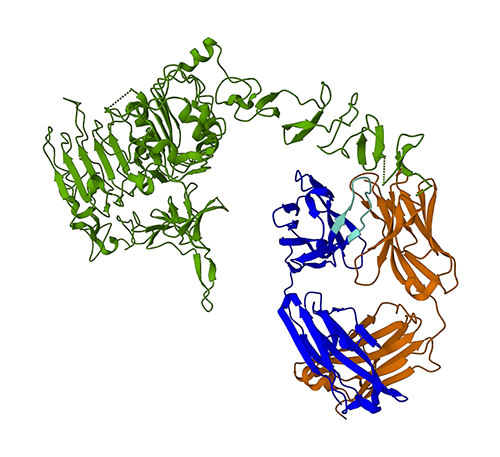
Vi vil først bruke ESMFold til å forutsi strukturen til den tunge kjeden (kjede B) fra dens aminosyresekvens. Deretter vil vi sammenligne prediksjonen med den eksperimentelt bestemte strukturen vist ovenfor.
Forutsi trastuzumab tungkjedestruktur fra dens sekvens ved å bruke ESMFold
La oss bruke ESMFold-modellen til å forutsi strukturen til den tunge kjeden og sammenligne den med det eksperimentelle resultatet. Til å begynne med bruker vi et forhåndsbygd notatbokmiljø i Studio som kommer med flere viktige biblioteker, som PyTorch, forhåndsinstallert. Selv om vi kunne bruke en akselerert forekomsttype for å forbedre ytelsen til den bærbare analysen vår, bruker vi i stedet en ikke-akselerert forekomst og kjører ESMFold-prediksjonen på en CPU.
Først laster vi den ferdigtrente ESMFold-modellen og tokenizeren fra Klemmer Face Hub:
Deretter kopierer vi modellen til enheten vår (CPU i dette tilfellet) og setter noen modellparametere:
For å forberede proteinsekvensen for analyse, må vi tokenisere den. Dette oversetter aminosyresymbolene (EVQLV...) til et numerisk format som ESMFold-modellen kan forstå (6,19,5,10,19,...):
Deretter kopierer vi den tokeniserte inngangen til modusen, lager en prediksjon og lagrer resultatet i en fil:
Dette tar omtrent 3 minutter på en ikke-akselerert forekomsttype, som en r5.
Vi kan sjekke nøyaktigheten til ESMFold-prediksjonen ved å sammenligne den med den eksperimentelle strukturen. Vi gjør dette ved å bruke US-Align verktøy utviklet av Zhang Lab ved University of Michigan:
| PDB-kjede1 | PDB-kjede2 | TM-score |
| data/prediksjon.pdb:A | data/eksperimentell.pdb:B | 0.802 |
De mal modellering poengsum (TM-score) er en beregning for å vurdere likheten mellom proteinstrukturer. En poengsum på 1.0 indikerer en perfekt match. Poeng over 0.7 indikerer at proteiner deler samme ryggradsstruktur. Poeng over 0.9 indikerer at proteinene er funksjonelt utskiftbare for nedstrøms bruk. I vårt tilfelle med å oppnå TM-score 0.802, vil ESMFold-prediksjonen sannsynligvis være passende for applikasjoner som strukturscoring eller ligandbindingseksperimenter, men kan ikke være egnet for brukstilfeller som molekylær erstatning som krever ekstremt høy nøyaktighet.
Vi kan validere dette resultatet ved å visualisere de justerte strukturene. De to strukturene viser en høy, men ikke perfekt, grad av overlapping. Proteinstrukturspådommer er et felt i rask utvikling, og mange forskerteam utvikler stadig mer nøyaktige algoritmer!
Distribuer ESMFold som et SageMaker-slutningsendepunkt
Å kjøre modellslutninger i en bærbar PC er greit for eksperimentering, men hva om du trenger å integrere modellen med en applikasjon? Eller en MLOps-rørledning? I dette tilfellet er et bedre alternativ å distribuere modellen din som et slutningsendepunkt. I det følgende eksempelet vil vi distribuere ESMFold som et SageMaker-endepunkt for sanntidsslutning på en akselerert forekomst. SageMaker sanntidsendepunkter gir en skalerbar, kostnadseffektiv og sikker måte å distribuere og være vert for maskinlæringsmodeller (ML). Med automatisk skalering kan du justere antall forekomster som kjører endepunktet for å møte kravene til applikasjonen din, optimalisere kostnadene og sikre høy tilgjengelighet.
Den ferdigbygde SageMaker beholder for Hugging Face gjør det enkelt å distribuere dyplæringsmodeller for vanlige oppgaver. For nye brukstilfeller som prediksjon av proteinstruktur, må vi imidlertid definere en skikk inference.py skript for å laste modellen, kjøre prediksjonen og formatere utdataene. Dette skriptet inneholder mye av den samme koden som vi brukte i notatboken vår. Vi lager også en requirements.txt fil for å definere noen Python-avhengigheter som endepunktet vårt skal bruke. Du kan se filene vi opprettet i GitHub repository.
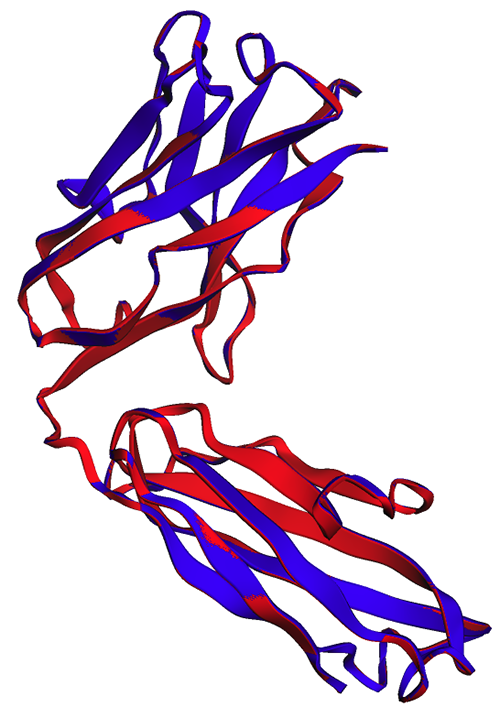
I den følgende figuren er de eksperimentelle (blå) og forutsagte (røde) strukturene til den tunge kjeden av trastuzumab svært like, men ikke identiske.

Etter at vi har opprettet de nødvendige filene i code katalogen, distribuerer vi modellen vår ved å bruke SageMaker HuggingFaceModel klasse. Dette bruker en forhåndsbygd beholder for å forenkle prosessen med å distribuere Hugging Face-modeller til SageMaker. Merk at det kan ta 10 minutter eller mer å opprette endepunktet, avhengig av tilgjengeligheten til ml.g4dn instanstyper i vår region.
Når endepunktsimplementeringen er fullført, kan vi sende inn proteinsekvensen på nytt og vise de første radene i prediksjonen:
Fordi vi distribuerte endepunktet vårt til en akselerert forekomst, bør prediksjonen bare ta noen få sekunder. Hver rad i resultatet tilsvarer et enkelt atom og inkluderer aminosyreidentiteten, tre romlige koordinater og en pLDDT-score som representerer prediksjonstilliten på det stedet.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | BELEGG | PLDDT | ATOM_ID |
| ATOM | 1 | N | G.L.U. | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | G.L.U. | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | G.L.U. | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | G.L.U. | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | G.L.U. | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | G.L.U. | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | G.L.U. | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | G.L.U. | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | G.L.U. | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Ved å bruke samme metode som før ser vi at notisboken og endepunktsprediksjonene er identiske.
| PDB-kjede1 | PDB-kjede2 | TM-score |
| data/endpoint_prediction.pdb:A | data/prediksjon.pdb:A | 1.0 |
Som observert i den følgende figuren, viser ESMFold-spådommene generert i den bærbare datamaskinen (rød) og ved endepunktet (blått) perfekt justering.
Rydd opp
For å unngå ytterligere gebyrer sletter vi slutningsendepunktet og testdataene våre:
Oppsummering
Beregningsbasert proteinstrukturprediksjon er et kritisk verktøy for å forstå funksjonen til proteiner. I tillegg til grunnforskning har algoritmer som AlphaFold og ESMFold mange bruksområder innen medisin og bioteknologi. Den strukturelle innsikten generert av disse modellene hjelper oss å bedre forstå hvordan biomolekyler samhandler. Dette kan da føre til bedre diagnostiske verktøy og terapier for pasienter.
I dette innlegget viser vi hvordan du distribuerer ESMFold-proteinspråkmodellen fra Hugging Face Hub som et skalerbart sluttpunkt ved bruk av SageMaker. For mer informasjon om distribusjon av Hugging Face-modeller på SageMaker, se Bruk Hugging Face med Amazon SageMaker. Du kan også finne flere proteinvitenskapelige eksempler i Fantastisk proteinanalyse på AWS GitHub repo. Legg igjen en kommentar hvis det er noen andre eksempler du vil se!
Om forfatterne
 Brian lojal er senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han har mer enn 17 års erfaring innen bioteknologi og maskinlæring, og brenner for å hjelpe kunder med å løse genomiske og proteomiske utfordringer. På fritiden liker han å lage mat og spise sammen med venner og familie.
Brian lojal er senior AI/ML Solutions Architect i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han har mer enn 17 års erfaring innen bioteknologi og maskinlæring, og brenner for å hjelpe kunder med å løse genomiske og proteomiske utfordringer. På fritiden liker han å lage mat og spise sammen med venner og familie.
 Shamika Ariyawansa er en AI/ML-spesialistløsningsarkitekt i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han jobber lidenskapelig med kunder for å akselerere deres AI- og ML-adopsjon ved å gi teknisk veiledning og hjelpe dem med å innovere og bygge sikre skyløsninger på AWS. Utenom jobben elsker han ski og terrengkjøring.
Shamika Ariyawansa er en AI/ML-spesialistløsningsarkitekt i Global Healthcare and Life Sciences-teamet hos Amazon Web Services. Han jobber lidenskapelig med kunder for å akselerere deres AI- og ML-adopsjon ved å gi teknisk veiledning og hjelpe dem med å innovere og bygge sikre skyløsninger på AWS. Utenom jobben elsker han ski og terrengkjøring.
 Yanjun Qi er Senior Applied Science Manager ved AWS Machine Learning Solution Lab. Hun innoverer og bruker maskinlæring for å hjelpe AWS-kunder med å få fart på AI og skyadopsjon.
Yanjun Qi er Senior Applied Science Manager ved AWS Machine Learning Solution Lab. Hun innoverer og bruker maskinlæring for å hjelpe AWS-kunder med å få fart på AI og skyadopsjon.
- SEO-drevet innhold og PR-distribusjon. Bli forsterket i dag.
- PlatoAiStream. Web3 Data Intelligence. Kunnskap forsterket. Tilgang her.
- Minting the Future med Adryenn Ashley. Tilgang her.
- Kjøp og selg aksjer i PRE-IPO-selskaper med PREIPO®. Tilgang her.
- kilde: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- : har
- :er
- :ikke
- $OPP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Om oss
- ovenfor
- AC
- akselerere
- akselerert
- nøyaktighet
- nøyaktig
- nøyaktig
- oppnå
- aktivitet
- tillegg
- Adopsjon
- AI
- AI / ML
- algoritme
- algoritmer
- justert
- innretting
- også
- Selv
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- analyse
- og
- En annen
- noen
- Søknad
- søknader
- anvendt
- hensiktsmessig
- ER
- AS
- vurdere
- At
- atom
- oppmerksomhet
- Automatisk
- tilgjengelighet
- unngå
- AWS
- AWS maskinlæring
- Backbone
- Bank
- grunnleggende
- BE
- før du
- begynne
- Bedre
- mellom
- bindende
- bioteknologi
- Blå
- bygge
- men
- by
- CAN
- Kreft
- tegnefilm
- saken
- saker
- feiret
- kjede
- utfordringer
- avgifter
- sjekk
- klasse
- Cloud
- skyadopsjon
- kode
- farge
- kommer
- kommentere
- Felles
- sammenligne
- sammenligne
- fullføre
- selvtillit
- Container
- kontrast
- tilsvarer
- kostnadseffektiv
- Kostnader
- kunne
- skape
- opprettet
- kritisk
- Kunder
- dato
- databaser
- dato tid
- dyp
- dyp læring
- DeepMind
- Grad
- krav
- avhengig
- utplassere
- utplassert
- utplasserings
- distribusjon
- Bestem
- bestemmes
- utviklet
- utvikle
- Utvikling
- enhet
- vanskelig
- Vise
- do
- gjør
- ikke
- nedlasting
- stasjonen
- Narkotika
- under
- hver enkelt
- lett
- effekt
- effekter
- muliggjør
- slutt
- Endpoint
- sikrer
- Miljø
- eksempel
- eksempler
- dyrt
- erfaring
- eksperimenter
- utvendig
- ekstremt
- Face
- familie
- raskere
- Noen få
- færre
- felt
- Figur
- filet
- Filer
- Finn
- slutt
- Først
- etter
- Til
- format
- venner
- fra
- funksjon
- videre
- generere
- generert
- GitHub
- Global
- Grønn
- veiledning
- Ha
- he
- helsetjenester
- tung
- hjelpe
- hjelpe
- Høy
- hans
- vert
- Hvordan
- Hvordan
- Men
- HTML
- HTTPS
- Hub
- Klem ansikt
- identiske
- Identitet
- if
- bilde
- importere
- viktig
- forbedre
- forbedret
- in
- inkluderer
- indikerer
- indikerer
- informasjon
- innovere
- inngang
- innsikt
- innsikt
- f.eks
- i stedet
- integrere
- samhandle
- interaksjoner
- interaktiv
- interaktiv
- inn
- IT
- DET ER
- lab
- Språk
- stor
- føre
- læring
- Permisjon
- bibliotekene
- Bibliotek
- Life
- Life Sciences
- lett
- i likhet med
- Sannsynlig
- laste
- plassering
- oppslag
- maskin
- maskinlæring
- gjøre
- GJØR AT
- leder
- mange
- Match
- Kan..
- medisin
- Møt
- metode
- metoder
- metrisk
- Michigan
- millioner
- minutter
- ML
- MLOps
- Mote
- modell
- modeller
- modifikasjoner
- molekyl~~POS=TRUNC
- mer
- mye
- navn
- nødvendig
- Trenger
- NIH
- bærbare
- roman
- Antall
- gjenstander
- foreldet
- of
- on
- ONE
- bare
- opererer
- optimalisere
- Alternativ
- or
- oransje
- OS
- Annen
- vår
- produksjon
- utenfor
- parametere
- lidenskapelig
- pasient
- pasienter
- mønstre
- perfekt
- ytelse
- rørledning
- plato
- Platon Data Intelligence
- PlatonData
- vær så snill
- Post
- potensielt
- forutsi
- spådd
- forutsi
- prediksjon
- Spådommer
- Forbered
- tidligere
- prosess
- Prosesser
- Protein
- Proteiner
- gi
- gir
- gi
- Python
- pytorch
- Qi
- raskt
- raskt
- felt i rask utvikling
- sanntids
- anbefaler
- Rød
- region
- i slekt
- avhengige
- representere
- representerer
- representerer
- krever
- forskning
- forskere
- resultere
- RAD
- Kjør
- rennende
- sagemaker
- SageMaker Inference
- samme
- Spar
- skalerbar
- skalering
- Vitenskap
- VITENSKAPER
- Resultat
- scoring
- skript
- sekunder
- sikre
- se
- senior
- Sequence
- Tjenester
- sett
- flere
- Del
- hun
- bør
- Vis
- vist
- side
- lignende
- forenkle
- enkelt
- løsning
- Solutions
- LØSE
- noen
- romlig
- spesialist
- Spektroskopi
- fart
- Begynn
- state-of-the-art
- Trinn
- strukturell
- struktur
- studio
- Studer
- slik
- egnet
- støtte
- overlevelse
- Ta
- tar
- mål
- oppgaver
- lag
- lag
- Teknisk
- test
- enn
- Det
- De
- deres
- Dem
- deretter
- Der.
- Disse
- denne
- tre
- tredimensjonal
- tid
- tidkrevende
- ganger
- til
- symbolisere
- symbolbaserte
- verktøy
- verktøy
- lommelykt
- trent
- Kurs
- transformers
- transportere
- behandling
- to
- typen
- typer
- forstå
- forståelse
- universitet
- I motsetning til
- us
- bruke
- brukt
- ved hjelp av
- VALIDERE
- veldig
- Se
- visualisering
- W
- ønsket
- Vei..
- we
- web
- webtjenester
- velkjent
- Hva
- Wikipedia
- vil
- med
- WordPress
- Arbeid
- virker
- ville
- røntgen
- Du
- Din
- zephyrnet