Białka napędzają wiele procesów biologicznych, takich jak aktywność enzymów, transport molekularny i wsparcie komórkowe. Trójwymiarowa struktura białka zapewnia wgląd w jego funkcję i sposób interakcji z innymi biomolekułami. Eksperymentalne metody określania struktury białek, takie jak krystalografia rentgenowska i spektroskopia NMR, są drogie i czasochłonne.
W przeciwieństwie do tego, niedawno opracowane metody obliczeniowe mogą szybko i dokładnie przewidzieć strukturę białka na podstawie jego sekwencji aminokwasowej. Metody te mają kluczowe znaczenie dla białek trudnych do badania eksperymentalnego, takich jak białka błonowe, będące celem wielu leków. Jednym z dobrze znanych przykładów jest to AlfaFold, algorytm oparty na głębokim uczeniu się, znany ze swoich dokładnych prognoz.
ESMfold to kolejna bardzo dokładna metoda oparta na głębokim uczeniu się, opracowana w celu przewidywania struktury białka na podstawie jego sekwencji aminokwasowej. ESMFold wykorzystuje duży model języka białek (pLM) jako szkielet i działa od początku do końca. W przeciwieństwie do AlphaFold2, nie wymaga wyszukiwania ani Dopasowanie wielu sekwencji (MSA), ani nie polega na zewnętrznych bazach danych do generowania prognoz. Zamiast tego zespół programistów przeszkolił model na milionach sekwencji białkowych z UniRef. Podczas treningu model rozwinął wzorce uwagi, które elegancko reprezentują ewolucyjne interakcje między aminokwasami w sekwencji. To zastosowanie pLM zamiast MSA zapewnia do 60 razy szybsze czasy przewidywania niż inne najnowocześniejsze modele.
W tym poście używamy wstępnie wytrenowanego modelu ESMFold z Hugging Face with Amazon Sage Maker przewidzieć strukturę łańcucha ciężkiego trastuzumab, A przeciwciało monoklonalne po raz pierwszy opracowany przez Genentech do leczenia HER2-dodatni rak piersi. Szybkie przewidywanie struktury tego białka może być przydatne, jeśli naukowcy chcą przetestować wpływ modyfikacji sekwencji. Może to potencjalnie prowadzić do poprawy przeżycia pacjentów lub mniejszej liczby skutków ubocznych.
Ten post zawiera przykładowy notatnik Jupyter i powiązane skrypty poniżej Repozytorium GitHub.
Wymagania wstępne
Zalecamy uruchomienie tego przykładu w pliku Studio Amazon SageMaker notatnik uruchomienie zoptymalizowanego pod kątem procesora obrazu PyTorch 1.13 Python 3.9 na instancji typu ml.r5.xlarge.
Wizualizuj eksperymentalną strukturę trastuzumabu
Na początek używamy biopython bibliotekę i skrypt pomocniczy do pobierania struktury trastuzumabu z pliku Bank danych białek RCSB:
Następnie używamy py3Dmol biblioteka do wizualizacji konstrukcji jako interaktywnej wizualizacji 3D:
Poniższy rysunek przedstawia trójwymiarową strukturę białka 3N1Z z Protein Data Bank (PDB). Na tym obrazie łańcuch lekki trastuzumabu jest zaznaczony na pomarańczowo, łańcuch ciężki na niebiesko (z regionem zmiennym na jasnoniebiesko), a antygen HER8 na zielono.
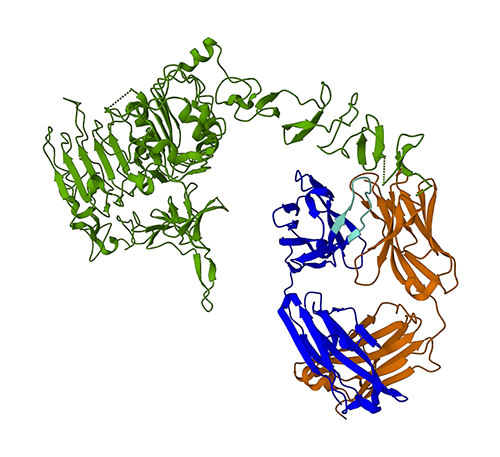
Najpierw użyjemy ESMFold do przewidywania struktury łańcucha ciężkiego (łańcuch B) na podstawie jego sekwencji aminokwasowej. Następnie porównamy prognozę z eksperymentalnie określoną strukturą pokazaną powyżej.
Przewiduj strukturę łańcucha ciężkiego trastuzumabu na podstawie jego sekwencji za pomocą ESMFold
Użyjmy modelu ESMFold do przewidzenia struktury łańcucha ciężkiego i porównajmy go z wynikiem eksperymentalnym. Na początek użyjemy gotowego środowiska notatnika w Studio, które zawiera kilka ważnych bibliotek, takich jak PyTorch, preinstalowany. Chociaż moglibyśmy użyć przyspieszonego typu instancji, aby poprawić wydajność naszej analizy notatnika, zamiast tego użyjemy instancji bez przyspieszenia i uruchomimy prognozę ESMFold na procesorze.
Najpierw ładujemy wstępnie wytrenowany model ESMFold i tokenizer z Przytulanie twarzy Hub:
Następnie kopiujemy model na nasze urządzenie (w tym przypadku CPU) i ustawiamy kilka parametrów modelu:
Aby przygotować sekwencję białka do analizy, musimy ją stokenizować. To tłumaczy symbole aminokwasów (EVQLV…) na format numeryczny zrozumiały dla modelu ESMFold (6,19,5,10,19,…):
Następnie kopiujemy tokenizowane dane wejściowe do trybu, dokonujemy prognozy i zapisujemy wynik do pliku:
Zajmuje to około 3 minut w przypadku typu instancji bez przyspieszenia, takiego jak r5.
Dokładność predykcji ESMFold możemy sprawdzić, porównując ją ze strukturą eksperymentalną. Robimy to za pomocą Wyrównaj do USA narzędzie opracowane przez Zhang Lab na Uniwersytecie Michigan:
| Łańcuch PDB1 | Łańcuch PDB2 | Wynik TM |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
Połączenia wynik modelowania szablonów (TM-score) jest miarą służącą do oceny podobieństwa struktur białek. Wynik 1.0 oznacza idealne dopasowanie. Wyniki powyżej 0.7 wskazują, że białka mają tę samą strukturę szkieletu. Wyniki powyżej 0.9 wskazują, że białka są funkcjonalnie wymienne do dalszego użytku. W naszym przypadku osiągnięcia TM-Score 0.802 prognoza ESMFold byłaby prawdopodobnie odpowiednia do zastosowań takich jak ocenianie struktury lub eksperymenty z wiązaniem ligandów, ale może nie być odpowiednia w przypadkach użycia, takich jak wymiana molekularna które wymagają bardzo dużej dokładności.
Możemy zweryfikować ten wynik, wizualizując wyrównane struktury. Obie struktury wykazują wysoki, ale nie doskonały stopień nakładania się. Przewidywanie struktury białek to szybko rozwijająca się dziedzina, a wiele zespołów badawczych opracowuje coraz dokładniejsze algorytmy!
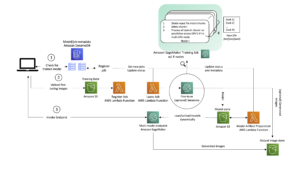
Wdróż ESMFold jako punkt końcowy wnioskowania SageMaker
Uruchamianie wnioskowania o modelu w notesie jest dobre do eksperymentowania, ale co zrobić, jeśli trzeba zintegrować model z aplikacją? A może potok MLOps? W takim przypadku lepszą opcją jest wdrożenie modelu jako punktu końcowego wnioskowania. W poniższym przykładzie wdrożymy ESMFold jako punkt końcowy wnioskowania w czasie rzeczywistym SageMaker w przyspieszonej instancji. Punkty końcowe SageMaker działające w czasie rzeczywistym zapewniają skalowalny, ekonomiczny i bezpieczny sposób wdrażania i hostowania modeli uczenia maszynowego (ML). Dzięki automatycznemu skalowaniu możesz dostosować liczbę instancji z uruchomionym punktem końcowym, aby sprostać wymaganiom aplikacji, optymalizując koszty i zapewniając wysoką dostępność.
Gotowy Pojemnik SageMaker do przytulania twarzy ułatwia wdrażanie modeli głębokiego uczenia do typowych zadań. Jednak w przypadku nowych przypadków użycia, takich jak przewidywanie struktury białek, musimy zdefiniować zwyczaj inference.py skrypt, aby załadować model, uruchomić prognozę i sformatować dane wyjściowe. Ten skrypt zawiera większość tego samego kodu, którego użyliśmy w naszym notatniku. Tworzymy również requirements.txt plik, aby zdefiniować niektóre zależności Pythona do użycia przez nasz punkt końcowy. Możesz zobaczyć pliki, które stworzyliśmy w Repozytorium GitHub.
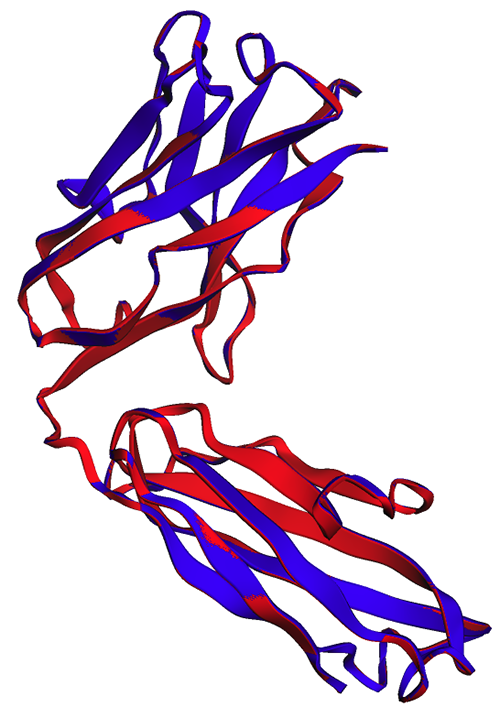
Na poniższym rysunku eksperymentalna (niebieska) i przewidywana (czerwona) struktura łańcucha ciężkiego trastuzumabu jest bardzo podobna, ale nie identyczna.

Po utworzeniu niezbędnych plików w code katalogu, wdrażamy nasz model za pomocą SageMaker HuggingFaceModel klasa. Wykorzystuje to gotowy kontener, aby uprościć proces wdrażania modeli Hugging Face w SageMaker. Pamiętaj, że utworzenie punktu końcowego może zająć 10 minut lub więcej, w zależności od dostępności ml.g4dn typów instancji w naszym Regionie.
Po zakończeniu wdrażania punktu końcowego możemy ponownie przesłać sekwencję białek i wyświetlić kilka pierwszych wierszy prognozy:
Ponieważ wdrożyliśmy nasz punkt końcowy w przyspieszonej instancji, przewidywanie powinno zająć tylko kilka sekund. Każdy wiersz w wyniku odpowiada pojedynczemu atomowi i zawiera tożsamość aminokwasu, trzy współrzędne przestrzenne i a wynik pLDDT reprezentujący pewność prognozy w tej lokalizacji.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | ID_ŁAŃCUCHA | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | OKUPACJA | PLDDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Używając tej samej metody co poprzednio, widzimy, że przewidywania notatnika i punktu końcowego są identyczne.
| Łańcuch PDB1 | Łańcuch PDB2 | Wynik TM |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
Jak widać na poniższym rysunku, prognozy ESMFold generowane w notatniku (czerwony) i przez punkt końcowy (niebieski) wykazują doskonałe wyrównanie.
Sprzątać
Aby uniknąć dalszych opłat, usuwamy nasz punkt końcowy wnioskowania i dane testowe:
Podsumowanie
Obliczeniowe przewidywanie struktury białek jest kluczowym narzędziem do zrozumienia funkcji białek. Oprócz badań podstawowych algorytmy takie jak AlphaFold i ESMFold mają wiele zastosowań w medycynie i biotechnologii. Wgląd strukturalny generowany przez te modele pomaga nam lepiej zrozumieć, w jaki sposób wchodzą w interakcje biomolekuły. Może to następnie prowadzić do lepszych narzędzi diagnostycznych i terapii dla pacjentów.
W tym poście pokazujemy, jak wdrożyć model języka białkowego ESMFold z Hugging Face Hub jako skalowalny punkt końcowy wnioskowania przy użyciu SageMaker. Aby uzyskać więcej informacji na temat wdrażania modeli Hugging Face w SageMaker, zobacz Użyj Przytulanie twarzy z Amazon SageMaker. Możesz także znaleźć więcej przykładów nauki o białkach w Niesamowita analiza białek na AWS Repozytorium GitHub. Zostaw nam komentarz, jeśli są jakieś inne przykłady, które chciałbyś zobaczyć!
O autorach
 Briana Loyala jest starszym architektem rozwiązań AI/ML w zespole Global Healthcare and Life Sciences w Amazon Web Services. Ma ponad 17-letnie doświadczenie w biotechnologii i uczeniu maszynowym, a jego pasją jest pomaganie klientom w rozwiązywaniu problemów genomicznych i proteomicznych. W wolnym czasie lubi gotować i jeść z przyjaciółmi i rodziną.
Briana Loyala jest starszym architektem rozwiązań AI/ML w zespole Global Healthcare and Life Sciences w Amazon Web Services. Ma ponad 17-letnie doświadczenie w biotechnologii i uczeniu maszynowym, a jego pasją jest pomaganie klientom w rozwiązywaniu problemów genomicznych i proteomicznych. W wolnym czasie lubi gotować i jeść z przyjaciółmi i rodziną.
 Shamika Ariyawanę jest Architektem Rozwiązań Specjalistycznych AI/ML w zespole Global Healthcare and Life Sciences w Amazon Web Services. Z pasją współpracuje z klientami, aby przyspieszyć wdrażanie AI i ML, udzielając wskazówek technicznych i pomagając im wprowadzać innowacje i budować bezpieczne rozwiązania chmurowe na AWS. Poza pracą uwielbia jeździć na nartach i w terenie.
Shamika Ariyawanę jest Architektem Rozwiązań Specjalistycznych AI/ML w zespole Global Healthcare and Life Sciences w Amazon Web Services. Z pasją współpracuje z klientami, aby przyspieszyć wdrażanie AI i ML, udzielając wskazówek technicznych i pomagając im wprowadzać innowacje i budować bezpieczne rozwiązania chmurowe na AWS. Poza pracą uwielbia jeździć na nartach i w terenie.
 Yanjun Qi jest starszym kierownikiem ds. nauk stosowanych w AWS Machine Learning Solution Lab. Wprowadza innowacje i stosuje uczenie maszynowe, aby pomóc klientom AWS przyspieszyć wdrażanie sztucznej inteligencji i chmury.
Yanjun Qi jest starszym kierownikiem ds. nauk stosowanych w AWS Machine Learning Solution Lab. Wprowadza innowacje i stosuje uczenie maszynowe, aby pomóc klientom AWS przyspieszyć wdrażanie sztucznej inteligencji i chmury.
- Dystrybucja treści i PR oparta na SEO. Uzyskaj wzmocnienie już dziś.
- PlatoAiStream. Analiza danych Web3. Wiedza wzmocniona. Dostęp tutaj.
- Wybijanie przyszłości w Adryenn Ashley. Dostęp tutaj.
- Kupuj i sprzedawaj akcje spółek PRE-IPO z PREIPO®. Dostęp tutaj.
- Źródło: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :ma
- :Jest
- :nie
- $W GÓRĘ
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- O nas
- powyżej
- AC
- przyśpieszyć
- przyśpieszony
- precyzja
- dokładny
- dokładnie
- osiągnięcia
- działalność
- dodatek
- Przyjęcie
- AI
- AI / ML
- algorytm
- Algorytmy
- wyrównany
- wyrównanie
- również
- Chociaż
- Amazonka
- Amazon Sage Maker
- Amazon Web Services
- an
- analiza
- i
- Inne
- każdy
- Zastosowanie
- aplikacje
- stosowany
- właściwy
- SĄ
- AS
- oceniając
- At
- atom
- Uwaga
- automatycznie
- dostępność
- uniknąć
- AWS
- Uczenie maszynowe AWS
- Kręgosłup
- Bank
- podstawowy
- BE
- zanim
- rozpocząć
- Ulepsz Swój
- pomiędzy
- wiążący
- biotechnologia
- Niebieski
- budować
- ale
- by
- CAN
- Rak
- rysunek
- walizka
- Etui
- celebrujący
- łańcuch
- wyzwania
- Opłaty
- ZOBACZ
- klasa
- Chmura
- adopcja chmury
- kod
- kolor
- byliśmy spójni, od początku
- komentarz
- wspólny
- porównać
- porównanie
- kompletny
- pewność siebie
- Pojemnik
- kontrast
- odpowiada
- opłacalne
- Koszty:
- mógłby
- Stwórz
- stworzony
- krytyczny
- Klientów
- dane
- Bazy danych
- data i godzina
- głęboko
- głęboka nauka
- DeepMind
- Stopień
- wymagania
- W zależności
- rozwijać
- wdrażane
- wdrażanie
- Wdrożenie
- Ustalać
- ustalona
- rozwinięty
- rozwijanie
- oprogramowania
- urządzenie
- trudny
- Wyświetlacz
- do
- robi
- Nie
- pobieranie
- napęd
- Narkotyki
- podczas
- każdy
- łatwo
- efekt
- ruchomości
- Umożliwia
- zakończenia
- Punkt końcowy
- zapewnienie
- Środowisko
- przykład
- przykłady
- drogi
- doświadczenie
- eksperymenty
- zewnętrzny
- niezwykle
- Twarz
- członków Twojej rodziny
- szybciej
- kilka
- mniej
- pole
- Postać
- filet
- Akta
- Znajdź
- w porządku
- i terminów, a
- następujący
- W razie zamówieenia projektu
- format
- przyjaciele
- od
- funkcjonować
- dalej
- Generować
- wygenerowane
- GitHub
- Globalne
- Zielony
- poradnictwo
- Have
- he
- opieki zdrowotnej
- ciężki
- pomoc
- pomoc
- Wysoki
- jego
- gospodarz
- W jaki sposób
- How To
- Jednak
- HTML
- HTTPS
- Piasta
- Przytulanie twarzy
- identiques
- tożsamość
- if
- obraz
- importować
- ważny
- podnieść
- ulepszony
- in
- obejmuje
- wskazać
- wskazuje
- Informacja
- wprowadzać innowacje
- wkład
- wgląd
- spostrzeżenia
- przykład
- zamiast
- integrować
- interakcji
- Interakcje
- interaktywne
- współdziała
- najnowszych
- IT
- JEGO
- laboratorium
- język
- duży
- prowadzić
- nauka
- Pozostawiać
- biblioteki
- Biblioteka
- życie
- Life Sciences
- lekki
- lubić
- Prawdopodobnie
- załadować
- lokalizacja
- wyszukiwania
- maszyna
- uczenie maszynowe
- robić
- WYKONUJE
- kierownik
- wiele
- Mecz
- Może..
- lekarstwo
- Poznaj nasz
- metoda
- metody
- metryczny
- Michigan
- miliony
- minut
- ML
- MLOps
- Moda
- model
- modele
- Modyfikacje
- Cząsteczkowa
- jeszcze
- dużo
- Nazwa
- niezbędny
- Potrzebować
- NIH
- notatnik
- powieść
- numer
- obiekty
- przestarzały
- of
- on
- ONE
- tylko
- działa
- optymalizacji
- Option
- or
- Orange
- OS
- Inne
- ludzkiej,
- wydajność
- zewnętrzne
- parametry
- namiętny
- pacjent
- pacjenci
- wzory
- doskonały
- jest gwarancją najlepszej jakości, które mogą dostarczyć Ci Twoje monitory,
- rurociąg
- plato
- Analiza danych Platona
- PlatoDane
- Proszę
- Post
- potencjalnie
- przewidzieć
- Przewiduje
- przewidywanie
- przepowiednia
- Przewidywania
- Przygotować
- poprzednio
- wygląda tak
- procesów
- Białko
- Białka
- zapewniać
- zapewnia
- że
- Python
- płomień
- Qi
- szybko
- szybko
- szybko rozwijającą się dziedziną
- w czasie rzeczywistym
- polecić
- Czerwony
- region
- związane z
- polegać
- reprezentować
- reprezentowanie
- reprezentuje
- wymagać
- Badania naukowe
- Badacze
- dalsze
- RZĄD
- run
- bieganie
- sagemaker
- Wnioskowanie SageMakera
- taki sam
- Zapisz
- skalowalny
- skalowaniem
- nauka
- NAUKI
- wynik
- punktacji
- skrypty
- sekund
- bezpieczne
- widzieć
- senior
- Sekwencja
- Usługi
- zestaw
- kilka
- Share
- ona
- powinien
- pokazać
- pokazane
- bok
- podobny
- upraszczać
- pojedynczy
- rozwiązanie
- Rozwiązania
- ROZWIĄZANIA
- kilka
- Przestrzenne
- specjalista
- Spektroskopia
- prędkość
- początek
- state-of-the-art
- Ewolucja krok po kroku
- strukturalny
- Struktura
- studio
- Badanie
- taki
- odpowiedni
- wsparcie
- przetrwanie
- Brać
- trwa
- cele
- zadania
- zespół
- Zespoły
- Techniczny
- test
- niż
- że
- Połączenia
- ich
- Im
- następnie
- Tam.
- Te
- to
- trzy
- trójwymiarowy
- czas
- czasochłonne
- czasy
- do
- tokenizować
- tokenizowany
- narzędzie
- narzędzia
- pochodnia
- przeszkolony
- Trening
- Transformatory
- transportu
- leczenie
- drugiej
- rodzaj
- typy
- zrozumieć
- zrozumienie
- uniwersytet
- w odróżnieniu
- us
- posługiwać się
- używany
- za pomocą
- UPRAWOMOCNIĆ
- początku.
- Zobacz i wysłuchaj
- wyobrażanie sobie
- W
- poszukiwany
- Droga..
- we
- sieć
- usługi internetowe
- znane
- Co
- Wikipedia
- będzie
- w
- WordPress
- Praca
- działa
- by
- rentgenowski
- You
- Twój
- zefirnet