As proteínas conduzem muitos processos biológicos, como atividade enzimática, transporte molecular e suporte celular. A estrutura tridimensional de uma proteína fornece informações sobre sua função e como ela interage com outras biomoléculas. Métodos experimentais para determinar a estrutura da proteína, como cristalografia de raios X e espectroscopia de RMN, são caros e demorados.
Em contraste, métodos computacionais desenvolvidos recentemente podem prever com rapidez e precisão a estrutura de uma proteína a partir de sua sequência de aminoácidos. Esses métodos são críticos para proteínas difíceis de estudar experimentalmente, como proteínas de membrana, alvos de muitos medicamentos. Um exemplo bem conhecido disso é AlfaFold, um algoritmo baseado em aprendizado profundo celebrado por suas previsões precisas.
ESMFold é outro método baseado em aprendizado profundo altamente preciso desenvolvido para prever a estrutura da proteína a partir de sua sequência de aminoácidos. O ESMFold usa um grande modelo de linguagem de proteína (pLM) como backbone e opera de ponta a ponta. Ao contrário do AlphaFold2, ele não precisa de uma pesquisa ou Alinhamento de Múltiplas Sequências (MSA), nem depende de bancos de dados externos para gerar previsões. Em vez disso, a equipe de desenvolvimento treinou o modelo em milhões de sequências de proteínas da UniRef. Durante o treinamento, o modelo desenvolveu padrões de atenção que representam elegantemente as interações evolutivas entre os aminoácidos da sequência. Esse uso de um pLM em vez de um MSA permite tempos de previsão até 60 vezes mais rápidos do que outros modelos de última geração.
Nesta postagem, usamos o modelo ESMFold pré-treinado de Hugging Face com Amazon Sage Maker para prever a estrutura da cadeia pesada de trastuzumab, um anticorpo monoclonal desenvolvido pela primeira vez por Genentech para o tratamento de Câncer de mama HER2-positivo. Prever rapidamente a estrutura dessa proteína pode ser útil se os pesquisadores quiserem testar o efeito das modificações na sequência. Isso poderia potencialmente levar a uma melhor sobrevida do paciente ou a menos efeitos colaterais.
Esta postagem fornece um exemplo de notebook Jupyter e scripts relacionados a seguir Repositório GitHub.
Pré-requisitos
Recomendamos executar este exemplo em um Estúdio Amazon SageMaker caderno executando a imagem otimizada para CPU PyTorch 1.13 Python 3.9 em um tipo de instância ml.r5.xlarge.
Visualize a estrutura experimental do trastuzumabe
Para começar, usamos o biopython biblioteca e um script auxiliar para baixar a estrutura trastuzumab do Banco de Dados de Proteína RCSB:
Em seguida, usamos o py3Dmol biblioteca para visualizar a estrutura como uma visualização 3D interativa:
A figura a seguir representa a estrutura 3D da proteína 1N8Z do Protein Data Bank (PDB). Nesta imagem, a cadeia leve do trastuzumabe é exibida em laranja, a cadeia pesada é azul (com a região variável em azul claro) e o antígeno HER2 é verde.
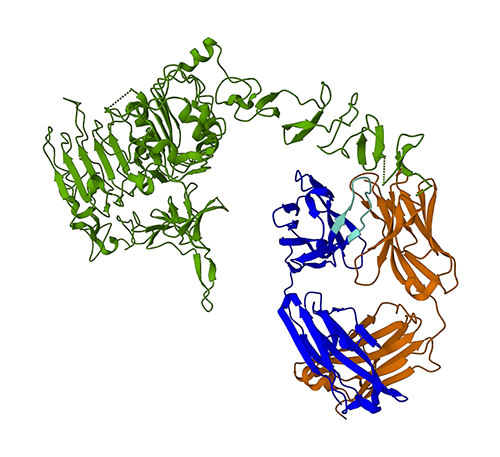
Primeiro usaremos o ESMFold para prever a estrutura da cadeia pesada (Cadeia B) a partir de sua sequência de aminoácidos. Em seguida, compararemos a previsão com a estrutura determinada experimentalmente mostrada acima.
Preveja a estrutura da cadeia pesada do trastuzumabe a partir de sua sequência usando o ESMFold
Vamos usar o modelo ESMFold para prever a estrutura da cadeia pesada e compará-la com o resultado experimental. Para começar, usaremos um ambiente de notebook pré-construído no Studio que vem com várias bibliotecas importantes, como PyTorch, pré-instalado. Embora possamos usar um tipo de instância acelerada para melhorar o desempenho de nossa análise de notebook, usaremos uma instância não acelerada e executaremos a previsão ESMFold em uma CPU.
Primeiro, carregamos o modelo ESMFold pré-treinado e o tokenizador de Abraçando o Face Hub:
Em seguida, copiamos o modelo para o nosso dispositivo (CPU neste caso) e definimos alguns parâmetros do modelo:
Para preparar a sequência de proteínas para análise, precisamos tokenizá-la. Isso traduz os símbolos de aminoácidos (EVQLV…) em um formato numérico que o modelo ESMFold pode entender (6,19,5,10,19,…):
Em seguida, copiamos a entrada tokenizada para o modo, fazemos uma previsão e salvamos o resultado em um arquivo:
Isso leva cerca de 3 minutos em um tipo de instância não acelerada, como uma r5.
Podemos verificar a precisão da previsão do ESMFold comparando-a com a estrutura experimental. Fazemos isso usando o Alinhamento dos EUA ferramenta desenvolvida pelo Zhang Lab da Universidade de Michigan:
| Cadeia PDB1 | Cadeia PDB2 | Pontuação TM |
| data/previsão.pdb:A | dados/experimental.pdb:B | 0.802 |
A pontuação de modelagem de modelo (TM-score) é uma métrica para avaliar a similaridade de estruturas de proteínas. Uma pontuação de 1.0 indica uma correspondência perfeita. Pontuações acima de 0.7 indicam que as proteínas compartilham a mesma estrutura principal. Pontuações acima de 0.9 indicam que as proteínas são funcionalmente intercambiável para uso a jusante. Em nosso caso de atingir TM-Score 0.802, a previsão ESMFold provavelmente seria apropriada para aplicações como pontuação de estrutura ou experimentos de ligação de ligante, mas pode não ser adequada para casos de uso como substituição molecular que exigem precisão extremamente alta.
Podemos validar este resultado visualizando as estruturas alinhadas. As duas estruturas mostram um alto, mas não perfeito, grau de sobreposição. A previsão da estrutura de proteínas é um campo em rápida evolução e muitas equipes de pesquisa estão desenvolvendo algoritmos cada vez mais precisos!
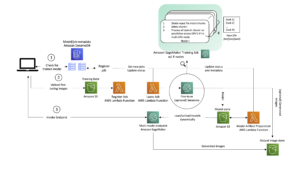
Implante o ESMFold como um endpoint de inferência do SageMaker
Executar inferência de modelo em um notebook é bom para experimentação, mas e se você precisar integrar seu modelo a um aplicativo? Ou um pipeline MLOps? Nesse caso, uma opção melhor é implantar seu modelo como um endpoint de inferência. No exemplo a seguir, implantaremos o ESMFold como um endpoint de inferência em tempo real do SageMaker em uma instância acelerada. Os endpoints em tempo real do SageMaker fornecem uma maneira escalonável, econômica e segura de implantar e hospedar modelos de aprendizado de máquina (ML). Com o dimensionamento automático, você pode ajustar o número de instâncias que executam o endpoint para atender às demandas de seu aplicativo, otimizando custos e garantindo alta disponibilidade.
O pré-construído Contêiner SageMaker para abraçar o rosto facilita a implantação de modelos de aprendizado profundo para tarefas comuns. No entanto, para novos casos de uso, como previsão da estrutura da proteína, precisamos definir um padrão personalizado inference.py script para carregar o modelo, executar a previsão e formatar a saída. Esse script inclui muito do mesmo código que usamos em nosso notebook. Também criamos um requirements.txt arquivo para definir algumas dependências Python para nosso endpoint usar. Você pode ver os arquivos que criamos no Repositório GitHub.
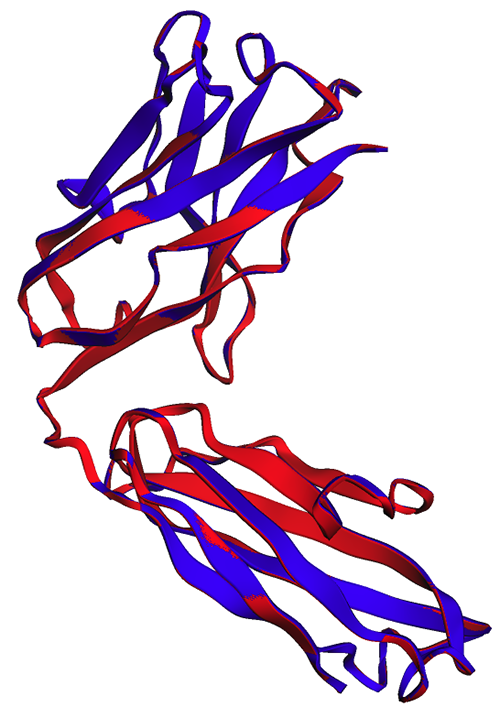
Na figura a seguir, as estruturas experimentais (azul) e previstas (vermelho) da cadeia pesada do trastuzumabe são muito semelhantes, mas não idênticas.

Depois de criarmos os arquivos necessários no code diretório, implantamos nosso modelo usando o SageMaker HuggingFaceModel aula. Isso usa um contêiner pré-construído para simplificar o processo de implantação de modelos Hugging Face no SageMaker. Observe que pode levar 10 minutos ou mais para criar o endpoint, dependendo da disponibilidade de ml.g4dn tipos de instância em nossa região.
Quando a implantação do endpoint estiver concluída, podemos reenviar a sequência da proteína e exibir as primeiras linhas da previsão:
Como implantamos nosso endpoint em uma instância acelerada, a previsão deve levar apenas alguns segundos. Cada linha no resultado corresponde a um único átomo e inclui a identidade do aminoácido, três coordenadas espaciais e um pontuação pLDDT representando a confiança da previsão naquele local.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | OCUPAÇÃO | PLDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Usando o mesmo método de antes, vemos que as previsões de notebook e endpoint são idênticas.
| Cadeia PDB1 | Cadeia PDB2 | Pontuação TM |
| dados/endpoint_prediction.pdb:A | data/previsão.pdb:A | 1.0 |
Conforme observado na figura a seguir, as previsões do ESMFold geradas no notebook (vermelho) e pelo endpoint (azul) mostram um alinhamento perfeito.
limpar
Para evitar cobranças adicionais, excluímos nosso endpoint de inferência e dados de teste:
Resumo
A previsão computacional da estrutura da proteína é uma ferramenta crítica para entender a função das proteínas. Além da pesquisa básica, algoritmos como AlphaFold e ESMFold têm muitas aplicações em medicina e biotecnologia. Os insights estruturais gerados por esses modelos nos ajudam a entender melhor como as biomoléculas interagem. Isso pode levar a melhores ferramentas de diagnóstico e terapias para os pacientes.
Nesta postagem, mostramos como implantar o modelo de linguagem de proteína ESMFold do Hugging Face Hub como um endpoint de inferência escalável usando o SageMaker. Para obter mais informações sobre a implantação de modelos Hugging Face no SageMaker, consulte Use o rosto abraçado com o Amazon SageMaker. Você também pode encontrar mais exemplos de ciência de proteínas no Análise de proteína incrível na AWS repositório GitHub. Por favor, deixe-nos um comentário se houver outros exemplos que você gostaria de ver!
Sobre os autores
 Brian Leal é arquiteto sênior de soluções de IA/ML da equipe global de saúde e ciências biológicas da Amazon Web Services. Ele tem mais de 17 anos de experiência em biotecnologia e aprendizado de máquina e é apaixonado por ajudar os clientes a resolver desafios genômicos e proteômicos. Em seu tempo livre, ele gosta de cozinhar e comer com seus amigos e familiares.
Brian Leal é arquiteto sênior de soluções de IA/ML da equipe global de saúde e ciências biológicas da Amazon Web Services. Ele tem mais de 17 anos de experiência em biotecnologia e aprendizado de máquina e é apaixonado por ajudar os clientes a resolver desafios genômicos e proteômicos. Em seu tempo livre, ele gosta de cozinhar e comer com seus amigos e familiares.
 Shamika Ariyawansa é arquiteto de soluções especialista em IA/ML da equipe global de saúde e ciências biológicas da Amazon Web Services. Ele trabalha com entusiasmo com os clientes para acelerar a adoção de IA e ML, fornecendo orientação técnica e ajudando-os a inovar e criar soluções de nuvem seguras na AWS. Fora do trabalho, ele adora esquiar e praticar off-road.
Shamika Ariyawansa é arquiteto de soluções especialista em IA/ML da equipe global de saúde e ciências biológicas da Amazon Web Services. Ele trabalha com entusiasmo com os clientes para acelerar a adoção de IA e ML, fornecendo orientação técnica e ajudando-os a inovar e criar soluções de nuvem seguras na AWS. Fora do trabalho, ele adora esquiar e praticar off-road.
 Yanjun Qi é gerente sênior de ciência aplicada no Laboratório de soluções de aprendizado de máquina da AWS. Ela inova e aplica aprendizado de máquina para ajudar os clientes da AWS a acelerar a adoção de IA e nuvem.
Yanjun Qi é gerente sênior de ciência aplicada no Laboratório de soluções de aprendizado de máquina da AWS. Ela inova e aplica aprendizado de máquina para ajudar os clientes da AWS a acelerar a adoção de IA e nuvem.
- Conteúdo com tecnologia de SEO e distribuição de relações públicas. Seja amplificado hoje.
- PlatoAiStream. Inteligência de Dados Web3. Conhecimento Amplificado. Acesse aqui.
- Cunhando o Futuro com Adryenn Ashley. Acesse aqui.
- Compre e venda ações em empresas PRE-IPO com PREIPO®. Acesse aqui.
- Fonte: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :tem
- :é
- :não
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Sobre
- acima
- AC
- acelerar
- acelerado
- precisão
- preciso
- exatamente
- alcançar
- atividade
- Adição
- Adoção
- AI
- AI / ML
- algoritmo
- algoritmos
- alinhado
- alinhamento
- tb
- Apesar
- Amazon
- Amazon Sage Maker
- Amazon Web Services
- an
- análise
- e
- Outro
- qualquer
- Aplicação
- aplicações
- aplicado
- apropriado
- SOMOS
- AS
- Avaliando
- At
- átomo
- por WhatsApp.
- Automático
- disponibilidade
- evitar
- AWS
- Aprendizado de máquina da AWS
- Espinha dorsal
- Bank
- basic
- BE
- antes
- começar
- Melhor
- entre
- obrigatório
- biotecnologia
- Azul
- construir
- mas a
- by
- CAN
- Câncer
- desenho animado
- casas
- casos
- celebrado
- cadeia
- desafios
- acusações
- verificar
- classe
- Na nuvem
- adoção de nuvem
- código
- cor
- vem
- comentar
- comum
- comparar
- comparando
- completar
- confiança
- Recipiente
- contraste
- corresponde
- relação custo-benefício
- custos
- poderia
- crio
- criado
- crítico
- Clientes
- dados,
- bases de dados
- datetime
- profundo
- deep learning
- DeepMind
- Grau
- demandas
- Dependendo
- implantar
- implantado
- Implantação
- desenvolvimento
- Determinar
- determinado
- desenvolvido
- em desenvolvimento
- Desenvolvimento
- dispositivo
- difícil
- Ecrã
- do
- parece
- Não faz
- download
- distância
- Drogas
- durante
- cada
- fácil
- efeito
- efeitos
- permite
- final
- Ponto final
- assegurando
- Meio Ambiente
- exemplo
- exemplos
- caro
- vasta experiência
- experimentos
- externo
- extremamente
- Rosto
- família
- mais rápido
- poucos
- menos
- campo
- Figura
- Envie o
- Arquivos
- Encontre
- final
- Primeiro nome
- seguinte
- Escolha
- formato
- amigos
- da
- função
- mais distante
- gerar
- gerado
- GitHub
- Global
- Verde
- orientações
- Ter
- he
- saúde
- pesado
- ajudar
- ajuda
- Alta
- sua
- hospedeiro
- Como funciona o dobrador de carta de canal
- Como Negociar
- Contudo
- HTML
- HTTPS
- Hub
- Abraçando o Rosto
- idêntico
- Identidade
- if
- imagem
- importar
- importante
- melhorar
- melhorado
- in
- inclui
- indicam
- indicam
- INFORMAÇÕES
- inovar
- entrada
- introspecção
- insights
- instância
- em vez disso
- integrar
- interagir
- interações
- interativo
- interage
- para dentro
- IT
- ESTÁ
- laboratório
- língua
- grande
- conduzir
- aprendizagem
- Deixar
- bibliotecas
- Biblioteca
- vida
- Ciências da Vida
- leve
- como
- Provável
- carregar
- localização
- pesquisa
- máquina
- aprendizado de máquina
- fazer
- FAZ
- Gerente
- muitos
- Match
- Posso..
- medicina
- Conheça
- método
- métodos
- métrico
- Michigan
- milhões
- Minutos
- ML
- MLOps
- Moda
- modelo
- modelos
- modificações
- molecular
- mais
- muito
- nome
- necessário
- você merece...
- NIH
- caderno
- romance
- número
- objetos
- obsoleto
- of
- on
- ONE
- só
- opera
- otimizando
- Opção
- or
- Laranja
- OS
- Outros
- A Nossa
- saída
- lado de fora
- parâmetros
- apaixonado
- paciente
- pacientes
- padrões
- perfeita
- atuação
- oleoduto
- platão
- Inteligência de Dados Platão
- PlatãoData
- por favor
- Publique
- potencialmente
- predizer
- previsto
- prevendo
- predição
- Previsões
- Preparar
- anteriormente
- processo
- processos
- Proteína
- Proteínas
- fornecer
- fornece
- fornecendo
- Python
- pytorch
- Qi
- rapidamente
- rapidamente
- campo em rápida evolução
- em tempo real
- recomendar
- Vermelho
- região
- relacionado
- depender
- representar
- representando
- representa
- requerer
- pesquisa
- pesquisadores
- resultar
- LINHA
- Execute
- corrida
- sábio
- Inferência do SageMaker
- mesmo
- Salvar
- escalável
- dimensionamento
- Ciência
- CIÊNCIAS
- Ponto
- marcar
- Scripts
- segundo
- seguro
- Vejo
- senior
- Seqüência
- Serviços
- conjunto
- vários
- Partilhar
- ela
- rede de apoio social
- mostrar
- mostrando
- lado
- semelhante
- simplificar
- solteiro
- solução
- Soluções
- RESOLVER
- alguns
- Espacial
- especialista
- Espectroscopia
- velocidade
- começo
- estado-da-arte
- Passo
- estrutural
- estrutura
- estudo
- Estudo
- tal
- adequado
- ajuda
- sobrevivência
- Tire
- toma
- tem como alvo
- tarefas
- Profissionais
- equipes
- Dados Técnicos:
- teste
- do que
- que
- A
- deles
- Eles
- então
- Lá.
- Este
- isto
- três
- tridimensional
- tempo
- demorado
- vezes
- para
- tokenize
- tokenized
- ferramenta
- ferramentas
- tocha
- treinado
- Training
- transformadores
- transporte
- tratamento
- dois
- tipo
- tipos
- compreender
- compreensão
- universidade
- ao contrário
- us
- usar
- usava
- utilização
- VALIDAR
- muito
- Ver
- visualização
- W
- querido
- Caminho..
- we
- web
- serviços web
- bem conhecido
- O Quê
- Wikipedia
- precisarão
- de
- WordPress
- Atividades:
- trabalho
- seria
- x-ray
- Vocês
- investimentos
- zefirnet