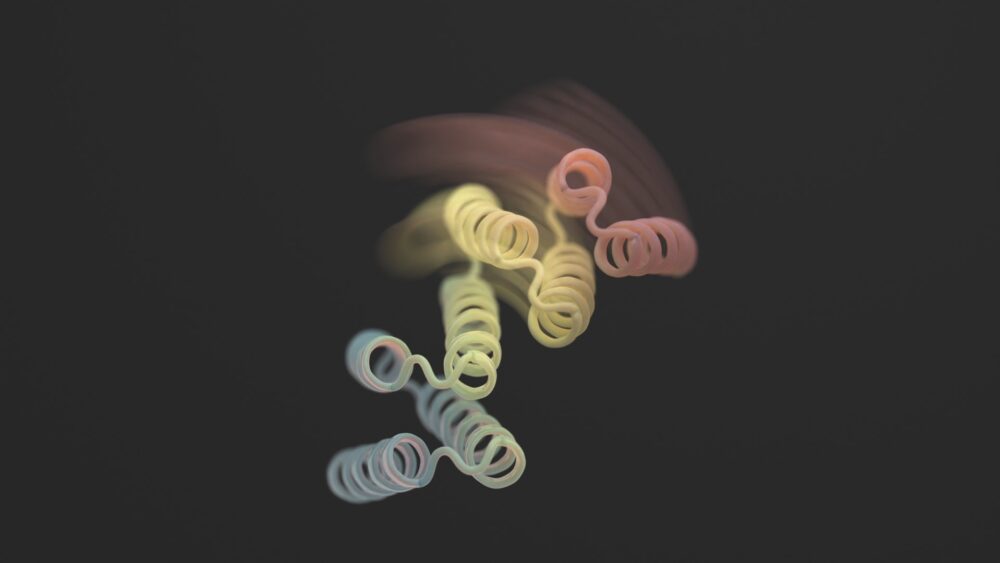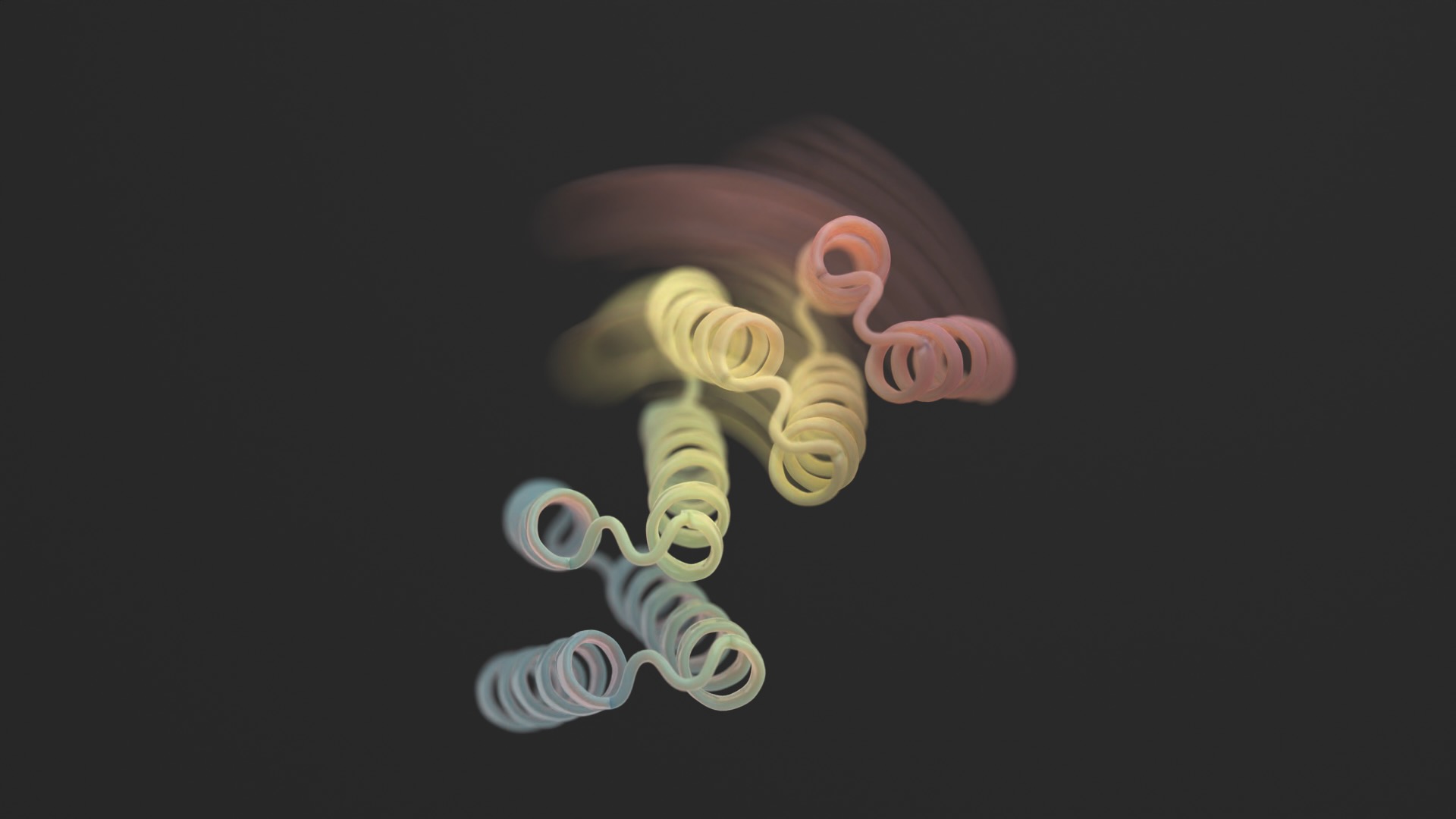
Muitas vezes pensamos nas proteínas como esculturas 3D imutáveis.
Isto não está completamente correto. Muitas proteínas são transformadores que se torcem e mudam de formato dependendo das necessidades biológicas. Uma configuração pode propagar sinais prejudiciais de um acidente vascular cerebral ou ataque cardíaco. Outro pode bloquear a cascata molecular resultante e limitar os danos.
De certa forma, as proteínas agem como transistores biológicos – interruptores liga-desliga na raiz do “computador” molecular do corpo, determinando como ele reage às forças externas e internas e ao feedback. Os cientistas há muito estudam essas proteínas que mudam de forma para decifrar como nosso corpo funciona.
Mas por que confiar apenas na natureza? Podemos criar “transistores” biológicos, desconhecidos do universo biológico, do zero?
Entre na IA. Vários métodos de aprendizagem profunda já podem prever com precisão as estruturas das proteínas -um avanço de meio século em formação. Estudos subsequentes usando algoritmos cada vez mais poderosos alucinaram estruturas proteicas livres das forças da evolução.
No entanto, estas estruturas geradas pela IA têm uma desvantagem: embora altamente complexas, a maioria é completamente estática – essencialmente, uma espécie de escultura digital de proteína congelada no tempo.
Um novo estudo in Ciência este mês quebrou o padrão ao adicionar flexibilidade às proteínas projetadas. As novas estruturas não são contorcionistas sem limites. No entanto, as proteínas projetadas podem se estabilizar em duas formas diferentes – pense em uma dobradiça em uma configuração aberta ou fechada – dependendo de uma “fechadura” biológica externa. Cada estado é análogo ao “0” ou “1” de um computador, que subsequentemente controla a saída da célula.
“Antes, só podíamos criar proteínas que tivessem uma configuração estável”, disse o autor do estudo, Dr. Florian Praetorius, da Universidade de Washington. “Agora podemos finalmente criar proteínas que se movem, o que deverá abrir uma gama extraordinária de aplicações.”
O autor principal, Dr. David Baker, tem ideias: “Desde a formação de nanoestruturas que respondem a produtos químicos no ambiente até aplicações na distribuição de medicamentos, estamos apenas começando a explorar seu potencial”.
Um casamento de proteínas feito em IA
Um pouco rápido de biologia 101.
As proteínas constroem e administram nossos corpos. Essas macromoléculas começam sua jornada a partir do DNA. A informação genética é traduzida em aminoácidos, os blocos de construção de uma proteína – contas em um cordão. Cada corda é então dobrada em formas 3D complexas, com algumas partes grudadas em outras. Chamadas de estruturas secundárias, algumas configurações parecem Twizzlers. Outros tecem lençóis semelhantes a carpetes. Essas formas se complementam, formando arquiteturas de proteínas altamente sofisticadas.
Ao compreender como as proteínas ganham a sua forma, podemos potencialmente criar novas formas a partir do zero, expandindo o universo biológico e criando novas armas contra infecções virais e outras doenças.
Em 2020, AlphaFold da DeepMind e RoseTTAFold do laboratório David Baker quebraram a internet da biologia estrutural ao prever com precisão estruturas de proteínas com base apenas em suas sequências de aminoácidos.
Desde então, os modelos de IA previram a forma de quase todas as proteínas conhecidas – e desconhecidas – pela ciência. Estas ferramentas poderosas já estão a remodelar a investigação biológica, ajudando os cientistas a identificar rapidamente alvos potenciais para combater a resistência aos antibióticos, estude o “alojamento” do nosso DNA, desenvolver novas vacinas ou até mesmo lançar luz sobre doenças que assolam o cérebro, como Doença de Parkinson.
Então veio uma bomba: modelos generativos de IA, como DALL-E e ChatGPT, ofereceram uma perspectiva tentadora. Em vez de simplesmente prever estruturas proteicas, por que não faça a IA sonhar completamente novo estruturas proteicas em vez disso? De uma proteína que se liga aos hormônios para regular os níveis de cálcio a enzimas artificiais que catalisam a bioluminescência, os resultados iniciais despertaram entusiasmo e o potencial para proteínas projetadas por IA parecia infinito.
No comando dessas descobertas está o laboratório de Baker. Pouco depois de lançar o RoseTTAFold, eles desenvolveram ainda mais o algoritmo para identificar locais funcionais em uma proteína – onde ela interage com outras proteínas, medicamentos ou anticorpos – abrindo caminho para que os cientistas sonhar novos medicamentos que ainda não imaginaram.
No entanto, faltava uma coisa: flexibilidade. Um grande número de proteínas “muda o código” de forma para alterar a sua mensagem biológica. O resultado poderia ser literalmente vida ou morte: uma proteína chamada Bax, por exemplo, altera sua forma em uma conformação que desencadeia a morte celular. A beta amilóide, uma proteína envolvida na doença de Alzheimer, notoriamente assume uma forma diferente, pois prejudica as células cerebrais.
Uma IA que alucina proteínas flip-flop semelhantes poderia levar-nos mais perto da compreensão e da recapitulação destes enigmas biológicos – levando a novas soluções médicas.
Dobradiça, linha e chumbada
Projetar uma proteína em nível atômico – e esperar que ela funcione em uma célula viva – é difícil. Projetar um com duas configurações é um pesadelo.
Como uma analogia vaga, pense em cristais de gelo em uma nuvem que eventualmente se transformam em flocos de neve, cada um com estrutura diferente. O trabalho da IA é produzir proteínas que possam alternar entre dois “flocos de neve” diferentes usando os mesmos “cristais de gelo” de aminoácidos, com cada estado correspondendo a um botão “ligado” ou “desligado”. Além disso, a proteína tem que funcionar bem dentro das células vivas.
A equipe começou com várias regras. Primeiro, cada estrutura deve parecer muito diferente entre os dois estados – como um perfil humano em pé ou sentado. Eles poderiam verificar isso medindo as distâncias entre os átomos, explicou a equipe. Em segundo lugar, a mudança precisa acontecer rapidamente. Isso significa que a proteína não pode se desenrolar completamente antes de se recompor em outra forma, o que leva tempo.
Depois, há algumas diretrizes básicas para uma proteína funcional: ela precisa funcionar bem com os líquidos corporais em ambos os estados. Finalmente, deve atuar como um interruptor, mudando sua forma dependendo das entradas e saídas.
Reunir todas “essas propriedades em um sistema proteico é um desafio”, disse a equipe.
Usando uma mistura de AlphaFold, Rosetta e proteinMPNN, o design final parece uma dobradiça. Possui duas partes rígidas que podem se mover uma em relação à outra, enquanto outra peça permanece dobrada. Normalmente a proteína está fechada. O gatilho é um pequeno peptídeo – uma cadeia curta de aminoácidos – que se liga às dobradiças e desencadeia sua mudança de forma. Esses chamados “peptídeos efetores” foram cuidadosamente projetados para serem específicos, diminuindo suas chances de se agarrarem a partes fora do alvo.
A equipe primeiro adicionou peptídeos desencadeadores que brilham no escuro a vários designs de dobradiças. A análise subsequente descobriu que o gatilho agarrou-se facilmente à dobradiça. A configuração da proteína mudou. Como verificação de sanidade, a forma foi previamente prevista usando análise de IA.
Estudos adicionais utilizando estruturas cristalizadas dos desenhos de proteínas, com ou sem o efetor, validaram ainda mais os resultados. Esses testes também buscaram princípios de design que faziam a dobradiça funcionar e parâmetros que direcionavam um estado para o outro.
A conclusão? A IA agora pode projetar proteínas com dois estados diferentes – essencialmente construindo transistores biológicos para biologia sintética. Por enquanto, o sistema utiliza apenas peptídeos efetores personalizados em seus estudos, o que pode limitar a pesquisa e o potencial clínico. Mas, segundo a equipe, a estratégia também pode se estender a peptídeos naturais, como aqueles que se ligam a proteínas envolvidas na regulação do açúcar no sangue, regulam a água nos tecidos ou influenciam a atividade cerebral.
“Como os transistores em circuitos eletrônicos, podemos acoplar os interruptores a saídas e entradas externas para criar dispositivos sensores e incorporá-los em sistemas de proteínas maiores”, disse a equipe.
O autor do estudo, Dr. Philip Leung, acrescenta: “Isso poderia revolucionar a biotecnologia da mesma forma que os transistores transformaram a eletrônica”.
Crédito da imagem: Ian C Haydon/Instituto UW de Design de Proteínas
- Conteúdo com tecnologia de SEO e distribuição de relações públicas. Seja amplificado hoje.
- PlatoData.Network Gerativa Vertical Ai. Capacite-se. Acesse aqui.
- PlatoAiStream. Inteligência Web3. Conhecimento Amplificado. Acesse aqui.
- PlatãoESG. Automotivo / EVs, Carbono Tecnologia Limpa, Energia, Ambiente, Solar, Gestão de resíduos. Acesse aqui.
- PlatoHealth. Inteligência em Biotecnologia e Ensaios Clínicos. Acesse aqui.
- ChartPrime. Eleve seu jogo de negociação com ChartPrime. Acesse aqui.
- BlockOffsets. Modernizando a Propriedade de Compensação Ambiental. Acesse aqui.
- Fonte: https://singularityhub.com/2023/08/22/ai-can-now-design-proteins-that-behave-like-biological-transistors/
- :tem
- :é
- :não
- $UP
- 2020
- 3d
- a
- AC
- Segundo
- exatamente
- Aja
- atividade
- adicionado
- acrescentando
- Adicionalmente
- Adiciona
- Depois de
- contra
- AI
- Modelos de IA
- algoritmo
- algoritmos
- Todos os Produtos
- sozinho
- já
- tb
- Apesar
- A doença de Alzheimer
- an
- análise
- e
- Outro
- aplicações
- SOMOS
- AS
- At
- ataque
- autor
- longe
- em caminho duplo
- padeiro
- baseado
- BE
- antes
- começou
- começar
- beta
- entre
- vincular
- biologia
- biotecnologia
- Pouco
- Bloquear
- Blocos
- sangue
- corpos
- ambos
- Cérebro
- Atividade cerebral
- Células cerebrais
- avanço
- Quebrou
- construir
- Prédio
- mas a
- by
- Cálcio
- chamado
- veio
- CAN
- cuidadosamente
- cachoeiras
- catalisar
- Células
- Century
- cadeia
- desafiante
- chances
- alterar
- mudado
- mudança
- ChatGPT
- verificar
- Clínico
- fechado
- mais próximo
- Na nuvem
- completamente
- Configuração
- controles
- Correspondente
- poderia
- Casal
- crio
- Criar
- crédito
- dall's
- danificar
- David
- Morte
- Decifrar
- profundo
- deep learning
- DeepMind
- Entrega
- Dependendo
- Design
- princípios de design
- projetado
- Designer
- concepção
- projetos
- determinando
- desenvolvido
- Dispositivos/Instrumentos
- diferente
- digital
- Doença
- doenças
- dna
- down
- queda
- dr
- seu "Sonho"
- droga
- Drogas
- cada
- facilmente
- borda
- ou
- Eletrônico
- Eletrônicos
- Interminável
- engenheiro
- entusiasmo
- Meio Ambiente
- Mesmo
- eventualmente
- Cada
- evolução
- exemplo
- expansão
- explicado
- estender
- externo
- extraordinário
- RÁPIDO
- retornos
- final
- Finalmente
- Primeiro nome
- Flexibilidade
- Escolha
- Forças
- formulário
- encontrado
- da
- congelado
- função
- funcional
- mais distante
- Ganho
- generativo
- IA generativa
- orientações
- tinha
- Metade
- acontecer
- Queijos duros
- prejudicar
- prejudica
- Ter
- Coração
- Heart Attack
- ajuda
- altamente
- Dobradiça
- esperando
- Como funciona o dobrador de carta de canal
- Contudo
- HTTPS
- humano
- ICE
- idéias
- imaginado
- imutável
- in
- incorporar
- cada vez mais
- infecções
- influência
- INFORMAÇÕES
- do estado inicial,
- inputs
- dentro
- em vez disso
- Instituto
- interage
- interno
- Internet
- para dentro
- envolvido
- IT
- ESTÁ
- se
- Trabalho
- viagem
- apenas por
- laboratório
- grande
- Maior
- aprendizagem
- níveis
- vida
- leve
- como
- LIMITE
- limites
- Line
- vida
- longo
- olhar
- parece
- OLHARES
- Baixa
- moldadas
- fazer
- muitos
- Posso..
- significa
- medição
- médico
- mensagem
- métodos
- desaparecido
- misturar
- modelos
- molecular
- Mês
- a maioria
- mover
- múltiplo
- natural
- Natureza
- Cria
- Novo
- agradável
- NIH
- normalmente
- agora
- número
- of
- oferecido
- frequentemente
- on
- ONE
- queridos
- só
- para
- aberto
- or
- Outros
- Outros
- A Nossa
- saída
- parâmetros
- peças
- peça
- platão
- Inteligência de Dados Platão
- PlatãoData
- Jogar
- potencial
- potencialmente
- poderoso
- predizer
- previsto
- prevendo
- anteriormente
- princípios
- Perfil
- Propriedades
- prospecto
- Proteína
- Proteínas
- Links
- rapidamente
- alcance
- em vez
- Reage
- Regular
- regulando
- relativo
- liberação
- depender
- permanece
- pesquisa
- Responder
- resultar
- resultando
- Resultados
- revolucionar
- certo
- rígido
- raiz
- regras
- Execute
- Dito
- mesmo
- Ciência
- cientistas
- arranhar
- Segundo
- secundário
- parecia
- vários
- Shape
- formas
- derramar
- mudança
- Baixo
- Em breve
- rede de apoio social
- sinais
- semelhante
- simplesmente
- Locais
- Sentado
- pequeno
- flocos de neve
- Soluções
- alguns
- sofisticado
- deflagrou
- especificidade
- estabilizar
- estável
- Comece
- Estado
- Unidos
- aderindo
- Estratégia
- Tanga
- estrutural
- estrutura
- estudado
- caso
- Estudo
- subseqüente
- Subseqüentemente
- tal
- açúcar
- Interruptor
- sintético
- .
- sistemas
- Tire
- toma
- Torneira
- tem como alvo
- Profissionais
- testes
- do que
- que
- A
- deles
- Eles
- então
- Lá.
- Este
- deles
- coisa
- think
- isto
- aqueles
- tempo
- tipo
- para
- juntos
- ferramentas
- transformado
- transformadores
- desencadear
- torção
- dois
- compreensão
- Universo
- universidade
- desconhecido
- us
- usos
- utilização
- validado
- muito
- viral
- foi
- Washington
- Água
- Caminho..
- we
- armas
- Tecer
- web
- foram
- qual
- enquanto
- porque
- de
- sem
- Atividades:
- trabalho
- ainda
- zefirnet