A transcriptômica espacial e a proteômica fornecem informações complementares que transformaram nossa compreensão de processos biológicos complexos. No entanto, a integração experimental destas modalidades é limitada.
Um novo método chamado Spatial Protein and Transcriptome Sequencing (SPOTS) pode iluminar as identidades e atividades das células em um órgão ou tumor com resolução sem precedentes. Desenvolvido por pesquisadores da Weill Cornell Medicine, NewYork-Presbyterian e New York Genome Center, o SPOTS pode iluminar as identidades e atividades das células em um órgão ou tumor com resolução sem precedentes.
A técnica preserva informações sobre a localização precisa das células enquanto registra padrões de atividade genética e a presença de substâncias essenciais. proteínas em células em amostras de tecido. Isto torna possível criar “mapas” de órgãos complexos e ricos em dados, incluindo órgãos doentes e tumores, que podem ser extremamente úteis tanto na investigação básica como na clínica.
O co-autor sênior do estudo, Dr. Dan Landau, professor associado de medicina na Divisão de Hematologia e Oncologia Médica e membro do Sandra and Edward Meyer Cancer Center da Weill Cornell Medicine e membro do corpo docente do New York Genome Center, disse, “Esta tecnologia é interessante porque nos permite mapear a organização espacial dos tecidos, incluindo tipos de células, atividades celulares e interações entre células, como nunca antes.”
A nova abordagem faz parte de uma iniciativa mais ampla de cientistas e engenheiros para criar meios mais eficazes de “ver” ao nível microscópico como funcionam os órgãos e tecidos. Nos últimos anos, assistimos a avanços significativos na investigação, nomeadamente em métodos para traçar perfis de atividade genética e outras camadas de dados em células individuais ou em pequenos grupos de células. células. A informação relativa às colocações originais das células perfiladas dentro dos tecidos precisa de ser recuperada, no entanto, porque estas abordagens pedem frequentemente a quebra dos tecidos e a separação das células dos seus vizinhos. A nova técnica também registra esses dados espaciais, e o faz com excelente resolução.
O método é baseado em parte na tecnologia 10x Genomics existente. Ele usa lâminas de vidro adequadas para imagens de amostras de tecido com métodos patológicos comuns baseados em microscópio, mas também é revestida com milhares de moléculas de sonda específicas.
O “código de barras” químico de cada molécula da sonda identifica sua localização bidimensional na lâmina. As moléculas da sonda na lâmina agarram o RNA mensageiro (mRNAs), essencialmente os transcritos de genes ativados, de células vizinhas, quando uma amostra de tecido em fatias finas é colocada na lâmina, suas células se tornam permeáveis. Anticorpos projetados são usados no procedimento e se ligam às moléculas e proteínas únicas da sonda de interesse no tecido.
Os pesquisadores podem identificar rápida e automaticamente os mRNAs coletados e as proteínas escolhidas e mapeá-los com precisão em seus locais originais em toda a amostra de tecido. Os mapas produzidos podem ser considerados de forma independente ou em comparação com as imagens patológicas de rotina da amostra.
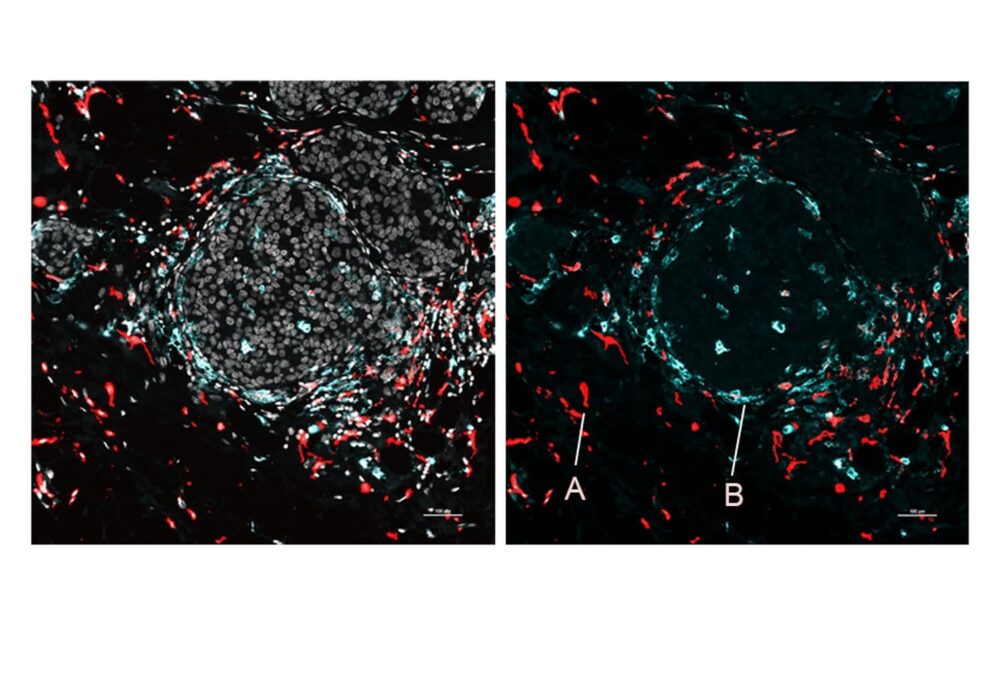
No tecido de um baço de rato saudável, a equipe usou SPOTS para mostrar a intrincada arquitetura funcional deste órgão, incluindo agrupamentos de vários tipos de células, seus estados funcionais e como esses estados se alteravam dependendo da localização das células.
Os pesquisadores também utilizaram o SPOTS para mapear a estrutura celular de um tumor de mama de camundongo, destacando seu potencial para uso em pesquisa sobre o câncer. O mapa gerado mostrou macrófagos e células imunológicas em dois estados diferentes, cada um indicado por um marcador proteico diferente: uma condição estava ativa e lutando contra tumores, enquanto a outra era imunossupressora e estabelecendo uma barreira para proteger o tumor.
Landau, oncologista do NewYork-Presbyterian/Weill Cornell Medical Center, dito, “Pudemos ver que esses dois subconjuntos de macrófagos são encontrados em diferentes áreas do tumor e interagem com células diferentes – e que a diferença no microambiente provavelmente está impulsionando seus estados de atividade distintos”.
“Tais detalhes do ambiente imunológico do tumor – detalhes que muitas vezes não podem ser resolvidos devido à escassez de células imunológicas dentro dos tumores – podem ajudar a explicar por que alguns pacientes respondem a terapia de reforço imunológico e alguns não, e assim poderiam informar o projeto de imunoterapias futuras”.
“Esta versão inicial do SPOTS tem uma resolução espacial tal que cada “pixel” do conjunto de dados resultante soma informações de atividade genética para pelo menos várias células. No entanto, os pesquisadores esperam em breve restringir esta resolução a células únicas, ao mesmo tempo que adicionam outras camadas de informações celulares importantes.”
Jornal de referência:
- Ben-Chetrit, N., Niu, X., Swett, AD et al. Integração do perfil espacial do transcriptoma completo com marcadores proteicos. Nat Biotecnologia (2023). DOI: 10.1038/s41587-022-01536-3