Белки управляют многими биологическими процессами, такими как активность ферментов, молекулярный транспорт и клеточная поддержка. Трехмерная структура белка дает представление о его функции и о том, как он взаимодействует с другими биомолекулами. Экспериментальные методы определения структуры белка, такие как рентгеновская кристаллография и ЯМР-спектроскопия, дороги и требуют много времени.
Напротив, недавно разработанные вычислительные методы могут быстро и точно предсказать структуру белка по его аминокислотной последовательности. Эти методы имеют решающее значение для белков, которые трудно изучать экспериментально, таких как мембранные белки, мишени для многих лекарств. Одним из известных примеров этого является AlphaFold, алгоритм на основе глубокого обучения, известный своими точными прогнозами.
ESMFold — еще один высокоточный метод, основанный на глубоком обучении, разработанный для предсказания структуры белка по его аминокислотной последовательности. ESMFold использует модель большого белкового языка (pLM) в качестве основы и работает от начала до конца. В отличие от AlphaFold2, он не требует поиска или Выравнивание нескольких последовательностей (MSA), а также не использует внешние базы данных для создания прогнозов. Вместо этого команда разработчиков обучила модель миллионам белковых последовательностей из UniRef. Во время обучения модель выработала паттерны внимания, которые изящно представляют эволюционные взаимодействия между аминокислотами в последовательности. Такое использование PLM вместо MSA позволяет в 60 раз сократить время прогнозирования по сравнению с другими современными моделями.
В этом посте мы используем предварительно обученную модель ESMFold из Hugging Face с Создатель мудреца Амазонки предсказать структуру тяжелой цепи трастузумаб, чтобы моноклональное антитело впервые разработан Genentech для лечения HER2-положительный рак груди. Быстрое предсказание структуры этого белка может оказаться полезным, если исследователи захотят проверить влияние модификаций последовательности. Это потенциально может привести к улучшению выживаемости пациентов или снижению побочных эффектов.
В этом посте приведен пример блокнота Jupyter и связанных с ним скриптов. Репозиторий GitHub.
Предпосылки
Мы рекомендуем запустить этот пример в Студия Amazon SageMaker ноутбук запуск образа PyTorch 1.13 Python 3.9, оптимизированного для ЦП, на экземпляре типа ml.r5.xlarge.
Визуализируйте экспериментальную структуру трастузумаба
Для начала воспользуемся biopython библиотека и вспомогательный скрипт для загрузки структуры трастузумаба из Банк белковых данных RCSB:
Далее мы используем py3Dmol библиотека для визуализации конструкции в виде интерактивной 3D-визуализации:
На следующем рисунке представлена трехмерная структура белка 3N1Z из банка данных белков (PDB). На этом изображении легкая цепь трастузумаба отображается оранжевым цветом, тяжелая цепь — синим (с вариабельной областью — светло-голубым), а антиген HER8 — зеленым.
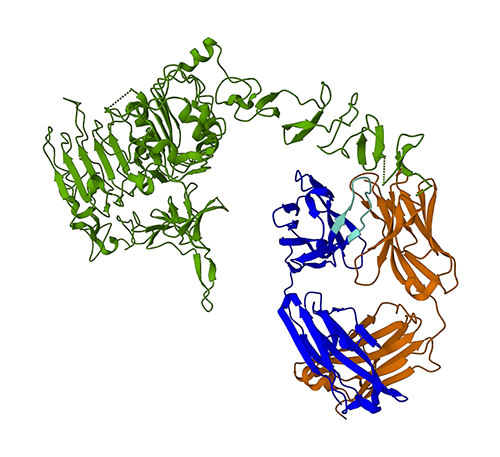
Сначала мы воспользуемся ESMFold, чтобы предсказать структуру тяжелой цепи (цепь B) по ее аминокислотной последовательности. Затем мы сравним предсказание с экспериментально определенной структурой, показанной выше.
Предсказать структуру тяжелой цепи трастузумаба по ее последовательности с помощью ESMFold
Давайте воспользуемся моделью ESMFold для предсказания структуры тяжелой цепи и сравним ее с экспериментальным результатом. Для начала мы будем использовать предварительно созданную среду блокнота в Studio, которая поставляется с несколькими важными библиотеками, такими как PyTorch, предварительно установлен. Хотя мы могли бы использовать ускоренный тип экземпляра для повышения производительности нашего анализа записной книжки, вместо этого мы будем использовать неускоренный экземпляр и запустим прогноз ESMFold на ЦП.
Сначала мы загружаем предварительно обученную модель ESMFold и токенизатор из Обниматься Face Hub:
Затем мы копируем модель на наше устройство (в данном случае ЦП) и устанавливаем некоторые параметры модели:
Чтобы подготовить последовательность белка для анализа, нам нужно ее токенизировать. Это переводит символы аминокислот (EVQLV…) в числовой формат, понятный модели ESMFold (6,19,5,10,19,…):
Затем мы копируем токенизированный ввод в режим, делаем прогноз и сохраняем результат в файл:
Это занимает около 3 минут на неускоренном типе инстанса, таком как r5.
Мы можем проверить точность прогноза ESMFold, сравнив его с экспериментальной структурой. Делаем это с помощью выравнивание по США инструмент, разработанный Zhang Lab в Мичиганском университете:
| PDBchain1 | PDBchain2 | ТМ-оценка |
| данные/прогноз.pdb:A | данные/экспериментальные.pdb:B | 0.802 |
Ассоциация оценка моделирования шаблона (TM-оценка) представляет собой метрику для оценки сходства белковых структур. Оценка 1.0 указывает на идеальное совпадение. Баллы выше 0.7 указывают на то, что белки имеют одинаковую структуру скелета. Баллы выше 0.9 указывают на то, что белки функционально взаимозаменяемый для последующего использования. В нашем случае достижения TM-Score 0.802 предсказание ESMFold, вероятно, будет подходящим для таких приложений, как оценка структуры или эксперименты по связыванию лиганда, но может не подходить для таких случаев использования, как молекулярная замена которые требуют чрезвычайно высокой точности.
Мы можем проверить этот результат, визуализируя выровненные структуры. Две структуры демонстрируют высокую, но не идеальную степень перекрытия. Прогнозирование структуры белков — это быстро развивающаяся область, и многие исследовательские группы разрабатывают все более точные алгоритмы!
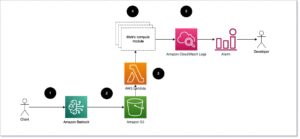
Разверните ESMFold в качестве конечной точки логического вывода SageMaker.
Вывод модели в записной книжке подходит для экспериментов, но что, если вам нужно интегрировать модель с приложением? Или конвейер MLOps? В этом случае лучшим вариантом является развертывание вашей модели в качестве конечной точки вывода. В следующем примере мы развернем ESMFold в качестве конечной точки логического вывода SageMaker в режиме реального времени на ускоренном экземпляре. Конечные точки SageMaker в режиме реального времени обеспечивают масштабируемый, экономичный и безопасный способ развертывания и размещения моделей машинного обучения (ML). Благодаря автоматическому масштабированию вы можете настроить количество экземпляров, на которых работает конечная точка, в соответствии с требованиями вашего приложения, оптимизируя расходы и обеспечивая высокую доступность.
Предварительно построенный Контейнер SageMaker для Hugging Face упрощает развертывание моделей глубокого обучения для общих задач. Однако для новых случаев использования, таких как предсказание структуры белка, нам нужно определить пользовательскую inference.py скрипт для загрузки модели, запуска прогноза и форматирования вывода. Этот сценарий включает в себя большую часть того же кода, который мы использовали в нашей записной книжке. Мы также создаем requirements.txt файл, чтобы определить некоторые зависимости Python для нашей конечной точки. Вы можете увидеть файлы, которые мы создали в Репозиторий GitHub.
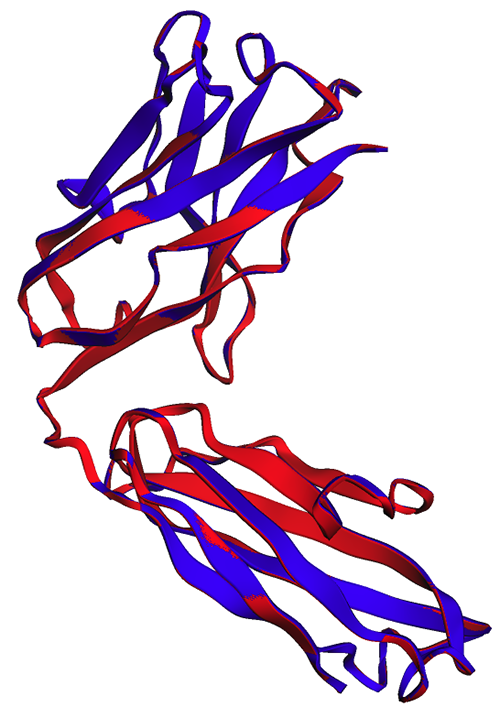
На следующем рисунке экспериментальная (синяя) и предполагаемая (красная) структуры тяжелой цепи трастузумаба очень похожи, но не идентичны.

После того, как мы создали необходимые файлы в code каталог, мы развертываем нашу модель с помощью SageMaker HuggingFaceModel сорт. При этом используется предварительно созданный контейнер для упрощения процесса развертывания моделей Hugging Face в SageMaker. Обратите внимание, что создание конечной точки может занять 10 минут или больше, в зависимости от доступности ml.g4dn типы экземпляров в нашем регионе.
Когда развертывание конечной точки завершено, мы можем повторно отправить последовательность белка и отобразить первые несколько строк прогноза:
Поскольку мы развернули нашу конечную точку на ускоренном экземпляре, прогноз должен занять всего несколько секунд. Каждая строка в результате соответствует одному атому и включает в себя идентичность аминокислоты, три пространственные координаты и оценка pLDDT представляющий достоверность прогноза в этом месте.
| PDB_GROUP | ID | ATOM_LABEL | РЕС_ИД | CHAIN_ID | SEQ_ID | КАРТН_X | КАРТН_Y | КАРТН_Z | ЗАНЯТИЕ | ПЛДДТ | АТОМ_ИД |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
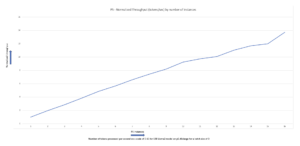
Используя тот же метод, что и раньше, мы видим, что предсказания ноутбука и конечной точки идентичны.
| PDBchain1 | PDBchain2 | ТМ-оценка |
| данные/endpoint_prediction.pdb:A | данные/прогноз.pdb:A | 1.0 |
Как видно на следующем рисунке, прогнозы ESMFold, сгенерированные в ноутбуке (красный) и по конечной точке (синий), показывают идеальное выравнивание.
Убирать
Чтобы избежать дополнительных расходов, мы удаляем нашу конечную точку вывода и тестовые данные:
Обзор
Компьютерное предсказание структуры белка является важным инструментом для понимания функции белков. Помимо фундаментальных исследований, такие алгоритмы, как AlphaFold и ESMFold, имеют множество применений в медицине и биотехнологии. Структурные идеи, полученные с помощью этих моделей, помогают нам лучше понять, как взаимодействуют биомолекулы. Затем это может привести к созданию более совершенных диагностических инструментов и методов лечения пациентов.
В этом посте мы покажем, как развернуть языковую модель белка ESMFold из Hugging Face Hub в качестве масштабируемой конечной точки логического вывода с помощью SageMaker. Дополнительные сведения о развертывании моделей Hugging Face в SageMaker см. Используйте обнимающее лицо с Amazon SageMaker. Вы также можете найти больше примеров из науки о белках в Потрясающий анализ белков на AWS Репозиторий GitHub. Пожалуйста, оставьте нам комментарий, если есть другие примеры, которые вы хотели бы увидеть!
Об авторах
 Брайан Лоял является старшим архитектором решений AI/ML в глобальной команде здравоохранения и медико-биологических наук в Amazon Web Services. У него более 17 лет опыта работы в области биотехнологий и машинного обучения, и он увлечен тем, что помогает клиентам решать геномные и протеомные задачи. В свободное время он любит готовить и есть с друзьями и семьей.
Брайан Лоял является старшим архитектором решений AI/ML в глобальной команде здравоохранения и медико-биологических наук в Amazon Web Services. У него более 17 лет опыта работы в области биотехнологий и машинного обучения, и он увлечен тем, что помогает клиентам решать геномные и протеомные задачи. В свободное время он любит готовить и есть с друзьями и семьей.
 Шамика Арияванса является архитектором решений специалиста по искусственному интеллекту и машинному обучению в глобальной команде здравоохранения и медико-биологических наук в Amazon Web Services. Он увлеченно работает с клиентами, чтобы ускорить внедрение ими искусственного интеллекта и машинного обучения, предоставляя технические рекомендации и помогая им внедрять инновации и создавать безопасные облачные решения на AWS. Вне работы он любит кататься на лыжах и бездорожье.
Шамика Арияванса является архитектором решений специалиста по искусственному интеллекту и машинному обучению в глобальной команде здравоохранения и медико-биологических наук в Amazon Web Services. Он увлеченно работает с клиентами, чтобы ускорить внедрение ими искусственного интеллекта и машинного обучения, предоставляя технические рекомендации и помогая им внедрять инновации и создавать безопасные облачные решения на AWS. Вне работы он любит кататься на лыжах и бездорожье.
 Янцзюнь Ци является старшим менеджером по прикладным наукам в лаборатории решений AWS для машинного обучения. Она внедряет инновации и применяет машинное обучение, чтобы помочь клиентам AWS ускорить внедрение ИИ и облачных технологий.
Янцзюнь Ци является старшим менеджером по прикладным наукам в лаборатории решений AWS для машинного обучения. Она внедряет инновации и применяет машинное обучение, чтобы помочь клиентам AWS ускорить внедрение ИИ и облачных технологий.
- SEO-контент и PR-распределение. Получите усиление сегодня.
- ПлатонАйСтрим. Анализ данных Web3. Расширение знаний. Доступ здесь.
- Чеканка будущего с Эдриенн Эшли. Доступ здесь.
- Покупайте и продавайте акции компаний PREIPO® с помощью PREIPO®. Доступ здесь.
- Источник: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :имеет
- :является
- :нет
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- О нас
- выше
- AC
- ускорять
- ускоренный
- точность
- точный
- точно
- достижение
- деятельность
- дополнение
- Принятие
- AI
- AI / ML
- алгоритм
- алгоритмы
- выровненный
- выравнивание
- причислены
- Несмотря на то, что
- Amazon
- Создатель мудреца Амазонки
- Amazon Web Services
- an
- анализ
- и
- Другой
- любой
- Применение
- Приложения
- прикладной
- соответствующий
- МЫ
- AS
- Оценка
- At
- атом
- внимание
- Автоматический
- свободных мест
- избежать
- AWS
- Машинное обучение AWS
- Позвоночник
- Банка
- основной
- BE
- до
- начинать
- Лучшая
- между
- переплет
- биотехнология
- Синии
- строить
- но
- by
- CAN
- рак
- мультфильм
- случаев
- случаев
- отмечается
- цепь
- проблемы
- расходы
- проверка
- класс
- облако
- принятие облака
- код
- цвет
- выходит
- комментарий
- Общий
- сравнить
- сравнив
- полный
- доверие
- Container
- контраст
- соответствует
- рентабельным
- Расходы
- может
- Создайте
- создали
- критической
- Клиенты
- данным
- базы данных
- Дата и время
- глубоко
- глубокое обучение
- DeepMind
- Степень
- запросы
- в зависимости
- развертывание
- развернуть
- развертывание
- развертывание
- Определять
- определены
- развитый
- развивающийся
- Развитие
- устройство
- трудный
- Дисплей
- do
- приносит
- не
- скачать
- управлять
- Наркотики
- в течение
- каждый
- легко
- эффект
- эффекты
- позволяет
- конец
- Конечная точка
- обеспечение
- Окружающая среда
- пример
- Примеры
- дорогим
- опыт
- Эксперименты
- и, что лучший способ
- чрезвычайно
- Face
- семья
- быстрее
- несколько
- меньше
- поле
- фигура
- Файл
- Файлы
- Найдите
- конец
- First
- после
- Что касается
- формат
- друзья
- от
- функция
- далее
- порождать
- генерируется
- GitHub
- Глобальный
- Зелёная
- руководство
- Есть
- he
- здравоохранение
- тяжелый
- помощь
- помощь
- High
- его
- кашель
- Как
- How To
- Однако
- HTML
- HTTPS
- хаб
- ОбниматьЛицо
- идентичный
- Личность
- if
- изображение
- Импортировать
- важную
- улучшать
- улучшенный
- in
- включает в себя
- указывать
- указывает
- информация
- обновлять
- вход
- понимание
- размышления
- пример
- вместо
- интегрировать
- взаимодействовать
- взаимодействие
- интерактивный
- взаимодействует
- в
- IT
- ЕГО
- лаборатория
- язык
- большой
- вести
- изучение
- Оставлять
- библиотеки
- Библиотека
- ЖИЗНЬЮ
- Медико-биологическая промышленность
- легкий
- такое как
- Вероятно
- загрузка
- расположение
- поиск
- машина
- обучение с помощью машины
- сделать
- ДЕЛАЕТ
- менеджер
- многих
- Совпадение
- Май..
- медицина
- Встречайте
- метод
- методы
- метрический
- Мичиган
- миллионы
- Минут
- ML
- млн операций в секунду
- режим
- модель
- Модели
- изменения
- молекулярный
- БОЛЕЕ
- много
- имя
- необходимо
- Необходимость
- NIH
- ноутбук
- роман
- номер
- объекты
- устаревший
- of
- on
- ONE
- только
- работает
- оптимизирующий
- Опция
- or
- Апельсин
- OS
- Другое
- наши
- выходной
- внешнюю
- параметры
- страстный
- пациент
- пациентов
- паттеранами
- ИДЕАЛЬНОЕ
- производительность
- трубопровод
- Платон
- Платон Интеллектуальные данные
- ПлатонДанные
- пожалуйста
- После
- потенциально
- предсказывать
- предсказанный
- прогнозирования
- прогноз
- Predictions
- Подготовить
- предварительно
- процесс
- Процессы
- Белкове продукты
- Белки
- обеспечивать
- приводит
- обеспечение
- Питон
- pytorch
- Qi
- быстро
- быстро
- быстро развивающаяся область
- реального времени
- рекомендовать
- Red
- область
- Связанный
- полагаться
- представлять
- представляющий
- представляет
- требовать
- исследованиям
- исследователи
- результат
- РЯД
- Run
- Бег
- sagemaker
- Вывод SageMaker
- то же
- Сохранить
- масштабируемые
- масштабирование
- Наука
- НАУКА
- Гол
- счет
- скрипты
- секунды
- безопасный
- посмотреть
- старший
- Последовательность
- Услуги
- набор
- несколько
- Поделиться
- она
- должен
- показывать
- показанный
- сторона
- аналогичный
- упростить
- одинарной
- Решение
- Решения
- РЕШАТЬ
- некоторые
- пространственный
- специалист
- Спектроскопия
- скорость
- Начало
- современное состояние
- Шаг
- структурный
- Структура
- студия
- Кабинет
- такие
- подходящее
- поддержка
- выживание
- взять
- принимает
- направлена против
- задачи
- команда
- команды
- Технический
- тестXNUMX
- чем
- который
- Ассоциация
- их
- Их
- тогда
- Там.
- Эти
- этой
- три
- трехмерный
- время
- кропотливый
- раз
- в
- токенизировать
- лексемы
- инструментом
- инструменты
- факел
- специалистов
- Обучение
- трансформеры
- перевозки
- лечение
- два
- напишите
- Типы
- понимать
- понимание
- Университет
- В отличие от
- us
- использование
- используемый
- через
- VALIDATE
- очень
- Вид
- визуализация
- W
- стремятся
- Путь..
- we
- Web
- веб-сервисы
- известный
- Что
- Википедия.
- будете
- WordPress
- Работа
- работает
- бы
- рентгеновский
- Ты
- ВАШЕ
- зефирнет