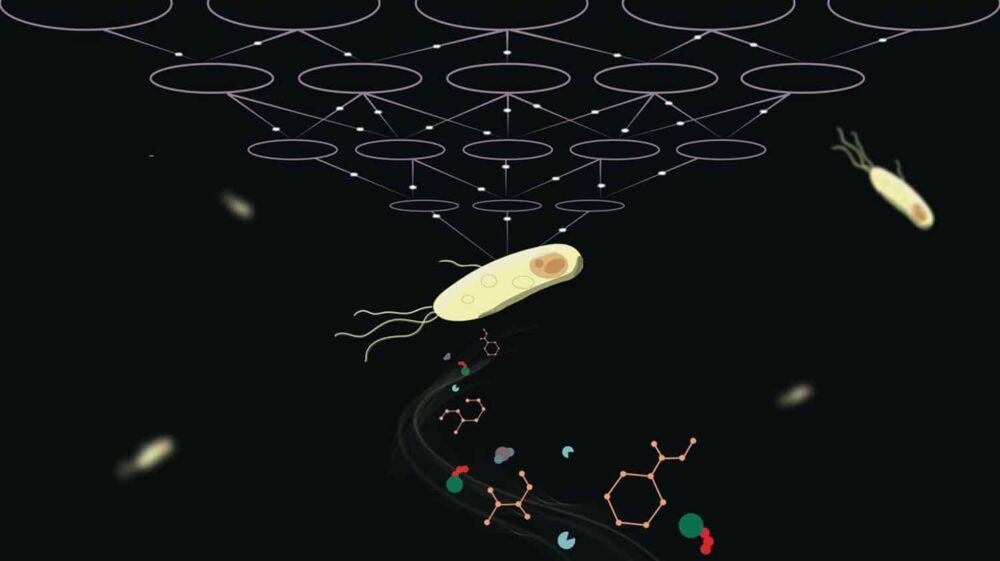
Allt levande kräver ämnesomsättning. Sättet som en organism metaboliserar näringsämnen är en komplex process, och att simulera de kemiska processer som håller livet igång är en svår utmaning.
Teoretiskt kan proceduren representeras av matematiska ekvationer med parametrar som är specifika för varje organism. Men att praktiskt bestämma dessa parametrar är dock en komplicerad sak på grund av bristen på experimentella data.
Forskare behöver i allmänhet mycket experimentell data och processorkraft för att hitta dessa parametrar. EPFL forskare föreslog ett djupinlärningsbaserat beräkningsramverk som reproducerar de dynamiska metaboliska egenskaper som observerats i celler. Ramverket kallat REKINDLE skulle kunna bana väg för mer effektiv och exakt modellering av metaboliska processer.
Ljubisa Miskovic från EPFL:s Laboratory of Computational Systems Biotechnology och co-PI i studien sa, "REKINDLE kommer att tillåta forskarvärlden att minska beräkningsansträngningarna för att generera kinetiska modeller med flera storleksordningar. Det kommer också att hjälpa till att postulera nya hypoteser genom att integrera biokemiska data i dessa modeller, belysa experimentella observationer och styra nya terapeutiska upptäckter och bioteknikdesigner."
Subham Choudhury, den första författaren till studien, sa: "Det övergripande syftet med metabolisk modellering är att beskriva cellulärt metaboliskt beteende till en sådan grad att förståelse och förutsägelse av effekterna av variationer i celltillstånd och miljöförhållanden på ett tillförlitligt sätt kan testas för ett brett spektrum av studier inom hälsa, bioteknik och system och syntetisk biologi. Vi hoppas att REKINDLE underlättar att bygga metaboliska modeller för det bredare samhället.”
Tekniken har direkta biotekniska tillämpningar eftersom kinetiska modeller är avgörande för många undersökningar, inklusive de om bioproduktion, läkemedelsinriktning, interaktioner mellan mikrober och bioremediering.
choudhury sade, “REKINDLE använder standard, ofta använda Python-bibliotek som gör det tillgängligt och enkelt att använda. Vårt huvudmål med den här studien är att bana väg för att göra den här typen av modelleringsinsatser öppen källkod och tillgängliga så att alla inom syntetiska och systembiologiska gemenskaper kan använda dem för sina egna forskningsmål, oavsett vad de kan vara.”
Tidskriftsreferens:
- Choudhury, S., Moret, M., Salvy, P. et al. Rekonstruera kinetiska modeller för dynamiska studier av metabolism med hjälp av generativa kontradiktoriska nätverk. Nat Mach Intell 4, 710–719 (2022). DOI: 10.1038 / s42256-022-00519-y