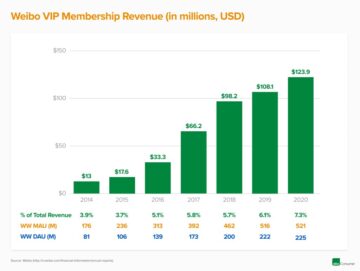
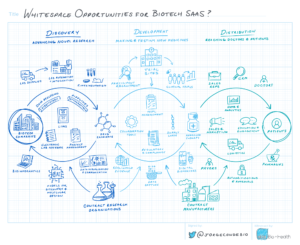
Jimmy Lin är CSO för Freenome, vilket finnas utveckla blod - baserade test för tidig cancerupptäckt som börjar med koloncancer. Han är en pionjär inom att utveckla beräkningsmetoder för att extrahera insikter från storskalig genomisk data, efter att ha lett beräkningsanalyserna av de första genomomfattande sekvenseringsstudierna i flera cancertyper.
Lin pratade med Future om utmaningarna med att utföra ett företagsuppdrag för att kombinera metoder för maskininlärning och biologisk data. Han förklarar vilka tre typer av människor du behöver anställa för att bygga ett balanserat techbioföretag, fällorna du bör undvika, hur du avgör när äktenskapet mellan två områden fungerar eller inte fungerar, och nyanserna av att anpassa biologiska studier och maskininlärning till varandra.
FRAMTID: Liksom många discipliner finns det mycket spänning kring potentialen att tillämpa maskininlärning på bio. Men framstegen har verkat mer svårvunna. Är det något annorlunda med biomolekylär data jämfört med de typer av data som vanligtvis används med maskininlärning?
JIMMY LIN: Traditionell maskininlärningsdata är mycket bred och ytlig. Den typ av problem som maskininlärning ofta löser är vad människor kan lösa på en nanosekund, som bildigenkänning. För att lära en dator att känna igen bilden av en katt skulle du ha miljarder och miljarder bilder att träna på, men varje bild är relativt begränsad i sitt datainnehåll. Biologiska data är vanligtvis det omvända. Vi har inte miljarder individer. Vi har turen att få tusentals. Men för varje individ har vi miljarder och åter miljarder datapunkter. Vi har mindre antal mycket djupa data.
Samtidigt är biologiska frågor mer sällan de problem som människor kan lösa. Vi gör saker som inte ens världsexperter på detta kan göra. Så, arten av problemen är väldigt olika, så det kräver nytänkande om hur vi ställer oss till detta.
Behöver tillvägagångssätten byggas från grunden för biomolekylär data, eller kan man anpassa befintliga metoder?
Det finns sätt du kan ta denna djupa information och presentera den så att du kan dra nytta av de befintliga verktygen, oavsett om det är statistisk inlärning eller metoder för djupinlärning. Det är inte en direkt copy-paste, men det finns många sätt som du kan överföra många av maskininlärningsmetoderna och tillämpa dem på biologiska problem även om det inte är en direkt en-till-en-karta.
Att gräva lite mer i datafrågan, med biologiska data finns det mycket variation – det finns biologiskt brus, det finns experimentellt brus. Vad är det bästa sättet att närma sig generering av maskinlärande biomedicinsk data?
Det är en bra fråga. Redan från början har Freenome tagit hänsyn till hur man genererar den bästa data som lämpar sig för maskininlärning. Under hela processen från studiedesign, till provinsamling, till att köra analyserna, till dataanalys, måste det finnas omsorg i varje steg för att kunna optimera för maskininlärning, speciellt när du har så många fler funktioner än prover. Det är det klassiska big-p little-n-problemet.
Först och främst har vi utformat vår studie för att minimera konfounders. Många företag har förlitat sig på historiska datauppsättningar och har gjort mycket arbete för att försöka minimera kohorteffekter och ta bort konfounders. Men är det verkligen det bästa sättet att göra det? Tja, nej, det bästa sättet att göra det är en prospektiv studie där du kontrollerar för confounders på förhand. Det är därför vi, även i våra upptäcktsinsatser, bestämde oss för att göra en stor prospektiv studie på flera platser som samlar in guldstandarddata i förväg, som i vår AI-EMERGE-försök.
Lyckligtvis har vi investerare som trodde tillräckligt på oss för att vi ska kunna generera dessa data. Det var faktiskt en stor risk att ta eftersom dessa studier är väldigt dyra.
Sedan när du får informationen, vad gör du med den?
Tja, du måste träna alla webbplatser på ett konsekvent sätt och kontrollera för confounders från alla olika platser så att patienterna ser så lika ut som möjligt. Och sedan när du väl kört proverna måste du tänka igenom hur du kan minimera batcheffekter, till exempel genom att placera rätt blandning av prover på olika maskiner i rätt proportioner.
Det här är väldigt svårt när du gör det multiomics eftersom maskinerna som analyserar en klass av biomolekyler kan ta hundratals prover vid en körning, medan maskinerna som analyserar en annan klass av biomolekyler bara kan ta ett fåtal. Utöver det vill du ta bort mänskliga fel. Så vi introducerade automatisering ganska mycket i förväg, i det skede då vi bara genererade träningsdata.
Dessutom, när du har miljarder datapunkter per person blir det väldigt, väldigt lätt att potentiellt överanpassa. Så vi ser till att vår träning är generaliserbar till de populationer som vi i slutändan vill tillämpa den på, med rätt statistiska korrigeringar och många på varandra följande uppsättningar av träning och test.
Att kombinera maskininlärning med biomolekylär data är något som många bioteknikföretag försöker göra, men ofta är det mycket oklarheter om hur de ska göra detta. Vad ser du som en väsentlig egenskap för att effektivt integrera dem?
At Freenome vi kombinerar maskininlärning och multiomics. För att göra det måste du göra båda bra. Nyckeln här är att du måste ha stark expertis i båda och sedan kunna tala bådas språk. Du måste vara tvåspråkig.
Det finns massor av företag som är experter på det ena och sedan strö i ett lager av det andra. Till exempel finns det tekniska företag som bestämmer sig för att de vill hoppa in i bio, men allt de gör är att anställa en handfull våta labbforskare. Å andra sidan finns det biologiföretag som anställer vissa maskininlärningsforskare, då kommer de att förklara att de är ett AI/ML-företag nu.
Vad du verkligen behöver är djup bänkstyrka i båda. Du behöver en djup biologisk förståelse av systemet, av de olika analyserna, av funktionerna i kunskapsrummet. Men du behöver också ha en djup förståelse för maskininlärning, datavetenskap, beräkningsmetoder och statistisk inlärning, och ha plattformarna för att tillämpa det.
Det är verkligen utmanande eftersom de två områdena ofta är mycket silobelagda. När du tänker på de personer du anställer för företaget, hur skapar du broar mellan dessa två olika domäner?
Jag tror att det finns typ tre typer av människor du vill anställa för att överbrygga teknik och bio. De två första är dina standard, domänexperterna inom maskininlärning eller biologi. Men de måste också vara öppna och villiga att lära sig om den andra domänen, eller ännu hellre, ha haft exponering och erfarenhet av att arbeta inom dessa ytterligare domäner.
För maskininlärningsexperter väljer vi personer som inte bara är där för att utveckla den senaste algoritmen, utan som vill ta de senaste algoritmerna och tillämpa dem på biologiska frågor.
Biologi är kladdigt. Inte nog med att vi inte har alla metoder för att mäta de olika analyterna, utan vi upptäcker hela tiden nya biomolekyler och egenskaper. Det finns också många störande faktorer och buller man måste ta hänsyn till. Dessa problem är i allmänhet mer komplexa än vanliga maskininlärningsproblem, där problemet och kunskapsutrymmet är mycket mer väldefinierat. ML-experter som vill tillämpa sitt hantverk inom biologi behöver ha ödmjukhet för att lära sig om komplexiteten som finns inom biologi och vara villiga att arbeta med mindre än optimala förhållanden och skillnader i datatillgänglighet.
Baksidan är att anställa biologer som tänker på sina problem i termer av generering av kvantitativ data i större skala, designstudier för att optimera signal-brus-förhållanden och är medvetna om förbehållen för konfounders och generaliserbarhet. Det är mer än att bara kunna tala och tänka på kodens språk. Många av våra biologer kodar redan och har en bra statistisk bakgrund och vill och vill växa in i dessa områden. Faktum är att vi på Freenome faktiskt har utbildningsprogram för biologer som vill lära sig mer om kodning för att kunna utveckla sina statistiska resonemang.
Vad som är ännu viktigare är att studiedesign, och de frågor vi kan ställa, ser annorlunda ut när de utformas i sammanhang med big data och ML.
Vilken är den tredje typen?
Den tredje typen av person att anställa är den svåraste att hitta. Dessa är överbryggarna – personer som har arbetat flytande inom båda dessa områden. Det finns väldigt få platser och labb i världen som ligger precis vid den här korsningen. Att få tag i människor som kan översätta och överbrygga båda områdena är väldigt, väldigt viktigt. Men du vill inte bygga ett företag som bara består av bryggare eftersom dessa människor ofta inte är experter på det ena eller det andra området, på grund av vad de gör. De är ofta mer generella i sin förståelse. Men de ger det kritiska arbetet med att föra de två områdena samman.
Så det är viktigt att ha alla tre grupper av människor. Om du bara har en av domänexperterna kommer du bara att vara stark inom ett område. Eller, om du inte har brobyggarna, så har du silos av människor som inte kommer att kunna prata med varandra. Optimalt sett bör teamen inkludera var och en av dessa tre typer av människor för att möjliggöra en djup förståelse av både ML och biologi samt ge effektiv synergi mellan båda dessa områden.
Ser du skillnader i hur specialister inom teknik eller datorangrepp attackerar problem jämfört med hur biologer närmar sig problem?
Ja. Till en ytterlighet, vi har definitivt människor som kommer från en statistisk och kvantitativ bakgrund och de talar i kod och ekvationer. Vi måste hjälpa dem att ta dessa ekvationer och förklara det på ett tydligt sätt så att en allmän publik kan förstå.
Biologer har stor fantasi eftersom de arbetar med saker som är osynliga. De använder många illustrationer i presentationer för att visualisera vad som händer molekylärt, och de har stor intuition om mekanismer och komplexitet. Mycket av detta tänkande är mer kvalitativt. Detta ger ett annat sätt att tänka och kommunicera.
Så hur människor kommunicerar kommer att vara väldigt, väldigt olika. Nyckeln är – vi säger lite skämtsamt – att vi måste kommunicera på ett sätt som till och med din mormor kan förstå.
Det kräver sann behärskning av din kunskap för att kunna förenkla den så att även en nybörjare kan förstå. Jag tror att det faktiskt är bra träning för någon att lära sig att kommunicera mycket svåra koncept utanför de vanliga genvägarna, jargongen och det tekniska språket.
Vad har inspirerat din speciella syn på hur man kan kombinera maskininlärning och biologi?
Så problemet är inte nytt, utan snarare den senaste iterationen av ett urgammalt problem. När fälten av beräkningsbiologi och bioinformatik skapades först, samma problem fanns. Datavetare, statistiker, datavetare eller till och med fysiker anslöt sig till biologiområdet och förde sitt kvantitativa tänkande till fältet. Samtidigt var biologer tvungna att börja modellera bortom att karakterisera gener som uppreglerade och nedreglerade, och börja närma sig data mer kvantitativt. Digitaliseringen av biologisk data har nu precis vuxit exponentiellt i skala. Problemet är mer akut och expansivt till sin omfattning, men de grundläggande utmaningarna är desamma.
Vad ser du som antingen framgångsmåtten eller röda flaggor som talar om för dig om äktenskapet fungerar eller inte?
Om man tittar på företag som försöker kombinera områden kan man väldigt snabbt se hur mycket de investerar i den ena eller andra sidan. Så om det är ett företag där 90 % av människorna är labbforskare, och sedan anställt en eller två forskare inom maskininlärning och de kallar sig ett ML-företag, så är det förmodligen mer av en eftertanke.
Finns det en hemläxa som du har lärt dig i hela processen att förena biologi och maskininlärning?
Jag tror att intellektuell ödmjukhet, särskilt kommer från den tekniska sidan. Med något som att lösa för sökning, till exempel, finns all information redan i en textform som du enkelt kan komma åt, och du vet vad du letar efter. Så det blir ett lösbart problem, eller hur? Problemet med biologi är att vi inte ens vet vilka datauppsättningar vi letar efter, om vi ens har rätt ficklampa för att lysa på rätt områden.
Så, ibland när tekniska experter hoppar in i bio faller de i en fälla av alltför förenkling. Låt oss säga, som ett exempel, för nästa generations sekvensering kan de säga, "Wow. Vi kan sekvensera DNA. Varför sekvenserar vi inte bara massor av DNA? Det blir ett dataproblem och då löser vi biologi.”
Men problemet är att DNA är en av dussintals olika analyter i kroppen. Det finns RNA, protein,modifieringar efter översättning, olika fack såsom extracellulära vesiklar, och skillnader i tid, rum, celltyp, bland annat. Vi måste förstå möjligheterna och begränsningarna för varje datamodalitet vi använder.
Även om det kan vara svårt att tro, är biologi fortfarande ett område i sin linda. Vi bara sekvenserade ett mänskligt genom för lite över två decennier sedan. För det mesta kan vi inte komma åt enskilda biologiska signaler så vi gör fortfarande mätningar som är ett konglomerat eller genomsnitt över många signaler. Vi har precis börjat mäta en cell i taget. Det finns fortfarande mycket att göra och det är därför det är en spännande tid att gå in i biologin.
Men med den barndomen kommer stor potential att lösa problem som kommer att ha enorma effekter på människors hälsa och välbefinnande. Det är en ganska fantastisk tid eftersom vi öppnar nya gränser för biologin.
Vilka typer av gränser? Finns det ett område inom biologi eller medicin där du är mest glad över att se beräkning tillämpas?
Ja - allt! Men låt mig tänka. När det gäller cancer tror jag att inom vår generation kommer de nya terapierna och insatserna för tidig upptäckt som kommer ut kommer att förvandla cancer till en kronisk sjukdom som inte längre är så skrämmande, som vi har gjort för HIV. Och vi kan förmodligen använda väldigt liknande typer av metoder för att se på sjukdomsupptäckt och förebyggande mer generellt. Det viktigaste jag är exalterad över är att vi kan börja upptäcka om sjukdomen redan finns där innan symtom.
Utanför cancerdiagnostik är det som också är riktigt coolt övergången till att bygga med biologi istället för att bara läsa och skriva. Jag är entusiastisk över de områden inom syntetisk biologi där vi använder biologi som teknologi, oavsett om det är CRISPR eller syntetiska peptider eller syntetiska nukleotider. Att utnyttja biologi som ett verktyg skapar expansiva möjligheter att helt omvandla traditionella resursgenererande industrier, från jordbruk till energi. Det här är verkligen en fantastisk tid att vara biolog!
Upplagt 5 oktober 2022
Teknik, innovation och framtiden, som berättas av dem som bygger den.
- Andreessen Horowitz
- Bio & ScienceData
- Bitcoin
- blockchain
- blockchain-efterlevnad
- blockchain konferens
- coinbase
- coingenius
- Konsensus
- kryptokonferens
- crypto mining
- kryptovaluta
- decentraliserad
- Defi
- Digitala tillgångar
- ethereum
- maskininlärning
- icke fungibelt symbol
- plato
- plato ai
- Platon Data Intelligence
- Platonblockchain
- PlatonData
- platogaming
- Polygon
- bevis på spel
- W3
- zephyrnet