โปรตีนขับเคลื่อนกระบวนการทางชีวภาพหลายอย่าง เช่น กิจกรรมของเอนไซม์ การขนส่งโมเลกุล และการสนับสนุนเซลล์ โครงสร้างสามมิติของโปรตีนให้ข้อมูลเชิงลึกเกี่ยวกับหน้าที่และปฏิกิริยาของโปรตีนกับสารชีวโมเลกุลอื่นๆ วิธีการทดลองเพื่อระบุโครงสร้างโปรตีน เช่น X-ray crystallography และ NMR spectroscopy มีราคาแพงและใช้เวลานาน
ในทางตรงกันข้าม วิธีการคำนวณที่พัฒนาขึ้นเมื่อเร็วๆ นี้สามารถทำนายโครงสร้างของโปรตีนจากลำดับกรดอะมิโนได้อย่างรวดเร็วและแม่นยำ วิธีการเหล่านี้มีความสำคัญอย่างยิ่งต่อโปรตีนที่ยากต่อการศึกษาเชิงทดลอง เช่น โปรตีนเมมเบรน ซึ่งเป็นเป้าหมายของยาหลายชนิด ตัวอย่างหนึ่งที่รู้จักกันดีคือ อัลฟ่าโฟลด์อัลกอริทึมที่ใช้การเรียนรู้เชิงลึกได้รับการยกย่องจากการคาดการณ์ที่แม่นยำ
ESMFพับ เป็นวิธีการเรียนรู้เชิงลึกที่มีความแม่นยำสูงอีกวิธีหนึ่งที่พัฒนาขึ้นเพื่อทำนายโครงสร้างโปรตีนจากลำดับกรดอะมิโน ESMFold ใช้โมเดลภาษาโปรตีนขนาดใหญ่ (pLM) เป็นแกนหลักและดำเนินการตั้งแต่ต้นจนจบ ซึ่งแตกต่างจาก AlphaFold2 ตรงที่ไม่ต้องค้นหาหรือ การจัดตำแหน่งหลายลำดับ ขั้นตอน (MSA) และไม่ใช้ฐานข้อมูลภายนอกเพื่อสร้างการคาดการณ์ ทีมพัฒนาได้ฝึกฝนแบบจำลองเกี่ยวกับลำดับโปรตีนนับล้านจาก UniRef ในระหว่างการฝึกอบรม แบบจำลองได้พัฒนารูปแบบความสนใจที่แสดงถึงปฏิสัมพันธ์เชิงวิวัฒนาการระหว่างกรดอะมิโนในลำดับอย่างสวยงาม การใช้ pLM แทน MSA ทำให้สามารถคาดการณ์ได้เร็วกว่าโมเดลล้ำสมัยอื่นๆ ถึง 60 เท่า
ในโพสต์นี้ เราใช้โมเดล ESMFold ที่ฝึกไว้ล่วงหน้าจาก Hugging Face ด้วย อเมซอน SageMaker เพื่อทำนายโครงสร้างห่วงโซ่หนักของ ทราสตูซูแมบที่ โมโนโคลนอลแอนติบอดี พัฒนาโดย Genentech สำหรับการรักษา มะเร็งเต้านม HER2-positive. การทำนายโครงสร้างของโปรตีนอย่างรวดเร็วอาจมีประโยชน์หากนักวิจัยต้องการทดสอบผลของการดัดแปลงลำดับ สิ่งนี้อาจนำไปสู่การรอดชีวิตของผู้ป่วยที่ดีขึ้นหรือผลข้างเคียงน้อยลง
โพสต์นี้มีตัวอย่างสมุดบันทึก Jupyter และสคริปต์ที่เกี่ยวข้องดังต่อไปนี้ พื้นที่เก็บข้อมูล GitHub.
เบื้องต้น
เราขอแนะนำให้เรียกใช้ตัวอย่างนี้ใน สตูดิโอ Amazon SageMaker สมุดบันทึก รันอิมเมจ PyTorch 1.13 Python 3.9 CPU-optimized บนอินสแตนซ์ประเภท ml.r5.xlarge
เห็นภาพโครงสร้างการทดลองของ trastuzumab
ในการเริ่มต้นเราใช้ biopython ไลบรารีและสคริปต์ตัวช่วยเพื่อดาวน์โหลดโครงสร้าง trastuzumab จาก ธนาคารข้อมูลโปรตีน RCSB:
ต่อไปเราใช้ py3Dmol ห้องสมุดเพื่อแสดงภาพโครงสร้างเป็นการแสดงภาพ 3 มิติแบบโต้ตอบ:
รูปต่อไปนี้แสดงถึงโครงสร้างโปรตีน 3 มิติ 1N8Z จาก Protein Data Bank (PDB) ในภาพนี้ สายเบาทราสตูซูแมบแสดงเป็นสีส้ม สายหนักเป็นสีน้ำเงิน (โดยมีบริเวณที่แปรผันเป็นสีฟ้าอ่อน) และแอนติเจน HER2 เป็นสีเขียว
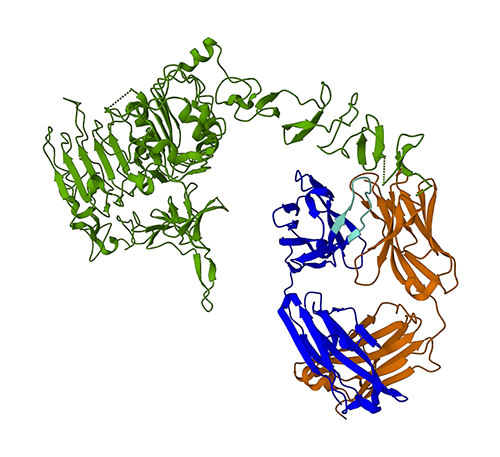
ก่อนอื่น เราจะใช้ ESMFold เพื่อทำนายโครงสร้างของสายหนัก (สาย B) จากลำดับกรดอะมิโน จากนั้นเราจะเปรียบเทียบการทำนายกับโครงสร้างที่กำหนดโดยการทดลองที่แสดงด้านบน
ทำนายโครงสร้างของโซ่หนัก Trastuzumab จากลำดับโดยใช้ ESMFold
ลองใช้แบบจำลอง ESMFold เพื่อทำนายโครงสร้างของห่วงโซ่หนักและเปรียบเทียบกับผลการทดลอง ในการเริ่มต้น เราจะใช้สภาพแวดล้อมของโน้ตบุ๊กที่สร้างไว้ล่วงหน้าใน Studio ที่มาพร้อมกับไลบรารี่สำคัญๆ มากมาย เช่น ไพทอร์ช, ติดตั้งล่วงหน้า. แม้ว่าเราจะสามารถใช้อินสแตนซ์ประเภทเร่งความเร็วเพื่อปรับปรุงประสิทธิภาพการวิเคราะห์โน้ตบุ๊กของเราได้ แต่เราจะใช้อินสแตนซ์ที่ไม่เร่งความเร็วแทน และเรียกใช้การคาดคะเน ESMFold บน CPU
ก่อนอื่น เราโหลดโมเดล ESMFold และโทเค็นไนเซอร์ที่ได้รับการฝึกอบรมล่วงหน้าจาก กอดใบหน้าฮับ:
ต่อไป เราคัดลอกโมเดลไปยังอุปกรณ์ของเรา (ในกรณีนี้คือ CPU) และตั้งค่าพารามิเตอร์ของโมเดล:
เพื่อเตรียมลำดับโปรตีนสำหรับการวิเคราะห์ เราจำเป็นต้องโทเค็น สิ่งนี้แปลสัญลักษณ์กรดอะมิโน (EVQLV…) เป็นรูปแบบตัวเลขที่โมเดล ESMFold สามารถเข้าใจได้ (6,19,5,10,19,…):
ต่อไป เราคัดลอกอินพุตที่เป็นโทเค็นไปยังโหมด ทำการคาดคะเน และบันทึกผลลัพธ์ลงในไฟล์:
ซึ่งจะใช้เวลาประมาณ 3 นาทีสำหรับประเภทอินสแตนซ์ที่ไม่เร่งความเร็ว เช่น r5
เราสามารถตรวจสอบความถูกต้องของการทำนาย ESMFold โดยเปรียบเทียบกับโครงสร้างการทดลอง เราทำสิ่งนี้โดยใช้ US-จัดตำแหน่ง เครื่องมือที่พัฒนาโดย Zhang Lab แห่งมหาวิทยาลัยมิชิแกน:
| พีดีบีเชน1 | พีดีบีเชน2 | คะแนน TM |
| data/prediction.pdb:ก | ข้อมูล/experimental.pdb:B | 0.802 |
พื้นที่ คะแนนการสร้างแบบจำลองเทมเพลต (TM-score) เป็นตัวชี้วัดสำหรับการประเมินความคล้ายคลึงกันของโครงสร้างโปรตีน คะแนน 1.0 หมายถึงการจับคู่ที่สมบูรณ์แบบ คะแนนที่สูงกว่า 0.7 แสดงว่าโปรตีนมีโครงสร้างกระดูกสันหลังเหมือนกัน คะแนนที่สูงกว่า 0.9 แสดงว่ามีโปรตีน ใช้แทนกันได้ สำหรับการใช้งานด้านปลายน้ำ ในกรณีของเราที่ได้ TM-Score 0.802 การคาดคะเน ESMFold น่าจะเหมาะสมสำหรับการใช้งาน เช่น การให้คะแนนโครงสร้างหรือการทดสอบการจับลิแกนด์ แต่อาจไม่เหมาะกับกรณีการใช้งานเช่น การแทนที่โมเลกุล ที่ต้องการความแม่นยำสูงมาก
เราสามารถตรวจสอบผลลัพธ์นี้ได้โดยการแสดงภาพโครงสร้างที่สอดคล้องกัน โครงสร้างทั้งสองแสดงระดับการทับซ้อนกันสูง แต่ไม่สมบูรณ์ การคาดคะเนโครงสร้างโปรตีนเป็นสาขาที่มีการพัฒนาอย่างรวดเร็ว และทีมวิจัยจำนวนมากกำลังพัฒนาอัลกอริทึมที่แม่นยำยิ่งขึ้น!
ปรับใช้ ESMFold เป็นจุดสิ้นสุดการอนุมานของ SageMaker
การเรียกใช้การอนุมานโมเดลในโน้ตบุ๊กนั้นดีสำหรับการทดลอง แต่ถ้าคุณต้องการรวมโมเดลของคุณเข้ากับแอปพลิเคชันล่ะ หรือไปป์ไลน์ MLOps? ในกรณีนี้ ตัวเลือกที่ดีกว่าคือการปรับใช้โมเดลของคุณเป็นจุดสิ้นสุดการอนุมาน ในตัวอย่างต่อไปนี้ เราจะปรับใช้ ESMFold เป็นจุดสิ้นสุดการอนุมานแบบเรียลไทม์ของ SageMaker บนอินสแตนซ์แบบเร่งความเร็ว ตำแหน่งข้อมูลแบบเรียลไทม์ของ SageMaker มอบวิธีการปรับใช้และโฮสต์โมเดลแมชชีนเลิร์นนิง (ML) ที่ปรับขนาดได้ ประหยัดค่าใช้จ่าย และปลอดภัย ด้วยการปรับขนาดอัตโนมัติ คุณสามารถปรับจำนวนอินสแตนซ์ที่เรียกใช้จุดสิ้นสุดเพื่อให้ตรงกับความต้องการของแอปพลิเคชันของคุณ ปรับต้นทุนให้เหมาะสมและรับประกันความพร้อมใช้งานสูง
ที่สร้างไว้ล่วงหน้า คอนเทนเนอร์ SageMaker สำหรับ Hugging Face ทำให้ปรับใช้โมเดลการเรียนรู้เชิงลึกสำหรับงานทั่วไปได้ง่าย อย่างไรก็ตาม สำหรับกรณีการใช้งานใหม่ เช่น การทำนายโครงสร้างโปรตีน เราจำเป็นต้องกำหนดแบบกำหนดเอง inference.py สคริปต์เพื่อโหลดโมเดล รันการคาดคะเน และจัดรูปแบบเอาต์พุต สคริปต์นี้มีรหัสเดียวกันกับที่เราใช้ในสมุดบันทึกของเรา เรายังสร้าง requirements.txt ไฟล์เพื่อกำหนดการอ้างอิง Python สำหรับปลายทางของเราที่จะใช้ คุณสามารถดูไฟล์ที่เราสร้างขึ้นในไฟล์ พื้นที่เก็บข้อมูล GitHub.
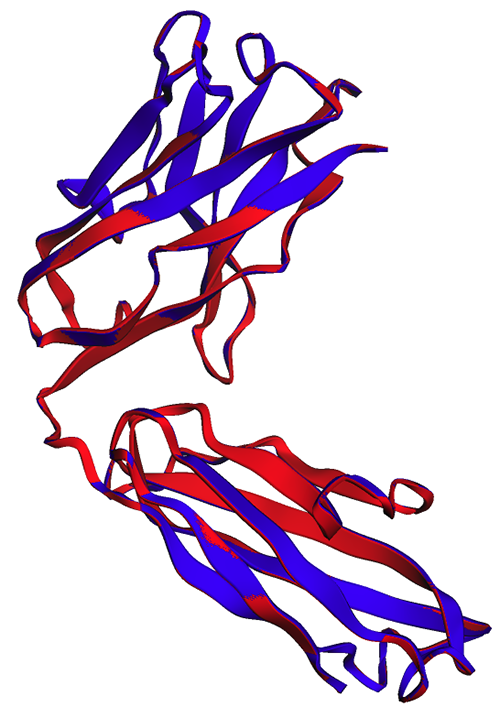
ในรูปต่อไปนี้ โครงสร้างการทดลอง (สีน้ำเงิน) และที่คาดการณ์ไว้ (สีแดง) ของสายโซ่หนักทราสตูซูแมบคล้ายกันมาก แต่ไม่เหมือนกัน

หลังจากที่เราสร้างไฟล์ที่จำเป็นในไฟล์ code ไดเร็กทอรี เราปรับใช้โมเดลของเราโดยใช้ SageMaker HuggingFaceModel ระดับ. สิ่งนี้ใช้คอนเทนเนอร์ที่สร้างไว้ล่วงหน้าเพื่อลดความซับซ้อนของกระบวนการปรับใช้โมเดล Hugging Face กับ SageMaker โปรดทราบว่าอาจใช้เวลา 10 นาทีขึ้นไปในการสร้างจุดสิ้นสุด ขึ้นอยู่กับความพร้อมใช้งานของ ml.g4dn ประเภทอินสแตนซ์ในภูมิภาคของเรา
เมื่อการใช้งานปลายทางเสร็จสิ้น เราสามารถส่งลำดับโปรตีนอีกครั้งและแสดงสองสามแถวแรกของการคาดคะเน:
เนื่องจากเราปรับใช้จุดสิ้นสุดของเรากับอินสแตนซ์แบบเร่ง การคาดคะเนจึงใช้เวลาเพียงไม่กี่วินาที แต่ละแถวในผลลัพธ์จะสอดคล้องกับอะตอมเดี่ยวและรวมถึงเอกลักษณ์ของกรดอะมิโน พิกัดเชิงพื้นที่สามพิกัด และ คะแนน pLDDT แสดงถึงความเชื่อมั่นในการทำนาย ณ ตำแหน่งนั้นๆ
| PDB_GROUP | ID | อะตอม_ฉลาก | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | ครอบครอง | พีแอลดีที | อะตอม_ไอดี |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
โดยใช้วิธีเดียวกับก่อนหน้านี้ เราจะเห็นว่าการคาดการณ์ของโน้ตบุ๊กและจุดสิ้นสุดนั้นเหมือนกัน
| พีดีบีเชน1 | พีดีบีเชน2 | คะแนน TM |
| data/endpoint_prediction.pdb:ก | data/prediction.pdb:ก | 1.0 |
ตามที่สังเกตในรูปต่อไปนี้ การคาดคะเน ESMFold ที่สร้างขึ้นในโน้ตบุ๊ก (สีแดง) และโดยจุดสิ้นสุด (สีน้ำเงิน) แสดงการจัดตำแหน่งที่สมบูรณ์แบบ
ทำความสะอาด
เพื่อหลีกเลี่ยงการเรียกเก็บเงินเพิ่มเติม เราจะลบข้อมูลปลายทางการอนุมานและข้อมูลการทดสอบ:
สรุป
การทำนายโครงสร้างโปรตีนด้วยคอมพิวเตอร์เป็นเครื่องมือสำคัญในการทำความเข้าใจการทำงานของโปรตีน นอกเหนือจากการวิจัยพื้นฐานแล้ว อัลกอริทึมอย่าง AlphaFold และ ESMFold ยังมีการประยุกต์ใช้มากมายในด้านการแพทย์และเทคโนโลยีชีวภาพ ข้อมูลเชิงลึกเชิงโครงสร้างที่สร้างขึ้นโดยแบบจำลองเหล่านี้ช่วยให้เราเข้าใจได้ดีขึ้นว่าชีวโมเลกุลมีปฏิสัมพันธ์กันอย่างไร สิ่งนี้สามารถนำไปสู่เครื่องมือวินิจฉัยและการรักษาที่ดีขึ้นสำหรับผู้ป่วย
ในโพสต์นี้ เราจะแสดงวิธีการปรับใช้โมเดลภาษาโปรตีน ESMFold จาก Hugging Face Hub เป็นจุดสิ้นสุดการอนุมานที่ปรับขนาดได้โดยใช้ SageMaker สำหรับข้อมูลเพิ่มเติมเกี่ยวกับการปรับใช้โมเดล Hugging Face บน SageMaker โปรดดูที่ ใช้การกอดใบหน้ากับ Amazon SageMaker. คุณยังสามารถหาตัวอย่างทางวิทยาศาสตร์เกี่ยวกับโปรตีนเพิ่มเติมได้ใน การวิเคราะห์โปรตีนที่ยอดเยี่ยมบน AWS ที่เก็บ GitHub โปรดแสดงความคิดเห็นหากมีตัวอย่างอื่นๆ ที่คุณต้องการดู!
เกี่ยวกับผู้เขียน
 ไบรอัน ภักดี เป็นสถาปนิกอาวุโสด้าน AI/ML Solutions ในทีม Global Healthcare and Life Sciences ที่ Amazon Web Services เขามีประสบการณ์มากกว่า 17 ปีในด้านเทคโนโลยีชีวภาพและแมชชีนเลิร์นนิง และมีความกระตือรือร้นในการช่วยลูกค้าแก้ปัญหาด้านจีโนมและโปรตีโอมิก ในเวลาว่าง เขาชอบทำอาหารและทานอาหารกับเพื่อนและครอบครัว
ไบรอัน ภักดี เป็นสถาปนิกอาวุโสด้าน AI/ML Solutions ในทีม Global Healthcare and Life Sciences ที่ Amazon Web Services เขามีประสบการณ์มากกว่า 17 ปีในด้านเทคโนโลยีชีวภาพและแมชชีนเลิร์นนิง และมีความกระตือรือร้นในการช่วยลูกค้าแก้ปัญหาด้านจีโนมและโปรตีโอมิก ในเวลาว่าง เขาชอบทำอาหารและทานอาหารกับเพื่อนและครอบครัว
 ชามิกา อริยวรรณศ เป็นสถาปนิก AI/ML Specialist Solutions ในทีม Global Healthcare and Life Sciences ที่ Amazon Web Services เขาทำงานร่วมกับลูกค้าอย่างกระตือรือร้นเพื่อเร่งการนำ AI และ ML ไปใช้โดยให้คำแนะนำทางเทคนิคและช่วยพวกเขาคิดค้นและสร้างโซลูชันระบบคลาวด์ที่ปลอดภัยบน AWS นอกเวลางาน เขาชอบเล่นสกีและขับรถวิบาก
ชามิกา อริยวรรณศ เป็นสถาปนิก AI/ML Specialist Solutions ในทีม Global Healthcare and Life Sciences ที่ Amazon Web Services เขาทำงานร่วมกับลูกค้าอย่างกระตือรือร้นเพื่อเร่งการนำ AI และ ML ไปใช้โดยให้คำแนะนำทางเทคนิคและช่วยพวกเขาคิดค้นและสร้างโซลูชันระบบคลาวด์ที่ปลอดภัยบน AWS นอกเวลางาน เขาชอบเล่นสกีและขับรถวิบาก
 หยานจุนฉี เป็นผู้จัดการวิทยาศาสตร์ประยุกต์อาวุโสที่ AWS Machine Learning Solution Lab เธอคิดค้นและใช้แมชชีนเลิร์นนิงเพื่อช่วยให้ลูกค้า AWS เร่งการนำ AI และระบบคลาวด์ไปใช้
หยานจุนฉี เป็นผู้จัดการวิทยาศาสตร์ประยุกต์อาวุโสที่ AWS Machine Learning Solution Lab เธอคิดค้นและใช้แมชชีนเลิร์นนิงเพื่อช่วยให้ลูกค้า AWS เร่งการนำ AI และระบบคลาวด์ไปใช้
- เนื้อหาที่ขับเคลื่อนด้วย SEO และการเผยแพร่ประชาสัมพันธ์ รับการขยายวันนี้
- เพลโตไอสตรีม. ข้อมูลอัจฉริยะ Web3 ขยายความรู้ เข้าถึงได้ที่นี่.
- การสร้างอนาคตโดย Adryenn Ashley เข้าถึงได้ที่นี่.
- ซื้อและขายหุ้นในบริษัท PRE-IPO ด้วย PREIPO® เข้าถึงได้ที่นี่.
- ที่มา: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :มี
- :เป็น
- :ไม่
- $ ขึ้น
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- เกี่ยวกับเรา
- ข้างบน
- AC
- เร่งความเร็ว
- เร่ง
- ความถูกต้อง
- ถูกต้อง
- แม่นยำ
- การบรรลุ
- อยากทำกิจกรรม
- นอกจากนี้
- การนำมาใช้
- AI
- AI / ML
- ขั้นตอนวิธี
- อัลกอริทึม
- ชิด
- การวางแนว
- ด้วย
- แม้ว่า
- อเมซอน
- อเมซอน SageMaker
- Amazon Web Services
- an
- การวิเคราะห์
- และ
- อื่น
- ใด
- การใช้งาน
- การใช้งาน
- ประยุกต์
- เหมาะสม
- เป็น
- AS
- การประเมิน
- At
- อะตอม
- ความสนใจ
- อัตโนมัติ
- ความพร้อมใช้งาน
- หลีกเลี่ยง
- AWS
- AWS Machine Learning AWS
- กระดูกสันหลัง
- ธนาคาร
- ขั้นพื้นฐาน
- BE
- ก่อน
- เริ่ม
- ดีกว่า
- ระหว่าง
- ผูกพัน
- เทคโนโลยีชีวภาพ
- สีน้ำเงิน
- สร้าง
- แต่
- by
- CAN
- โรคมะเร็ง
- การ์ตูน
- กรณี
- กรณี
- โด่งดัง
- โซ่
- ความท้าทาย
- โหลด
- ตรวจสอบ
- ชั้น
- เมฆ
- การยอมรับระบบคลาวด์
- รหัส
- สี
- มา
- ความเห็น
- ร่วมกัน
- เปรียบเทียบ
- เปรียบเทียบ
- สมบูรณ์
- ความมั่นใจ
- ภาชนะ
- ตรงกันข้าม
- สอดคล้อง
- ค่าใช้จ่ายที่มีประสิทธิภาพ
- ค่าใช้จ่าย
- ได้
- สร้าง
- ที่สร้างขึ้น
- วิกฤติ
- ลูกค้า
- ข้อมูล
- ฐานข้อมูล
- วันเวลา
- ลึก
- การเรียนรู้ลึก ๆ
- Deepmind
- องศา
- ความต้องการ
- ทั้งนี้ขึ้นอยู่กับ
- ปรับใช้
- นำไปใช้
- ปรับใช้
- การใช้งาน
- กำหนด
- แน่นอน
- พัฒนา
- ที่กำลังพัฒนา
- พัฒนาการ
- เครื่อง
- ยาก
- แสดง
- do
- ทำ
- ไม่
- ดาวน์โหลด
- ขับรถ
- ยาเสพติด
- ในระหว่าง
- แต่ละ
- ง่าย
- ผล
- ผลกระทบ
- ช่วยให้
- ปลาย
- ปลายทาง
- การสร้างความมั่นใจ
- สิ่งแวดล้อม
- ตัวอย่าง
- ตัวอย่าง
- แพง
- ประสบการณ์
- การทดลอง
- ภายนอก
- อย่างยิ่ง
- ใบหน้า
- ครอบครัว
- เร็วขึ้น
- สองสาม
- น้อยลง
- สนาม
- รูป
- เนื้อไม่มีมัน
- ไฟล์
- หา
- ปลาย
- ชื่อจริง
- ดังต่อไปนี้
- สำหรับ
- รูป
- เพื่อน
- ราคาเริ่มต้นที่
- ฟังก์ชัน
- ต่อไป
- สร้าง
- สร้าง
- GitHub
- เหตุการณ์ที่
- สีเขียว
- คำแนะนำ
- มี
- he
- การดูแลสุขภาพ
- หนัก
- ช่วย
- การช่วยเหลือ
- จุดสูง
- ของเขา
- เจ้าภาพ
- สรุป ความน่าเชื่อถือของ Olymp Trade?
- ทำอย่างไร
- อย่างไรก็ตาม
- HTML
- HTTPS
- Hub
- กอดใบหน้า
- identiques
- เอกลักษณ์
- if
- ภาพ
- นำเข้า
- สำคัญ
- ปรับปรุง
- การปรับปรุง
- in
- รวมถึง
- แสดง
- บ่งชี้ว่า
- ข้อมูล
- เราสร้างสรรค์สิ่งใหม่ ๆ
- อินพุต
- ความเข้าใจ
- ข้อมูลเชิงลึก
- ตัวอย่าง
- แทน
- รวบรวม
- โต้ตอบ
- ปฏิสัมพันธ์
- การโต้ตอบ
- เชิงโต้ตอบ
- เข้าไป
- IT
- ITS
- ห้องปฏิบัติการ
- ภาษา
- ใหญ่
- นำ
- การเรียนรู้
- ทิ้ง
- ห้องสมุด
- ห้องสมุด
- ชีวิต
- วิทยาศาสตร์สิ่งมีชีวิต
- เบา
- กดไลก์
- น่าจะ
- โหลด
- ที่ตั้ง
- ค้นหา
- เครื่อง
- เรียนรู้เครื่อง
- ทำ
- ทำให้
- ผู้จัดการ
- หลาย
- การจับคู่
- อาจ..
- ยา
- พบ
- วิธี
- วิธีการ
- เมตริก
- มิชิแกน
- ล้าน
- นาที
- ML
- ม.ป.ป
- โหมด
- แบบ
- โมเดล
- การปรับเปลี่ยน
- โมเลกุล
- ข้อมูลเพิ่มเติม
- มาก
- ชื่อ
- จำเป็น
- จำเป็นต้อง
- NIH
- สมุดบันทึก
- นวนิยาย
- จำนวน
- วัตถุ
- ล้าสมัย
- of
- on
- ONE
- เพียง
- ดำเนินการ
- การเพิ่มประสิทธิภาพ
- ตัวเลือกเสริม (Option)
- or
- ส้ม
- OS
- อื่นๆ
- ของเรา
- เอาท์พุต
- ด้านนอก
- พารามิเตอร์
- หลงใหล
- ผู้ป่วย
- ผู้ป่วย
- รูปแบบ
- สมบูรณ์
- การปฏิบัติ
- ท่อ
- เพลโต
- เพลโตดาต้าอินเทลลิเจนซ์
- เพลโตดาต้า
- กรุณา
- โพสต์
- ที่อาจเกิดขึ้น
- คาดการณ์
- ที่คาดการณ์
- ทำนาย
- คำทำนาย
- การคาดการณ์
- เตรียมการ
- ก่อนหน้านี้
- กระบวนการ
- กระบวนการ
- โปรตีน
- โปรตีน
- ให้
- ให้
- การให้
- หลาม
- ไฟฉาย
- Qi
- อย่างรวดเร็ว
- อย่างรวดเร็ว
- เขตข้อมูลที่มีการพัฒนาอย่างรวดเร็ว
- เรียลไทม์
- แนะนำ
- สีแดง
- ภูมิภาค
- ที่เกี่ยวข้อง
- วางใจ
- แสดง
- เป็นตัวแทนของ
- แสดงให้เห็นถึง
- ต้องการ
- การวิจัย
- นักวิจัย
- ผล
- แถว
- วิ่ง
- วิ่ง
- sagemaker
- การอนุมาน SageMaker
- เดียวกัน
- ลด
- ที่ปรับขนาดได้
- ปรับ
- วิทยาศาสตร์
- วิทยาศาสตร์
- คะแนน
- คะแนน
- สคริปต์
- วินาที
- ปลอดภัย
- เห็น
- ระดับอาวุโส
- ลำดับ
- บริการ
- ชุด
- หลาย
- Share
- เธอ
- น่า
- โชว์
- แสดง
- ด้าน
- คล้ายคลึงกัน
- ลดความซับซ้อน
- เดียว
- ทางออก
- โซลูชัน
- แก้
- บาง
- เกี่ยวกับอวกาศ
- ผู้เชี่ยวชาญ
- สเปก
- ความเร็ว
- เริ่มต้น
- รัฐของศิลปะ
- ขั้นตอน
- โครงสร้าง
- โครงสร้าง
- สตูดิโอ
- ศึกษา
- อย่างเช่น
- เหมาะสม
- สนับสนุน
- การอยู่รอด
- เอา
- ใช้เวลา
- เป้าหมาย
- งาน
- ทีม
- ทีม
- วิชาการ
- ทดสอบ
- กว่า
- ที่
- พื้นที่
- ของพวกเขา
- พวกเขา
- แล้วก็
- ที่นั่น
- ล้อยางขัดเหล่านี้ติดตั้งบนแกน XNUMX (มม.) ผลิตภัณฑ์นี้ถูกผลิตในหลายรูปทรง และหลากหลายเบอร์ความแน่นหนาของปริมาณอนุภาคขัดของมัน จะทำให้ท่านได้รับประสิทธิภาพสูงในการขัดและการใช้งานที่ยาวนาน
- นี้
- สาม
- สามมิติ
- เวลา
- ต้องใช้เวลามาก
- ครั้ง
- ไปยัง
- โทเค็น
- โทเค็น
- เครื่องมือ
- เครื่องมือ
- ไฟฉาย
- ผ่านการฝึกอบรม
- การฝึกอบรม
- หม้อแปลง
- การขนส่ง
- การรักษา
- สอง
- ชนิด
- ชนิด
- เข้าใจ
- ความเข้าใจ
- มหาวิทยาลัย
- แตกต่าง
- us
- ใช้
- มือสอง
- การใช้
- ตรวจสอบความถูกต้อง
- มาก
- รายละเอียด
- การสร้างภาพ
- W
- อยาก
- ทาง..
- we
- เว็บ
- บริการเว็บ
- โด่งดัง
- อะไร
- วิกิพีเดีย
- จะ
- กับ
- WordPress
- งาน
- โรงงาน
- จะ
- รังสีเอกซ์
- คุณ
- ของคุณ
- ลมทะเล