Білки керують багатьма біологічними процесами, такими як активність ферментів, молекулярний транспорт і підтримка клітин. Тривимірна структура білка дає розуміння його функції та того, як він взаємодіє з іншими біомолекулами. Експериментальні методи визначення структури білка, такі як рентгенівська кристалографія та ЯМР-спектроскопія, є дорогими та трудомісткими.
Навпаки, нещодавно розроблені обчислювальні методи можуть швидко й точно передбачити структуру білка за його амінокислотною послідовністю. Ці методи мають вирішальне значення для білків, які важко дослідити експериментально, таких як мембранні білки, мішені для багатьох ліків. Одним із відомих прикладів цього є AlphaFold, алгоритм на основі глибокого навчання, відомий своїми точними передбаченнями.
ESMFold це ще один високоточний метод, заснований на глибокому навчанні, розроблений для прогнозування структури білка за його амінокислотною послідовністю. ESMFold використовує модель великої білкової мови (pLM) як основу та працює наскрізно. На відміну від AlphaFold2, він не потребує пошуку або Вирівнювання кількох послідовностей (MSA), а також не покладається на зовнішні бази даних для створення прогнозів. Замість цього команда розробників навчила модель на мільйонах білкових послідовностей з UniRef. Під час навчання модель розробила шаблони уваги, які елегантно представляють еволюційні взаємодії між амінокислотами в послідовності. Таке використання pLM замість MSA забезпечує до 60 разів швидший час прогнозування, ніж інші найсучасніші моделі.
У цій публікації ми використовуємо попередньо підготовлену модель ESMFold від Hugging Face with Amazon SageMaker для прогнозування структури важкого ланцюга трастузумаб, то моноклональне антитіло вперше розроблено Genentech для лікування HER2-позитивний рак молочної залози. Швидке передбачення структури цього білка може бути корисним, якщо дослідники хочуть перевірити ефект модифікації послідовності. Це потенційно може призвести до покращення виживаності пацієнтів або зменшення побічних ефектів.
У цьому дописі наведено приклад блокнота Jupyter і пов’язаних із ним сценаріїв GitHub сховище.
Передумови
Ми рекомендуємо запустити цей приклад у Студія Amazon SageMaker ноутбук запуск зображення PyTorch 1.13 Python 3.9, оптимізованого для ЦП, на типі примірника ml.r5.xlarge.
Візуалізуйте експериментальну структуру трастузумабу
Для початку використовуємо biopython бібліотеку та допоміжний сценарій для завантаження структури трастузумабу з Банк даних білка RCSB:
Далі використовуємо py3Dmol бібліотека для візуалізації конструкції як інтерактивної 3D візуалізації:
На наступному малюнку зображено тривимірну структуру білка 3N1Z з банку даних про білки (PDB). На цьому зображенні легкий ланцюг трастузумабу позначено помаранчевим кольором, важкий ланцюг — синім (з варіабельною областю світло-блакитним), а антиген HER8 — зеленим.
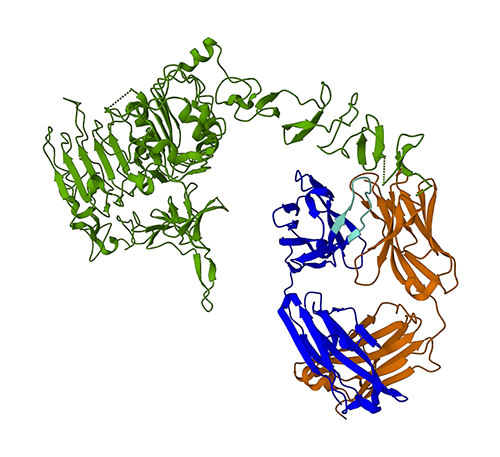
Спочатку ми використаємо ESMFold, щоб передбачити структуру важкого ланцюга (ланцюг B) за його амінокислотною послідовністю. Потім ми порівняємо передбачення з експериментально визначеною структурою, показаною вище.
Передбачте структуру важкого ланцюга трастузумабу за його послідовністю за допомогою ESMFold
Давайте використаємо модель ESMFold, щоб передбачити структуру важкого ланцюга та порівняти її з експериментальним результатом. Для початку ми використаємо попередньо створене середовище блокнота в Studio, яке постачається з кількома важливими бібліотеками, як-от PyTorch, попередньо встановлено. Хоча ми могли б використовувати прискорений тип екземпляра для покращення продуктивності нашого аналізу блокнота, натомість ми використаємо неприскорений екземпляр і запустимо передбачення ESMFold на ЦП.
Спочатку ми завантажуємо попередньо навчену модель ESMFold і токенізер з Hugging Face Hub:
Далі ми копіюємо модель на наш пристрій (у цьому випадку ЦП) і встановлюємо деякі параметри моделі:
Щоб підготувати білкову послідовність для аналізу, нам потрібно її токенізувати. Це переводить символи амінокислот (EVQLV…) у числовий формат, який може зрозуміти модель ESMFold (6,19,5,10,19,…):
Далі ми копіюємо токенізоване введення в режим, робимо прогноз і зберігаємо результат у файл:
Це займає приблизно 3 хвилини на неприскореному типі екземпляра, наприклад r5.
Ми можемо перевірити точність прогнозу ESMFold, порівнявши його з експериментальною структурою. Ми робимо це за допомогою US-Align Інструмент, розроблений лабораторією Zhang Lab Мічиганського університету:
| PDBchain1 | PDBchain2 | TM-Score |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
Команда шаблон моделювання партитури (TM-score) є показником для оцінки подібності білкових структур. Оцінка 1.0 означає ідеальний збіг. Оцінки вище 0.7 вказують на те, що білки мають однакову структуру магістралі. Оцінки вище 0.9 вказують на наявність білків функціонально взаємозамінні для подальшого використання. У нашому випадку досягнення TM-Score 0.802 прогноз ESMFold, ймовірно, підійде для таких застосувань, як оцінка структури або експерименти зі зв’язуванням ліганду, але може не підійти для таких випадків використання молекулярна заміна які вимагають надзвичайно високої точності.
Ми можемо підтвердити цей результат, візуалізувавши вирівняні структури. Дві структури демонструють високий, але не ідеальний ступінь перекриття. Прогнозування структури білка – це галузь, що швидко розвивається, і багато дослідницьких груп розробляють дедалі точніші алгоритми!
Розгорніть ESMFold як кінцеву точку висновку SageMaker
Запуск моделювання в блокноті чудово підходить для експериментів, але що, якщо вам потрібно інтегрувати свою модель із програмою? Або конвеєр MLOps? У цьому випадку кращим варіантом є розгортання вашої моделі як кінцевої точки висновку. У наступному прикладі ми розгорнемо ESMFold як кінцеву точку висновку SageMaker у реальному часі на прискореному екземплярі. Кінцеві точки реального часу SageMaker забезпечують масштабований, економічно ефективний і безпечний спосіб розгортання та розміщення моделей машинного навчання (ML). За допомогою автоматичного масштабування ви можете регулювати кількість екземплярів, на яких працює кінцева точка, щоб відповідати вимогам вашої програми, оптимізуючи витрати та забезпечуючи високу доступність.
Попередньо побудований Контейнер SageMaker для Hugging Face дозволяє легко розгортати моделі глибокого навчання для звичайних завдань. Однак для нових випадків використання, як-от передбачення структури білка, нам потрібно визначити настроювання inference.py сценарій для завантаження моделі, запуску прогнозу та форматування виводу. Цей сценарій містить майже той самий код, який ми використовували в нашому блокноті. Ми також створюємо a requirements.txt файл для визначення деяких залежностей Python для використання нашою кінцевою точкою. Ви можете переглянути створені нами файли в GitHub сховище.
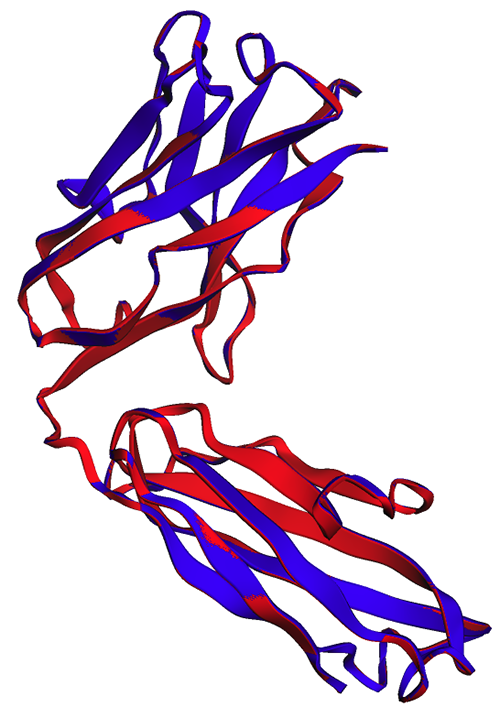
На наступному малюнку експериментальна (синій) і прогнозована (червоний) структури важкого ланцюга трастузумабу дуже схожі, але не ідентичні.

Після того як ми створили необхідні файли в code ми розгортаємо нашу модель за допомогою SageMaker HuggingFaceModel клас. Для цього використовується попередньо створений контейнер для спрощення процесу розгортання моделей Hugging Face у SageMaker. Зверніть увагу, що створення кінцевої точки може зайняти 10 хвилин або більше, залежно від доступності ml.g4dn типи примірників у нашому регіоні.
Коли розгортання кінцевої точки завершено, ми можемо повторно надіслати послідовність білка та відобразити кілька перших рядків прогнозу:
Оскільки ми розгорнули нашу кінцеву точку в прискореному екземплярі, передбачення має тривати лише кілька секунд. Кожен рядок результату відповідає одному атому та містить ідентичність амінокислоти, три просторові координати та оцінка pLDDT що представляє достовірність передбачення в цьому місці.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | ЗАСЕЛЕННЯ | PLDDT | ATOM_ID |
| ATOM | 1 | N | ГЛУ | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | ГЛУ | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | ГЛУ | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | ГЛУ | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | ГЛУ | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | ГЛУ | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | ГЛУ | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | ГЛУ | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | ГЛУ | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Використовуючи той самий метод, що й раніше, ми бачимо, що блокнот і передбачення кінцевої точки ідентичні.
| PDBchain1 | PDBchain2 | TM-Score |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
Як показано на наступному малюнку, передбачення ESMFold, створені в блокноті (червоний) і кінцевою точкою (синій), демонструють ідеальне вирівнювання.
Прибирати
Щоб уникнути подальших платежів, ми видаляємо нашу кінцеву точку висновку та дані тестування:
Підсумки
Обчислювальне прогнозування структури білків є критичним інструментом для розуміння функції білків. Окрім фундаментальних досліджень, такі алгоритми, як AlphaFold і ESMFold, мають багато застосувань у медицині та біотехнологіях. Структурні ідеї, отримані за допомогою цих моделей, допомагають нам краще зрозуміти, як взаємодіють біомолекули. Це може призвести до кращих діагностичних інструментів і терапії для пацієнтів.
У цій публікації ми показуємо, як розгорнути модель білкової мови ESMFold із Hugging Face Hub як масштабовану кінцеву точку висновку за допомогою SageMaker. Додаткову інформацію про розгортання моделей Hugging Face на SageMaker див Використовуйте Hugging Face з Amazon SageMaker. Ви також можете знайти більше наукових прикладів білків у Чудовий аналіз білка на AWS Репо GitHub. Будь ласка, залиште нам коментар, якщо є інші приклади, які ви хотіли б побачити!
Про авторів
 Браян Лоял є старшим архітектором рішень AI/ML у глобальній команді охорони здоров’я та наук про життя в Amazon Web Services. Він має понад 17 років досвіду в біотехнологіях і машинному навчанні, і він захоплений тим, що допомагає клієнтам вирішувати геномні та протеомні проблеми. У вільний час він любить готувати та їсти з друзями та родиною.
Браян Лоял є старшим архітектором рішень AI/ML у глобальній команді охорони здоров’я та наук про життя в Amazon Web Services. Він має понад 17 років досвіду в біотехнологіях і машинному навчанні, і він захоплений тим, що допомагає клієнтам вирішувати геномні та протеомні проблеми. У вільний час він любить готувати та їсти з друзями та родиною.
 Шаміка Аріяванса є архітектором спеціалістів із штучного інтелекту та ML у групі Global Healthcare and Life Sciences у Amazon Web Services. Він пристрасно працює з клієнтами, щоб прискорити впровадження штучного інтелекту та машинного навчання, надаючи технічне керівництво та допомагаючи їм впроваджувати інновації та створювати безпечні хмарні рішення на AWS. Поза роботою любить кататися на лижах і бездоріжжі.
Шаміка Аріяванса є архітектором спеціалістів із штучного інтелекту та ML у групі Global Healthcare and Life Sciences у Amazon Web Services. Він пристрасно працює з клієнтами, щоб прискорити впровадження штучного інтелекту та машинного навчання, надаючи технічне керівництво та допомагаючи їм впроваджувати інновації та створювати безпечні хмарні рішення на AWS. Поза роботою любить кататися на лижах і бездоріжжі.
 Яньцзюнь Ці є старшим менеджером із прикладних наук у лабораторії рішень машинного навчання AWS. Вона впроваджує інновації та застосовує машинне навчання, щоб допомогти клієнтам AWS прискорити впровадження ШІ та хмари.
Яньцзюнь Ці є старшим менеджером із прикладних наук у лабораторії рішень машинного навчання AWS. Вона впроваджує інновації та застосовує машинне навчання, щоб допомогти клієнтам AWS прискорити впровадження ШІ та хмари.
- Розповсюдження контенту та PR на основі SEO. Отримайте посилення сьогодні.
- PlatoAiStream. Web3 Data Intelligence. Розширення знань. Доступ тут.
- Карбування майбутнього з Адріенн Ешлі. Доступ тут.
- Купуйте та продавайте акції компаній, які вийшли на IPO, за допомогою PREIPO®. Доступ тут.
- джерело: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- : має
- :є
- : ні
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- МЕНЮ
- вище
- AC
- прискорювати
- прискорений
- точність
- точний
- точно
- досягнення
- діяльність
- доповнення
- Прийняття
- AI
- AI / ML
- алгоритм
- алгоритми
- вирівняні
- вирівнювання
- Також
- хоча
- Amazon
- Amazon SageMaker
- Amazon Web Services
- an
- аналіз
- та
- Інший
- будь-який
- додаток
- застосування
- прикладної
- відповідний
- ЕСТЬ
- AS
- Оцінювання
- At
- атом
- увагу
- автоматичний
- наявність
- уникнути
- AWS
- AWS Машинне навчання
- Хребет
- Банк
- основний
- BE
- перед тим
- починати
- Краще
- між
- обов'язковий
- біотехнології
- синій
- будувати
- але
- by
- CAN
- рак
- мультиплікація
- випадок
- випадків
- знаменитий
- ланцюг
- проблеми
- вантажі
- перевірка
- клас
- хмара
- прийняття хмари
- код
- color
- приходить
- коментар
- загальний
- порівняти
- порівняння
- повний
- довіра
- Контейнер
- контрастність
- відповідає
- рентабельним
- витрати
- може
- створювати
- створений
- критичний
- Клієнти
- дані
- базами даних
- дата, час
- глибокий
- глибоке навчання
- Deepmind
- Ступінь
- запити
- Залежно
- розгортання
- розгорнути
- розгортання
- розгортання
- Визначати
- певний
- розвиненою
- розвивається
- розробка
- пристрій
- важкий
- дисплей
- do
- робить
- Ні
- скачати
- управляти
- Наркотики
- під час
- кожен
- легко
- ефект
- ефекти
- дозволяє
- кінець
- Кінцева точка
- забезпечення
- Навколишнє середовище
- приклад
- Приклади
- дорогий
- досвід
- Експерименти
- зовнішній
- надзвичайно
- Face
- сім'я
- швидше
- кілька
- менше
- поле
- Рисунок
- філе
- Файли
- знайти
- кінець
- Перший
- після
- для
- формат
- друзі
- від
- функція
- далі
- породжувати
- генерується
- GitHub
- Глобальний
- зелений
- керівництво
- Мати
- he
- охорона здоров'я
- важкий
- допомога
- допомогу
- Високий
- його
- господар
- Як
- How To
- Однак
- HTML
- HTTPS
- Концентратор
- HuggingFace
- однаковий
- Особистість
- if
- зображення
- імпорт
- важливо
- удосконалювати
- поліпшений
- in
- includes
- вказувати
- вказує
- інформація
- оновлювати
- вхід
- розуміння
- розуміння
- екземпляр
- замість
- інтегрувати
- взаємодіяти
- Взаємодії
- інтерактивний
- взаємодіє
- в
- IT
- ЙОГО
- lab
- мова
- великий
- вести
- вивчення
- Залишати
- libraries
- бібліотека
- життя
- Life Sciences
- світло
- як
- Ймовірно
- загрузка
- розташування
- пошук
- машина
- навчання за допомогою машини
- зробити
- РОБОТИ
- менеджер
- багато
- матч
- Може..
- медицина
- Зустрічатися
- метод
- методика
- метрика
- Мічиган
- мільйони
- хвилин
- ML
- MLOps
- режим
- модель
- Моделі
- Поправки
- молекулярний
- більше
- багато
- ім'я
- необхідно
- Необхідність
- NIH
- ноутбук
- роман
- номер
- об'єкти
- застарілий
- of
- on
- ONE
- тільки
- працює
- оптимізуючий
- варіант
- or
- помаранчевий
- OS
- Інше
- наші
- вихід
- поза
- параметри
- пристрасний
- пацієнт
- pacientes
- моделі
- ідеальний
- продуктивність
- трубопровід
- plato
- Інформація про дані Платона
- PlatoData
- будь ласка
- пошта
- потенційно
- передбачати
- передвіщений
- прогнозування
- прогноз
- Прогнози
- Готувати
- раніше
- процес
- процеси
- Білок
- Білки
- забезпечувати
- забезпечує
- забезпечення
- Python
- піторх
- Qi
- швидко
- швидко
- поле, що швидко розвивається
- реального часу
- рекомендувати
- червоний
- регіон
- пов'язаний
- покладатися
- представляти
- представляє
- представляє
- вимагати
- дослідження
- Дослідники
- результат
- ROW
- прогін
- біг
- мудрець
- Висновок SageMaker
- то ж
- зберегти
- масштабовані
- Масштабування
- наука
- НАУКИ
- рахунок
- рахунок
- scripts
- seconds
- безпечний
- побачити
- старший
- Послідовність
- Послуги
- комплект
- кілька
- Поділитись
- вона
- Повинен
- Показувати
- показаний
- сторона
- аналогічний
- спростити
- один
- рішення
- Рішення
- ВИРІШИТИ
- деякі
- просторовий
- спеціаліст
- Спектроскопія
- швидкість
- старт
- впроваджений
- Крок
- структурний
- структура
- студія
- Вивчення
- такі
- підходящий
- підтримка
- виживання
- Приймати
- приймає
- цілі
- завдання
- команда
- команди
- технічний
- тест
- ніж
- Що
- Команда
- їх
- Їх
- потім
- Там.
- Ці
- це
- три
- тривимірний
- час
- трудомісткий
- times
- до
- токенізувати
- токенізований
- інструмент
- інструменти
- факел
- навчений
- Навчання
- Трансформатори
- перевезення
- лікування
- два
- тип
- Типи
- розуміти
- розуміння
- університет
- на відміну від
- us
- використання
- використовуваний
- використання
- ПЕРЕВІР
- дуже
- вид
- візуалізації
- W
- хотів
- шлях..
- we
- Web
- веб-сервіси
- добре відомі
- Що
- Вікіпедія
- волі
- з
- WordPress
- Work
- працює
- б
- рентгенівський
- Ти
- вашу
- зефірнет