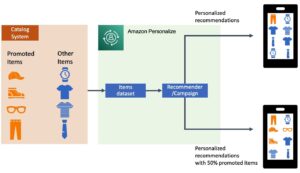
Protein thúc đẩy nhiều quá trình sinh học, chẳng hạn như hoạt động của enzyme, vận chuyển phân tử và hỗ trợ tế bào. Cấu trúc ba chiều của protein cung cấp cái nhìn sâu sắc về chức năng của nó và cách nó tương tác với các phân tử sinh học khác. Các phương pháp thí nghiệm để xác định cấu trúc protein, chẳng hạn như tinh thể học tia X và quang phổ NMR, rất tốn kém và tốn thời gian.
Ngược lại, các phương pháp tính toán được phát triển gần đây có thể dự đoán nhanh chóng và chính xác cấu trúc của protein từ trình tự axit amin của nó. Những phương pháp này rất quan trọng đối với các protein khó nghiên cứu bằng thực nghiệm, chẳng hạn như protein màng, mục tiêu của nhiều loại thuốc. Một ví dụ nổi tiếng về điều này là AlphaFold, một thuật toán dựa trên học sâu được tôn vinh vì những dự đoán chính xác của nó.
ESMF Gấp là một phương pháp dựa trên học sâu, có độ chính xác cao khác được phát triển để dự đoán cấu trúc protein từ trình tự axit amin của nó. ESMFold sử dụng mô hình ngôn ngữ protein lớn (pLM) làm xương sống và hoạt động từ đầu đến cuối. Không giống như AlphaFold2, nó không cần tra cứu hoặc Sắp xếp nhiều trình tự (MSA), cũng như không dựa vào cơ sở dữ liệu bên ngoài để tạo dự đoán. Thay vào đó, nhóm phát triển đã đào tạo mô hình trên hàng triệu chuỗi protein từ UniRef. Trong quá trình đào tạo, mô hình đã phát triển các mẫu chú ý thể hiện một cách trang nhã các tương tác tiến hóa giữa các axit amin trong chuỗi. Việc sử dụng pLM thay vì MSA này cho phép thời gian dự đoán nhanh hơn tới 60 lần so với các mô hình hiện đại khác.
Trong bài đăng này, chúng tôi sử dụng mô hình ESMFold được đào tạo trước từ Hugging Face with Amazon SageMaker để dự đoán cấu trúc chuỗi nặng của trastuzumab, Một kháng thể đơn dòng phát triển đầu tiên bởi Genentech để điều trị Ung thư vú dương tính với HER2. Dự đoán nhanh cấu trúc của protein này có thể hữu ích nếu các nhà nghiên cứu muốn kiểm tra tác động của việc sửa đổi trình tự. Điều này có khả năng dẫn đến cải thiện khả năng sống sót của bệnh nhân hoặc ít tác dụng phụ hơn.
Bài đăng này cung cấp sổ ghi chép ví dụ về Jupyter và các tập lệnh liên quan sau đây Kho GitHub.
Điều kiện tiên quyết
Chúng tôi khuyên bạn nên chạy ví dụ này trong một Xưởng sản xuất Amazon SageMaker máy tính xách tay chạy hình ảnh được tối ưu hóa cho CPU PyTorch 1.13 Python 3.9 trên loại phiên bản ml.r5.xlarge.
Hình dung cấu trúc thử nghiệm của trastuzumab
Để bắt đầu, chúng tôi sử dụng biopython thư viện và tập lệnh trợ giúp để tải xuống cấu trúc trastuzumab từ Ngân hàng Dữ liệu Protein RCSB:
Tiếp theo, chúng tôi sử dụng py3Dmol thư viện để trực quan hóa cấu trúc dưới dạng trực quan hóa 3D tương tác:
Hình dưới đây biểu thị cấu trúc protein 3D 1N8Z từ Ngân hàng Dữ liệu Protein (PDB). Trong hình ảnh này, chuỗi nhẹ trastuzumab có màu cam, chuỗi nặng có màu xanh lam (với vùng biến đổi có màu xanh lam nhạt) và kháng nguyên HER2 có màu xanh lá cây.
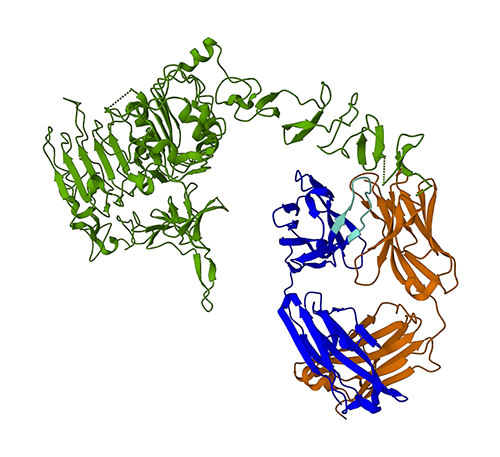
Trước tiên chúng ta sẽ sử dụng ESMFold để dự đoán cấu trúc của chuỗi nặng (Chuỗi B) từ trình tự axit amin của nó. Sau đó, chúng tôi sẽ so sánh dự đoán với cấu trúc được xác định bằng thực nghiệm được hiển thị ở trên.
Dự đoán cấu trúc chuỗi nặng trastuzumab từ trình tự của nó bằng ESMFold
Hãy sử dụng mô hình ESMFold để dự đoán cấu trúc của chuỗi nặng và so sánh nó với kết quả thực nghiệm. Để bắt đầu, chúng ta sẽ sử dụng môi trường sổ ghi chép dựng sẵn trong Studio đi kèm với một số thư viện quan trọng, chẳng hạn như Kim tự tháp, được cài đặt sẵn. Mặc dù chúng tôi có thể sử dụng loại phiên bản tăng tốc để cải thiện hiệu suất phân tích sổ ghi chép của mình, thay vào đó, chúng tôi sẽ sử dụng phiên bản không tăng tốc và chạy dự đoán ESMFold trên CPU.
Đầu tiên, chúng tôi tải mô hình ESMFold được đào tạo trước và mã thông báo từ Trung tâm khuôn mặt ôm:
Tiếp theo, chúng tôi sao chép mô hình vào thiết bị của mình (CPU trong trường hợp này) và đặt một số tham số mô hình:
Để chuẩn bị chuỗi protein để phân tích, chúng ta cần mã hóa nó. Điều này chuyển các ký hiệu axit amin (EVQLV…) sang định dạng số mà mô hình ESMFold có thể hiểu được (6,19,5,10,19,…):
Tiếp theo, chúng tôi sao chép đầu vào được mã hóa vào chế độ, đưa ra dự đoán và lưu kết quả vào một tệp:
Quá trình này mất khoảng 3 phút đối với loại phiên bản không tăng tốc, chẳng hạn như r5.
Chúng ta có thể kiểm tra tính chính xác của dự đoán ESMFold bằng cách so sánh nó với cấu trúc thử nghiệm. Chúng tôi làm điều này bằng cách sử dụng Căn chỉnh Hoa Kỳ công cụ được phát triển bởi Phòng thí nghiệm Zhang tại Đại học Michigan:
| PDBchain1 | PDBchain2 | TM-Điểm |
| dữ liệu/dự đoán.pdb:A | dữ liệu/thử nghiệm.pdb:B | 0.802 |
Sản phẩm điểm mô hình mẫu (TM-score) là thước đo để đánh giá mức độ giống nhau của cấu trúc protein. Điểm 1.0 cho thấy một kết hợp hoàn hảo. Điểm trên 0.7 cho thấy các protein có chung cấu trúc xương sống. Điểm trên 0.9 chỉ ra rằng các protein là chức năng hoán đổi cho nhau cho hạ lưu sử dụng. Trong trường hợp đạt được TM-Score 0.802 của chúng tôi, dự đoán ESMFold có thể phù hợp với các ứng dụng như chấm điểm cấu trúc hoặc thử nghiệm ràng buộc phối tử, nhưng có thể không phù hợp với các trường hợp sử dụng như thay thế phân tử đòi hỏi độ chính xác cực cao.
Chúng tôi có thể xác thực kết quả này bằng cách trực quan hóa các cấu trúc được căn chỉnh. Hai cấu trúc cho thấy mức độ chồng chéo cao, nhưng không hoàn hảo. Dự đoán cấu trúc protein là một lĩnh vực phát triển nhanh chóng và nhiều nhóm nghiên cứu đang phát triển các thuật toán ngày càng chính xác hơn!
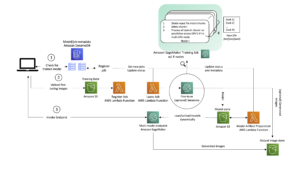
Triển khai ESMFold làm điểm cuối suy luận SageMaker
Chạy suy luận mô hình trong sổ ghi chép là phù hợp để thử nghiệm, nhưng nếu bạn cần tích hợp mô hình của mình với một ứng dụng thì sao? Hoặc một đường dẫn MLOps? Trong trường hợp này, tùy chọn tốt hơn là triển khai mô hình của bạn làm điểm cuối suy luận. Trong ví dụ sau, chúng tôi sẽ triển khai ESMFold dưới dạng điểm cuối suy luận thời gian thực của SageMaker trên một phiên bản được tăng tốc. Các điểm cuối thời gian thực của SageMaker cung cấp một cách an toàn, tiết kiệm chi phí và có thể mở rộng để triển khai và lưu trữ các mô hình máy học (ML). Với tính năng tự động thay đổi quy mô, bạn có thể điều chỉnh số lượng phiên bản đang chạy điểm cuối để đáp ứng nhu cầu của ứng dụng, tối ưu hóa chi phí và đảm bảo tính khả dụng cao.
Xây dựng trước Hộp đựng SageMaker dành cho Ôm mặt giúp dễ dàng triển khai các mô hình học sâu cho các tác vụ thông thường. Tuy nhiên, đối với các trường hợp sử dụng mới như dự đoán cấu trúc protein, chúng tôi cần xác định tùy chỉnh inference.py tập lệnh để tải mô hình, chạy dự đoán và định dạng đầu ra. Tập lệnh này bao gồm nhiều mã giống như chúng tôi đã sử dụng trong sổ ghi chép của mình. Chúng tôi cũng tạo ra một requirements.txt tệp để xác định một số phụ thuộc Python cho điểm cuối của chúng tôi sử dụng. Bạn có thể xem các tệp chúng tôi đã tạo trong Kho GitHub.
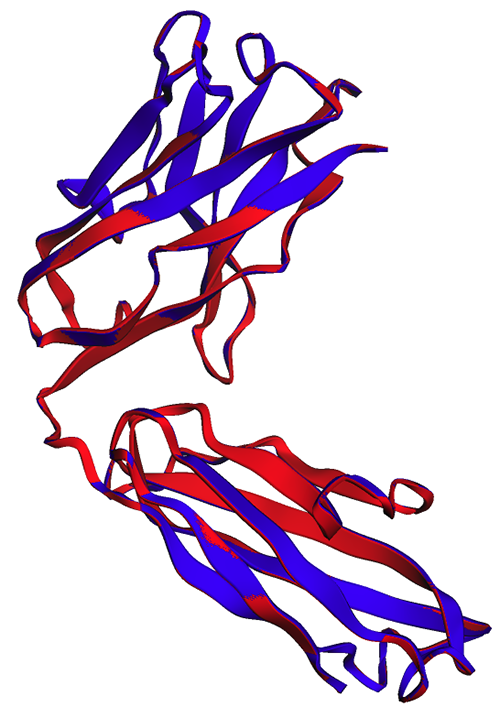
Trong hình dưới đây, cấu trúc thử nghiệm (màu xanh) và dự đoán (màu đỏ) của chuỗi nặng trastuzumab rất giống nhau, nhưng không đồng nhất.

Sau khi chúng tôi đã tạo các tệp cần thiết trong code thư mục, chúng tôi triển khai mô hình của mình bằng SageMaker HuggingFaceModel lớp học. Điều này sử dụng một vùng chứa dựng sẵn để đơn giản hóa quá trình triển khai các mô hình Hugging Face cho SageMaker. Lưu ý rằng có thể mất 10 phút trở lên để tạo điểm cuối, tùy thuộc vào tính khả dụng của ml.g4dn các loại phiên bản trong Khu vực của chúng tôi.
Khi quá trình triển khai điểm cuối hoàn tất, chúng tôi có thể gửi lại chuỗi protein và hiển thị một vài hàng đầu tiên của dự đoán:
Bởi vì chúng tôi đã triển khai điểm cuối của mình cho một phiên bản tăng tốc, dự đoán sẽ chỉ mất vài giây. Mỗi hàng trong kết quả tương ứng với một nguyên tử và bao gồm nhận dạng axit amin, ba tọa độ không gian và điểm pLDDT đại diện cho sự tự tin dự đoán tại vị trí đó.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | GIỎ HÀNG_X | GIỎ HÀNG_Y | GIỎ HÀNG_Z | NÂNG CAO | PLDDT | ATOM_ID |
| ATOM | 1 | N | G.L.U. | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | G.L.U. | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | G.L.U. | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | G.L.U. | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | G.L.U. | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | G.L.U. | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | G.L.U. | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | G.L.U. | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | G.L.U. | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Sử dụng phương pháp tương tự như trước đây, chúng tôi thấy rằng các dự đoán về sổ ghi chép và điểm cuối giống hệt nhau.
| PDBchain1 | PDBchain2 | TM-Điểm |
| dữ liệu/endpoint_prediction.pdb:A | dữ liệu/dự đoán.pdb:A | 1.0 |
Như được quan sát trong hình dưới đây, các dự đoán ESMFold được tạo trong sổ ghi chép (màu đỏ) và theo điểm cuối (màu xanh) hiển thị sự liên kết hoàn hảo.
Làm sạch
Để tránh bị tính thêm phí, chúng tôi xóa dữ liệu thử nghiệm và điểm cuối suy luận của mình:
Tổng kết
Dự đoán cấu trúc protein tính toán là một công cụ quan trọng để hiểu chức năng của protein. Ngoài nghiên cứu cơ bản, các thuật toán như AlphaFold và ESMFold còn có nhiều ứng dụng trong y học và công nghệ sinh học. Những hiểu biết sâu sắc về cấu trúc do các mô hình này tạo ra giúp chúng ta hiểu rõ hơn về cách các phân tử sinh học tương tác. Điều này sau đó có thể dẫn đến các công cụ chẩn đoán và điều trị tốt hơn cho bệnh nhân.
Trong bài đăng này, chúng tôi trình bày cách triển khai mô hình ngôn ngữ protein ESMFold từ Hugging Face Hub dưới dạng điểm cuối suy luận có thể mở rộng bằng SageMaker. Để biết thêm thông tin về việc triển khai các mô hình Hugging Face trên SageMaker, hãy tham khảo Sử dụng khuôn mặt ôm với Amazon SageMaker. Bạn cũng có thể tìm thêm các ví dụ về khoa học protein trong Phân tích Protein tuyệt vời trên AWS Kho lưu trữ GitHub. Vui lòng để lại nhận xét cho chúng tôi nếu có bất kỳ ví dụ nào khác mà bạn muốn xem!
Về các tác giả
 Brian trung thành là Kiến trúc sư giải pháp AI/ML cấp cao trong nhóm Khoa học đời sống và chăm sóc sức khỏe toàn cầu tại Amazon Web Services. Ông có hơn 17 năm kinh nghiệm trong lĩnh vực công nghệ sinh học và học máy, đồng thời đam mê giúp khách hàng giải quyết các thách thức về gen và protein. Trong thời gian rảnh rỗi, anh ấy thích nấu ăn và ăn uống với bạn bè và gia đình.
Brian trung thành là Kiến trúc sư giải pháp AI/ML cấp cao trong nhóm Khoa học đời sống và chăm sóc sức khỏe toàn cầu tại Amazon Web Services. Ông có hơn 17 năm kinh nghiệm trong lĩnh vực công nghệ sinh học và học máy, đồng thời đam mê giúp khách hàng giải quyết các thách thức về gen và protein. Trong thời gian rảnh rỗi, anh ấy thích nấu ăn và ăn uống với bạn bè và gia đình.
 Shamika Ariyawansa là Kiến trúc sư giải pháp chuyên gia AI/ML trong nhóm Khoa học đời sống và chăm sóc sức khỏe toàn cầu tại Amazon Web Services. Anh nhiệt tình làm việc với khách hàng để đẩy nhanh quá trình áp dụng AI và ML của họ bằng cách cung cấp hướng dẫn kỹ thuật, đồng thời giúp họ đổi mới và xây dựng các giải pháp đám mây an toàn trên AWS. Ngoài công việc, anh ấy thích trượt tuyết và địa hình.
Shamika Ariyawansa là Kiến trúc sư giải pháp chuyên gia AI/ML trong nhóm Khoa học đời sống và chăm sóc sức khỏe toàn cầu tại Amazon Web Services. Anh nhiệt tình làm việc với khách hàng để đẩy nhanh quá trình áp dụng AI và ML của họ bằng cách cung cấp hướng dẫn kỹ thuật, đồng thời giúp họ đổi mới và xây dựng các giải pháp đám mây an toàn trên AWS. Ngoài công việc, anh ấy thích trượt tuyết và địa hình.
 Yên Quân Tề là Giám đốc khoa học ứng dụng cấp cao tại AWS Machine Learning Solution Lab. Cô đổi mới và áp dụng công nghệ máy học để giúp các khách hàng của AWS tăng tốc độ tiếp nhận AI và đám mây của họ.
Yên Quân Tề là Giám đốc khoa học ứng dụng cấp cao tại AWS Machine Learning Solution Lab. Cô đổi mới và áp dụng công nghệ máy học để giúp các khách hàng của AWS tăng tốc độ tiếp nhận AI và đám mây của họ.
- Phân phối nội dung và PR được hỗ trợ bởi SEO. Được khuếch đại ngay hôm nay.
- PlatoAiStream. Thông minh dữ liệu Web3. Kiến thức khuếch đại. Truy cập Tại đây.
- Đúc kết tương lai với Adryenn Ashley. Truy cập Tại đây.
- Mua và bán cổ phần trong các công ty PRE-IPO với PREIPO®. Truy cập Tại đây.
- nguồn: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- : có
- :là
- :không phải
- $ LÊN
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Giới thiệu
- ở trên
- AC
- đẩy nhanh tiến độ
- tăng tốc
- chính xác
- chính xác
- chính xác
- đạt được
- hoạt động
- Ngoài ra
- Nhận con nuôi
- AI
- AI / ML
- thuật toán
- thuật toán
- căn chỉnh
- liên kết
- Ngoài ra
- Mặc dù
- đàn bà gan dạ
- Amazon SageMaker
- Amazon Web Services
- an
- phân tích
- và
- Một
- bất kì
- Các Ứng Dụng
- các ứng dụng
- áp dụng
- thích hợp
- LÀ
- AS
- Đánh giá
- At
- nguyên tử
- sự chú ý
- Tự động
- sẵn có
- tránh
- AWS
- Học máy AWS
- Xương sống
- Ngân hàng
- cơ bản
- BE
- trước
- bắt đầu
- Hơn
- giữa
- ràng buộc
- công nghệ sinh học
- Màu xanh da trời
- xây dựng
- nhưng
- by
- CAN
- Ung thư
- hoạt hình
- trường hợp
- trường hợp
- nổi tiếng
- chuỗi
- thách thức
- tải
- kiểm tra
- tốt nghiệp lớp XNUMX
- đám mây
- áp dụng đám mây
- mã
- màu sắc
- đến
- bình luận
- Chung
- so sánh
- so sánh
- hoàn thành
- sự tự tin
- Container
- Ngược lại
- tương ứng
- chi phí-hiệu quả
- Chi phí
- có thể
- tạo
- tạo ra
- quan trọng
- khách hàng
- dữ liệu
- cơ sở dữ liệu
- ngày giờ
- sâu
- học kĩ càng
- Deepmind
- Bằng cấp
- nhu cầu
- Tùy
- triển khai
- triển khai
- triển khai
- triển khai
- Xác định
- xác định
- phát triển
- phát triển
- Phát triển
- thiết bị
- khó khăn
- Giao diện
- do
- làm
- Không
- tải về
- lái xe
- Thuốc
- suốt trong
- mỗi
- dễ dàng
- hiệu lực
- hiệu ứng
- cho phép
- cuối
- Điểm cuối
- đảm bảo
- Môi trường
- ví dụ
- ví dụ
- đắt tiền
- kinh nghiệm
- thí nghiệm
- ngoài
- cực kỳ
- Đối mặt
- gia đình
- nhanh hơn
- vài
- ít hơn
- lĩnh vực
- Hình
- Tập tin
- Các tập tin
- Tìm kiếm
- cuối
- Tên
- tiếp theo
- Trong
- định dạng
- bạn bè
- từ
- chức năng
- xa hơn
- tạo ra
- tạo ra
- GitHub
- Toàn cầu
- màu xanh lá
- hướng dẫn
- Có
- he
- chăm sóc sức khỏe
- nặng
- giúp đỡ
- giúp đỡ
- Cao
- của mình
- chủ nhà
- Độ đáng tin của
- Hướng dẫn
- Tuy nhiên
- HTML
- HTTPS
- Hub
- ÔmKhuôn Mặt
- giống hệt nhau
- Bản sắc
- if
- hình ảnh
- nhập khẩu
- quan trọng
- nâng cao
- cải thiện
- in
- bao gồm
- chỉ
- chỉ
- thông tin
- đổi mới
- đầu vào
- cái nhìn sâu sắc
- những hiểu biết
- ví dụ
- thay vì
- tích hợp
- tương tác
- tương tác
- tương tác
- tương tác
- trong
- IT
- ITS
- phòng thí nghiệm
- Ngôn ngữ
- lớn
- dẫn
- học tập
- Rời bỏ
- thư viện
- Thư viện
- Cuộc sống
- Khoa học đời sống
- ánh sáng
- Lượt thích
- Có khả năng
- tải
- địa điểm thư viện nào
- tra cứu
- máy
- học máy
- làm cho
- LÀM CHO
- giám đốc
- nhiều
- Trận đấu
- Có thể..
- y học
- Gặp gỡ
- phương pháp
- phương pháp
- số liệu
- Michigan
- hàng triệu
- Phút
- ML
- MLOps
- Chế độ
- kiểu mẫu
- mô hình
- Sửa đổi
- phân tử
- chi tiết
- nhiều
- tên
- cần thiết
- Cần
- NIH
- máy tính xách tay
- tiểu thuyết
- con số
- đối tượng
- lỗi thời
- of
- on
- ONE
- có thể
- hoạt động
- tối ưu hóa
- Tùy chọn
- or
- trái cam
- OS
- Nền tảng khác
- vfoXNUMXfipXNUMXhfpiXNUMXufhpiXNUMXuf
- đầu ra
- bên ngoài
- thông số
- đam mê
- bệnh nhân
- bệnh nhân
- mô hình
- hoàn hảo
- hiệu suất
- đường ống dẫn
- plato
- Thông tin dữ liệu Plato
- PlatoDữ liệu
- xin vui lòng
- Bài đăng
- có khả năng
- dự đoán
- dự đoán
- dự đoán
- dự đoán
- Dự đoán
- Chuẩn bị
- trước đây
- quá trình
- Quy trình
- Protein
- Protein
- cho
- cung cấp
- cung cấp
- Python
- ngọn đuốc
- Qi
- Mau
- nhanh chóng
- lĩnh vực phát triển nhanh chóng
- thời gian thực
- giới thiệu
- đỏ
- khu
- liên quan
- dựa
- đại diện
- đại diện
- đại diện cho
- yêu cầu
- nghiên cứu
- nhà nghiên cứu
- kết quả
- HÀNG
- chạy
- chạy
- nhà làm hiền triết
- Suy luận của SageMaker
- tương tự
- Lưu
- khả năng mở rộng
- mở rộng quy mô
- Khoa học
- KHOA HỌC
- Điểm số
- ghi bàn
- kịch bản
- giây
- an toàn
- xem
- cao cấp
- Trình tự
- DỊCH VỤ
- định
- một số
- Chia sẻ
- chị ấy
- nên
- hiển thị
- thể hiện
- bên
- tương tự
- đơn giản hóa
- duy nhất
- giải pháp
- Giải pháp
- động SOLVE
- một số
- không gian
- chuyên gia
- Quang phổ
- tốc độ
- Bắt đầu
- nhà nước-of-the-art
- Bước
- cấu trúc
- cấu trúc
- phòng thu
- Học tập
- như vậy
- phù hợp
- hỗ trợ
- sống còn
- Hãy
- mất
- mục tiêu
- nhiệm vụ
- nhóm
- đội
- Kỹ thuật
- thử nghiệm
- hơn
- việc này
- Sản phẩm
- cung cấp their dịch
- Them
- sau đó
- Đó
- Kia là
- điều này
- số ba
- ba chiều
- thời gian
- mất thời gian
- thời gian
- đến
- token hóa
- được mã hóa
- công cụ
- công cụ
- ngọn đuốc
- đào tạo
- Hội thảo
- máy biến áp
- vận chuyển
- điều trị
- hai
- kiểu
- loại
- hiểu
- sự hiểu biết
- trường đại học
- không giống
- us
- sử dụng
- đã sử dụng
- sử dụng
- HIỆU LỰC
- rất
- Xem
- hình dung
- W
- muốn
- Đường..
- we
- web
- các dịch vụ web
- nổi tiếng
- Điều gì
- Wikipedia
- sẽ
- với
- WordPress
- Công việc
- công trinh
- sẽ
- X-quang
- Bạn
- trên màn hình
- zephyrnet