Proteine treiben viele biologische Prozesse an, wie Enzymaktivität, molekularen Transport und zelluläre Unterstützung. Die dreidimensionale Struktur eines Proteins gibt Aufschluss über seine Funktion und wie es mit anderen Biomolekülen interagiert. Experimentelle Methoden zur Bestimmung der Proteinstruktur, wie Röntgenkristallographie und NMR-Spektroskopie, sind teuer und zeitaufwändig.
Im Gegensatz dazu können kürzlich entwickelte Computerverfahren schnell und genau die Struktur eines Proteins aus seiner Aminosäuresequenz vorhersagen. Diese Methoden sind entscheidend für Proteine, die experimentell schwer zu untersuchen sind, wie z. B. Membranproteine, die Ziele vieler Medikamente. Ein bekanntes Beispiel dafür ist AlphaFold, ein auf Deep Learning basierender Algorithmus, der für seine genauen Vorhersagen gefeiert wird.
ESMFold ist eine weitere hochgenaue, auf Deep Learning basierende Methode, die entwickelt wurde, um die Proteinstruktur anhand ihrer Aminosäuresequenz vorherzusagen. ESMFold verwendet ein Large Protein Language Model (pLM) als Backbone und arbeitet End-to-End. Im Gegensatz zu AlphaFold2 benötigt es keine Suche oder Ausrichtung mehrerer Sequenzen (MSA) und stützt sich auch nicht auf externe Datenbanken, um Vorhersagen zu generieren. Stattdessen trainierte das Entwicklungsteam das Modell mit Millionen von Proteinsequenzen aus UniRef. Während des Trainings entwickelte das Modell Aufmerksamkeitsmuster, die die evolutionären Wechselwirkungen zwischen Aminosäuren in der Sequenz elegant darstellen. Diese Verwendung eines pLM anstelle eines MSA ermöglicht bis zu 60-mal schnellere Vorhersagezeiten als andere hochmoderne Modelle.
In diesem Beitrag verwenden wir das vortrainierte ESMFold-Modell von Hugging Face mit Amazon Sage Maker um die Struktur der schweren Kette vorherzusagen Trastuzumab, eine monoklonaler Antikörper zuerst entwickelt von Genentech zur Behandlung von HER2-positiver Brustkrebs. Die schnelle Vorhersage der Struktur dieses Proteins könnte nützlich sein, wenn Forscher die Wirkung von Sequenzmodifikationen testen wollten. Dies könnte möglicherweise zu einem verbesserten Überleben der Patienten oder weniger Nebenwirkungen führen.
Dieser Beitrag enthält im Folgenden ein Beispiel für ein Jupyter-Notebook und zugehörige Skripts GitHub-Repository.
Voraussetzungen:
Wir empfehlen, dieses Beispiel in einem auszuführen Amazon SageMaker-Studio Notizbuch Ausführen des CPU-optimierten PyTorch 1.13 Python 3.9-Images auf einem ml.r5.xlarge-Instance-Typ.
Visualisieren Sie die experimentelle Struktur von Trastuzumab
Zu Beginn verwenden wir die biopython Bibliothek und ein Hilfsskript zum Herunterladen der Trastuzumab-Struktur von der RCSB-Proteindatenbank:
Als nächstes verwenden wir die py3Dmol Bibliothek zur Visualisierung der Struktur als interaktive 3D-Visualisierung:
Die folgende Abbildung zeigt die 3D-Proteinstruktur 1N8Z aus der Protein Data Bank (PDB). In diesem Bild ist die leichte Kette von Trastuzumab orange dargestellt, die schwere Kette blau (mit der variablen Region hellblau) und das HER2-Antigen grün.
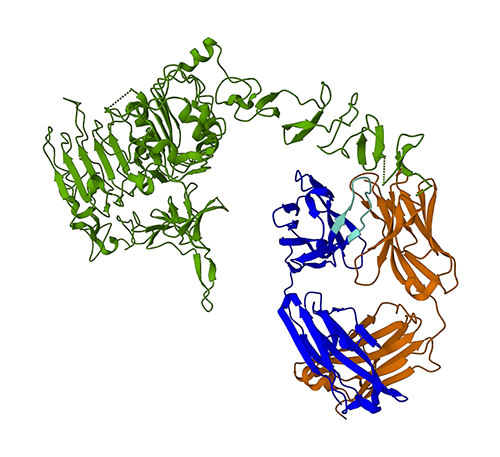
Wir werden zuerst ESMFold verwenden, um die Struktur der schweren Kette (Kette B) aus ihrer Aminosäuresequenz vorherzusagen. Dann werden wir die Vorhersage mit der oben gezeigten experimentell bestimmten Struktur vergleichen.
Prognostizieren Sie die Struktur der schweren Kette von Trastuzumab anhand ihrer Sequenz mit ESMFold
Lassen Sie uns das ESMFold-Modell verwenden, um die Struktur der schweren Kette vorherzusagen und mit dem experimentellen Ergebnis vergleichen. Zu Beginn verwenden wir eine vorgefertigte Notebook-Umgebung in Studio, die mit mehreren wichtigen Bibliotheken geliefert wird, wie z PyTorch, vorinstalliert. Obwohl wir einen beschleunigten Instance-Typ verwenden könnten, um die Leistung unserer Notebook-Analyse zu verbessern, verwenden wir stattdessen eine nicht beschleunigte Instance und führen die ESMFold-Vorhersage auf einer CPU aus.
Zuerst laden wir das vortrainierte ESMFold-Modell und den Tokenizer aus Face Hub umarmen:
Als nächstes kopieren wir das Modell auf unser Gerät (CPU in diesem Fall) und stellen einige Modellparameter ein:
Um die Proteinsequenz für die Analyse vorzubereiten, müssen wir sie tokenisieren. Dies übersetzt die Aminosäuresymbole (EVQLV…) in ein numerisches Format, das das ESMFold-Modell verstehen kann (6,19,5,10,19,…):
Als nächstes kopieren wir die tokenisierte Eingabe in den Modus, machen eine Vorhersage und speichern das Ergebnis in einer Datei:
Dies dauert auf einem nicht beschleunigten Instance-Typ, wie z. B. einem r3, etwa 5 Minuten.
Wir können die Genauigkeit der ESMFold-Vorhersage überprüfen, indem wir sie mit der experimentellen Struktur vergleichen. Wir machen das mit der US-Align Tool entwickelt vom Zhang Lab an der University of Michigan:
| PDBchain1 | PDBchain2 | TM-Score |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
Das Partitur für Vorlagenmodellierung (TM-Score) ist ein Maß zur Beurteilung der Ähnlichkeit von Proteinstrukturen. Eine Punktzahl von 1.0 bedeutet eine perfekte Übereinstimmung. Werte über 0.7 zeigen an, dass Proteine dieselbe Rückgratstruktur aufweisen. Werte über 0.9 zeigen an, dass die Proteine sind funktional austauschbar für nachgeschaltete Verwendung. In unserem Fall des Erreichens von TM-Score 0.802 wäre die ESMFold-Vorhersage wahrscheinlich für Anwendungen wie Strukturbewertung oder Ligandenbindungsexperimente geeignet, aber möglicherweise nicht für Anwendungsfälle wie molekularer Ersatz die eine extrem hohe Genauigkeit erfordern.
Wir können dieses Ergebnis validieren, indem wir die ausgerichteten Strukturen visualisieren. Die beiden Strukturen weisen einen hohen, aber nicht perfekten Überlappungsgrad auf. Proteinstrukturvorhersagen sind ein sich schnell entwickelndes Gebiet und viele Forschungsteams entwickeln immer genauere Algorithmen!
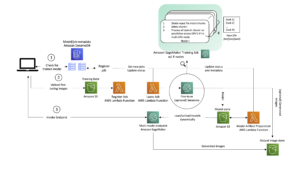
Stellen Sie ESMFold als SageMaker-Inferenzendpunkt bereit
Das Ausführen von Modellrückschlüssen in einem Notebook ist zum Experimentieren in Ordnung, aber was ist, wenn Sie Ihr Modell in eine Anwendung integrieren müssen? Oder eine MLOps-Pipeline? In diesem Fall ist es besser, Ihr Modell als Inferenzendpunkt bereitzustellen. Im folgenden Beispiel stellen wir ESMFold als SageMaker-Echtzeit-Inferenzendpunkt auf einer beschleunigten Instanz bereit. SageMaker-Echtzeit-Endpunkte bieten eine skalierbare, kostengünstige und sichere Möglichkeit, Modelle für maschinelles Lernen (ML) bereitzustellen und zu hosten. Mit der automatischen Skalierung können Sie die Anzahl der Instanzen anpassen, auf denen der Endpunkt ausgeführt wird, um die Anforderungen Ihrer Anwendung zu erfüllen, die Kosten zu optimieren und eine hohe Verfügbarkeit sicherzustellen.
Das vorgefertigte SageMaker-Container für Hugging Face erleichtert die Bereitstellung von Deep-Learning-Modellen für allgemeine Aufgaben. Für neuartige Anwendungsfälle wie die Vorhersage der Proteinstruktur müssen wir jedoch eine Gewohnheit definieren inference.py -Skript, um das Modell zu laden, die Vorhersage auszuführen und die Ausgabe zu formatieren. Dieses Skript enthält einen Großteil des gleichen Codes, den wir in unserem Notebook verwendet haben. Wir erstellen auch eine requirements.txt -Datei, um einige Python-Abhängigkeiten zu definieren, die unser Endpunkt verwenden soll. Sie können die von uns erstellten Dateien im sehen GitHub-Repository.
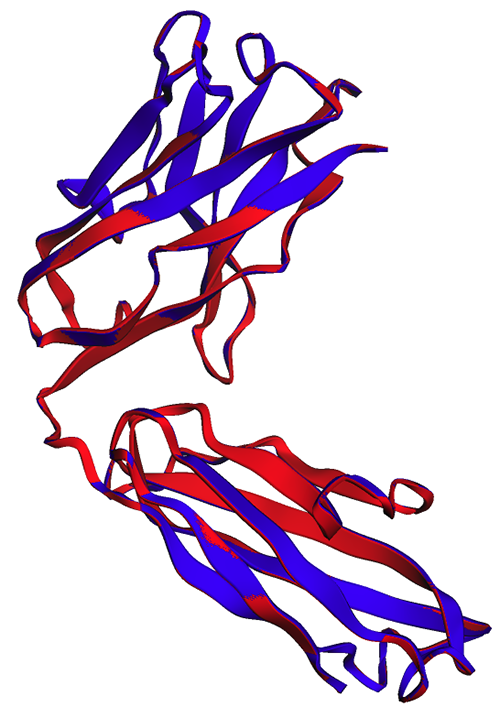
In der folgenden Abbildung sind die experimentelle (blau) und vorhergesagte (rot) Struktur der schweren Kette von Trastuzumab sehr ähnlich, aber nicht identisch.

Nachdem wir die notwendigen Dateien in der code Verzeichnis stellen wir unser Modell mit SageMaker bereit HuggingFaceModel Klasse. Dabei wird ein vorgefertigter Container verwendet, um den Prozess der Bereitstellung von Hugging Face-Modellen in SageMaker zu vereinfachen. Beachten Sie, dass das Erstellen des Endpunkts je nach Verfügbarkeit 10 Minuten oder länger dauern kann ml.g4dn Instanztypen in unserer Region.
Wenn die Bereitstellung des Endpunkts abgeschlossen ist, können wir die Proteinsequenz erneut übermitteln und die ersten paar Zeilen der Vorhersage anzeigen:
Da wir unseren Endpunkt auf einer beschleunigten Instanz bereitgestellt haben, sollte die Vorhersage nur wenige Sekunden dauern. Jede Zeile im Ergebnis entspricht einem einzelnen Atom und enthält die Aminosäureidentität, drei räumliche Koordinaten und a pLDDT-Score die Vorhersagekonfidenz an diesem Ort darstellt.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | WARENKORB_X | CARTN_Y | CARTN_Z | BELEGUNG | PLDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | -19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | -19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | -18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | -18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | -17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | -18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | -18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | -17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | -17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | -19.063 | -0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | -18.309 | -1.415 | 1 | 0.87 | C |
Mit der gleichen Methode wie zuvor sehen wir, dass die Notebook- und Endpunktvorhersagen identisch sind.
| PDBchain1 | PDBchain2 | TM-Score |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
Wie in der folgenden Abbildung zu sehen ist, zeigen die im Notebook (rot) und vom Endpunkt (blau) generierten ESMFold-Vorhersagen eine perfekte Ausrichtung.
Aufräumen
Um weitere Gebühren zu vermeiden, löschen wir unsere Inferenzendpunkt- und Testdaten:
Zusammenfassung
Die rechnergestützte Proteinstrukturvorhersage ist ein entscheidendes Werkzeug zum Verständnis der Funktion von Proteinen. Neben der Grundlagenforschung finden Algorithmen wie AlphaFold und ESMFold viele Anwendungen in Medizin und Biotechnologie. Die durch diese Modelle gewonnenen strukturellen Erkenntnisse helfen uns, besser zu verstehen, wie Biomoleküle interagieren. Dies kann dann zu besseren Diagnosewerkzeugen und Therapien für Patienten führen.
In diesem Beitrag zeigen wir, wie das ESMFold-Protein-Sprachmodell von Hugging Face Hub als skalierbarer Inferenzendpunkt mit SageMaker bereitgestellt wird. Weitere Informationen zum Bereitstellen von Hugging Face-Modellen in SageMaker finden Sie unter Verwenden Sie Hugging Face mit Amazon SageMaker. Weitere proteinwissenschaftliche Beispiele finden Sie auch in der Fantastische Proteinanalyse auf AWS GitHub-Repository. Bitte hinterlassen Sie uns einen Kommentar, wenn Sie weitere Beispiele sehen möchten!
Über die Autoren
 Brian Treue ist Senior AI/ML Solutions Architect im Team Global Healthcare and Life Sciences bei Amazon Web Services. Er verfügt über mehr als 17 Jahre Erfahrung in Biotechnologie und maschinellem Lernen und unterstützt Kunden leidenschaftlich gerne bei der Lösung genomischer und proteomischer Herausforderungen. In seiner Freizeit kocht und isst er gerne mit seinen Freunden und seiner Familie.
Brian Treue ist Senior AI/ML Solutions Architect im Team Global Healthcare and Life Sciences bei Amazon Web Services. Er verfügt über mehr als 17 Jahre Erfahrung in Biotechnologie und maschinellem Lernen und unterstützt Kunden leidenschaftlich gerne bei der Lösung genomischer und proteomischer Herausforderungen. In seiner Freizeit kocht und isst er gerne mit seinen Freunden und seiner Familie.
 Shamika Ariyawansa ist AI/ML Specialist Solutions Architect im Team Global Healthcare and Life Sciences bei Amazon Web Services. Er arbeitet leidenschaftlich mit Kunden zusammen, um ihre KI- und ML-Einführung zu beschleunigen, indem er technische Anleitungen gibt und ihnen hilft, Innovationen zu entwickeln und sichere Cloud-Lösungen auf AWS zu entwickeln. Außerhalb der Arbeit liebt er Skifahren und Geländefahrten.
Shamika Ariyawansa ist AI/ML Specialist Solutions Architect im Team Global Healthcare and Life Sciences bei Amazon Web Services. Er arbeitet leidenschaftlich mit Kunden zusammen, um ihre KI- und ML-Einführung zu beschleunigen, indem er technische Anleitungen gibt und ihnen hilft, Innovationen zu entwickeln und sichere Cloud-Lösungen auf AWS zu entwickeln. Außerhalb der Arbeit liebt er Skifahren und Geländefahrten.
 Yanjun Qi ist Senior Applied Science Manager im AWS Machine Learning Solution Lab. Sie ist innovativ und wendet maschinelles Lernen an, um AWS-Kunden dabei zu helfen, ihre KI- und Cloud-Einführung zu beschleunigen.
Yanjun Qi ist Senior Applied Science Manager im AWS Machine Learning Solution Lab. Sie ist innovativ und wendet maschinelles Lernen an, um AWS-Kunden dabei zu helfen, ihre KI- und Cloud-Einführung zu beschleunigen.
- SEO-gestützte Content- und PR-Distribution. Holen Sie sich noch heute Verstärkung.
- PlatoAiStream. Web3-Datenintelligenz. Wissen verstärkt. Hier zugreifen.
- Die Zukunft prägen mit Adryenn Ashley. Hier zugreifen.
- Kaufen und verkaufen Sie Anteile an PRE-IPO-Unternehmen mit PREIPO®. Hier zugreifen.
- Quelle: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :hast
- :Ist
- :nicht
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- Über Uns
- oben
- AC
- beschleunigen
- beschleunigt
- Genauigkeit
- genau
- genau
- Erreichen
- Aktivität
- Zusatz
- Adoption
- AI
- AI / ML
- Algorithmus
- Algorithmen
- ausgerichtet
- Ausrichtung
- ebenfalls
- Obwohl
- Amazon
- Amazon Sage Maker
- Amazon Web Services
- an
- Analyse
- und
- Ein anderer
- jedem
- Anwendung
- Anwendungen
- angewandt
- angemessen
- SIND
- AS
- Beurteilung
- At
- Atom
- Aufmerksamkeit
- automatische
- Verfügbarkeit
- vermeiden
- AWS
- AWS Maschinelles Lernen
- Backbone (Rückgrat)
- Bank
- basic
- BE
- Bevor
- beginnen
- Besser
- zwischen
- Bindung
- Biotechnologie
- Blau
- bauen
- aber
- by
- CAN
- Krebs
- Karikatur
- Häuser
- Fälle
- Gefeiert
- Kette
- Herausforderungen
- Gebühren
- aus der Ferne überprüfen
- Klasse
- Cloud
- Cloud-Einführung
- Code
- Farbe
- kommt
- Kommentar
- gemeinsam
- vergleichen
- Vergleich
- abschließen
- Vertrauen
- Container
- Kontrast
- entspricht
- kostengünstiger
- Kosten
- könnte
- erstellen
- erstellt
- kritischem
- Kunden
- technische Daten
- Datenbanken
- datetime
- tief
- tiefe Lernen
- DeepMind
- Grad
- Anforderungen
- Abhängig
- einsetzen
- Einsatz
- Bereitstellen
- Einsatz
- Bestimmen
- entschlossen
- entwickelt
- Entwicklung
- Entwicklung
- Gerät
- schwer
- Display
- do
- die
- Tut nicht
- herunterladen
- Antrieb
- Drogen
- im
- jeder
- Einfache
- bewirken
- Effekten
- ermöglicht
- Ende
- Endpunkt
- Gewährleistung
- Arbeitsumfeld
- Beispiel
- Beispiele
- teuer
- ERFAHRUNGEN
- Experimente
- extern
- äußerst
- Gesicht
- Familie
- beschleunigt
- wenige
- Weniger
- Feld
- Abbildung
- Reichen Sie das
- Mappen
- Finden Sie
- Ende
- Vorname
- Folgende
- Aussichten für
- Format
- Freunde
- für
- Funktion
- weiter
- erzeugen
- erzeugt
- GitHub
- Global
- Grün
- die Vermittlung von Kompetenzen,
- Haben
- he
- Gesundheitswesen
- schwer
- Hilfe
- Unternehmen
- GUTE
- seine
- Gastgeber
- Ultraschall
- Hilfe
- aber
- HTML
- HTTPS
- Nabe
- Umarmendes Gesicht
- identisch
- Identitätsschutz
- if
- Image
- importieren
- wichtig
- zu unterstützen,
- verbessert
- in
- Dazu gehören
- zeigen
- zeigt
- Information
- wir innovieren
- Varianten des Eingangssignals:
- Einblick
- Einblicke
- Instanz
- beantragen müssen
- integrieren
- interagieren
- Interaktionen
- interaktive
- interagiert
- in
- IT
- SEINE
- Labor
- Sprache
- grosse
- führen
- lernen
- Verlassen
- Bibliotheken
- Bibliothek
- Lebensdauer
- Biowissenschaften
- !
- Gefällt mir
- wahrscheinlich
- Belastung
- Standorte
- Nachschlagen
- Maschine
- Maschinelles Lernen
- um
- MACHT
- Manager
- viele
- Spiel
- Kann..
- Medizin
- Triff
- Methode
- Methoden
- Metrisch
- Michigan
- Millionen
- Minuten
- ML
- MLOps
- Model
- Modell
- für
- Änderungen
- Molekular-
- mehr
- viel
- Name
- notwendig,
- Need
- NIH
- Notizbuch
- Roman
- Anzahl
- Objekte
- überholt
- of
- on
- EINEM
- einzige
- arbeitet
- Optimierung
- Option
- or
- Orange
- OS
- Andere
- UNSERE
- Möglichkeiten für das Ausgangssignal:
- aussen
- Parameter
- leidenschaftlich
- Vertrauen bei Patienten
- Patienten
- Muster
- perfekt
- Leistung
- Pipeline
- Plato
- Datenintelligenz von Plato
- PlatoData
- Bitte
- Post
- möglicherweise
- vorhersagen
- vorhergesagt
- Vorhersage
- Prognose
- Prognosen
- Danach
- vorher
- Prozessdefinierung
- anpassen
- Proteine
- Proteine
- die
- bietet
- Bereitstellung
- Python
- Pytorch
- Qi
- schnell
- schnell
- sich schnell entwickelndes Feld
- Echtzeit
- empfehlen
- Rot
- Region
- bezogene
- verlassen
- vertreten
- Darstellen
- representiert
- erfordern
- Forschungsprojekte
- Forscher
- Folge
- REIHE
- Führen Sie
- Laufen
- sagemaker
- SageMaker-Inferenz
- gleich
- Speichern
- skalierbaren
- Skalierung
- Wissenschaft
- WISSENSCHAFTEN
- Ergebnis
- Wertung
- Skripte
- Sekunden
- Verbindung
- sehen
- Senior
- Reihenfolge
- Lösungen
- kompensieren
- mehrere
- Teilen
- sie
- sollte
- erklären
- gezeigt
- Seite
- ähnlich
- vereinfachen
- Single
- Lösung
- Lösungen
- LÖSEN
- einige
- räumlich
- Spezialist
- Spektroskopie
- Geschwindigkeit
- Anfang
- State-of-the-art
- Schritt
- strukturell
- Struktur
- Studio Adressen
- Studie
- so
- geeignet
- Support
- Überleben
- Nehmen
- nimmt
- Ziele
- und Aufgaben
- Team
- Teams
- Technische
- Test
- als
- zur Verbesserung der Gesundheitsgerechtigkeit
- Das
- ihr
- Sie
- dann
- Dort.
- Diese
- fehlen uns die Worte.
- nach drei
- dreidimensional
- Zeit
- Zeitaufwendig
- mal
- zu
- tokenisieren
- Tokenisiert
- Werkzeug
- Werkzeuge
- Fackel
- trainiert
- Ausbildung
- Transformer
- Transportwesen
- Behandlungen
- XNUMX
- tippe
- Typen
- verstehen
- Verständnis
- Universität
- nicht wie
- us
- -
- benutzt
- Verwendung von
- BESTÄTIGEN
- sehr
- Anzeigen
- Visualisierung
- W
- wollte
- Weg..
- we
- Netz
- Web-Services
- bekannt
- Was
- Wikipedia
- werden wir
- mit
- WordPress
- Arbeiten
- Werk
- würde
- Röntgenstrahl
- Du
- Ihr
- Zephyrnet