Les protéines pilotent de nombreux processus biologiques, tels que l'activité enzymatique, le transport moléculaire et le support cellulaire. La structure tridimensionnelle d'une protéine donne un aperçu de sa fonction et de la façon dont elle interagit avec d'autres biomolécules. Les méthodes expérimentales pour déterminer la structure des protéines, telles que la cristallographie aux rayons X et la spectroscopie RMN, sont coûteuses et prennent du temps.
En revanche, les méthodes de calcul récemment développées peuvent prédire rapidement et avec précision la structure d'une protéine à partir de sa séquence d'acides aminés. Ces méthodes sont critiques pour les protéines difficiles à étudier expérimentalement, comme les protéines membranaires, cibles de nombreux médicaments. Un exemple bien connu en est AlphaFold, un algorithme basé sur l'apprentissage en profondeur réputé pour ses prédictions précises.
ESMFold est une autre méthode très précise basée sur l'apprentissage en profondeur développée pour prédire la structure des protéines à partir de sa séquence d'acides aminés. ESMFold utilise un grand modèle de langage protéique (pLM) comme colonne vertébrale et fonctionne de bout en bout. Contrairement à AlphaFold2, il n'a pas besoin d'une recherche ou Alignement de séquences multiples (MSA), ni ne s'appuie sur des bases de données externes pour générer des prédictions. Au lieu de cela, l'équipe de développement a formé le modèle sur des millions de séquences de protéines d'UniRef. Au cours de la formation, le modèle a développé des schémas d'attention qui représentent avec élégance les interactions évolutives entre les acides aminés de la séquence. Cette utilisation d'un pLM au lieu d'un MSA permet des temps de prédiction jusqu'à 60 fois plus rapides que les autres modèles de pointe.
Dans cet article, nous utilisons le modèle ESMFold pré-formé de Hugging Face avec Amazon Sage Maker prédire la structure de la chaîne lourde de trastuzumabun anticorps monoclonal d'abord développé par Genentech pour le traitement de Cancer du sein HER2-positif. Prédire rapidement la structure de cette protéine pourrait être utile si les chercheurs voulaient tester l'effet des modifications de séquence. Cela pourrait potentiellement améliorer la survie des patients ou réduire les effets secondaires.
Cet article fournit un exemple de bloc-notes Jupyter et de scripts associés dans ce qui suit GitHub référentiel.
Pré-requis
Nous vous recommandons d'exécuter cet exemple dans un Amazon SageMakerStudio cahier exécutant l'image optimisée pour le processeur PyTorch 1.13 Python 3.9 sur un type d'instance ml.r5.xlarge.
Visualisez la structure expérimentale du trastuzumab
Pour commencer, nous utilisons le biopython bibliothèque et un script d'aide pour télécharger la structure du trastuzumab à partir du Banque de données sur les protéines du RCSB:
Ensuite, nous utilisons le py3Dmol bibliothèque pour visualiser la structure sous forme de visualisation 3D interactive :
La figure suivante représente la structure protéique 3D 1N8Z de la Protein Data Bank (PDB). Dans cette image, la chaîne légère du trastuzumab est affichée en orange, la chaîne lourde est bleue (avec la région variable en bleu clair) et l'antigène HER2 est vert.
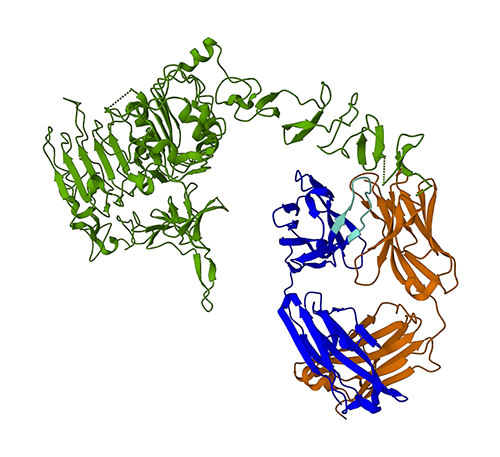
Nous allons d'abord utiliser ESMFold pour prédire la structure de la chaîne lourde (chaîne B) à partir de sa séquence d'acides aminés. Ensuite, nous comparerons la prédiction à la structure déterminée expérimentalement illustrée ci-dessus.
Prédire la structure de la chaîne lourde du trastuzumab à partir de sa séquence à l'aide d'ESMFold
Utilisons le modèle ESMFold pour prédire la structure de la chaîne lourde et la comparer au résultat expérimental. Pour commencer, nous allons utiliser un environnement de bloc-notes prédéfini dans Studio, fourni avec plusieurs bibliothèques importantes, telles que PyTorch, Pre installé. Bien que nous puissions utiliser un type d'instance accéléré pour améliorer les performances de notre analyse de bloc-notes, nous utiliserons à la place une instance non accélérée et exécuterons la prédiction ESMFold sur un processeur.
Tout d'abord, nous chargeons le modèle et le tokenizer ESMFold pré-formés à partir de Visage câlin:
Ensuite, nous copions le modèle sur notre appareil (CPU dans ce cas) et définissons certains paramètres du modèle :
Pour préparer la séquence protéique pour l'analyse, nous devons la tokeniser. Cela traduit les symboles d'acides aminés (EVQLV…) dans un format numérique que le modèle ESMFold peut comprendre (6,19,5,10,19,…) :
Ensuite, nous copions l'entrée tokenisée dans le mode, faisons une prédiction et enregistrons le résultat dans un fichier :
Cela prend environ 3 minutes sur un type d'instance non accéléré, comme un r5.
Nous pouvons vérifier l'exactitude de la prédiction ESMFold en la comparant à la structure expérimentale. Nous le faisons en utilisant le Alignement US outil développé par le Zhang Lab de l'Université du Michigan :
| PDBchain1 | PDBchain2 | Score TM |
| data/prediction.pdb:A | data/experimental.pdb:B | 0.802 |
La score de modélisation de modèle (TM-score) est une métrique pour évaluer la similarité des structures protéiques. Un score de 1.0 indique une correspondance parfaite. Les scores supérieurs à 0.7 indiquent que les protéines partagent la même structure de squelette. Les scores supérieurs à 0.9 indiquent que les protéines sont fonctionnellement interchangeable pour une utilisation en aval. Dans notre cas d'obtention de TM-Score 0.802, la prédiction ESMFold serait probablement appropriée pour des applications telles que la notation de structure ou les expériences de liaison de ligand, mais peut ne pas convenir à des cas d'utilisation tels que remplacement moléculaire qui nécessitent une très grande précision.
Nous pouvons valider ce résultat en visualisant les structures alignées. Les deux structures présentent un degré de chevauchement élevé, mais pas parfait. La prédiction de la structure des protéines est un domaine en pleine évolution et de nombreuses équipes de recherche développent des algorithmes toujours plus précis !
Déployer ESMFold en tant que point de terminaison d'inférence SageMaker
L'exécution de l'inférence de modèle dans un bloc-notes convient à l'expérimentation, mais que se passe-t-il si vous devez intégrer votre modèle à une application ? Ou un pipeline MLOps ? Dans ce cas, une meilleure option consiste à déployer votre modèle en tant que point de terminaison d'inférence. Dans l'exemple suivant, nous allons déployer ESMFold en tant que point de terminaison d'inférence en temps réel SageMaker sur une instance accélérée. Les points de terminaison en temps réel SageMaker offrent un moyen évolutif, rentable et sécurisé de déployer et d'héberger des modèles d'apprentissage automatique (ML). Avec la mise à l'échelle automatique, vous pouvez ajuster le nombre d'instances exécutant le point de terminaison pour répondre aux exigences de votre application, optimiser les coûts et garantir une haute disponibilité.
Le pré-construit Conteneur SageMaker pour Hugging Face facilite le déploiement de modèles d'apprentissage en profondeur pour les tâches courantes. Cependant, pour de nouveaux cas d'utilisation comme la prédiction de la structure des protéines, nous devons définir un inference.py script pour charger le modèle, exécuter la prédiction et formater la sortie. Ce script inclut une grande partie du même code que nous avons utilisé dans notre cahier. Nous créons également un requirements.txt fichier pour définir certaines dépendances Python à utiliser par notre point de terminaison. Vous pouvez voir les fichiers que nous avons créés dans le GitHub référentiel.
Dans la figure suivante, les structures expérimentales (bleues) et prédites (rouges) de la chaîne lourde du trastuzumab sont très similaires, mais pas identiques.

Après avoir créé les fichiers nécessaires dans le code répertoire, nous déployons notre modèle à l'aide du SageMaker HuggingFaceModel classe. Cela utilise un conteneur pré-construit pour simplifier le processus de déploiement des modèles Hugging Face sur SageMaker. Notez que la création du point de terminaison peut prendre 10 minutes ou plus, selon la disponibilité de ml.g4dn types d'instances dans notre région.
Une fois le déploiement du point de terminaison terminé, nous pouvons resoumettre la séquence protéique et afficher les premières lignes de la prédiction :
Étant donné que nous avons déployé notre point de terminaison sur une instance accélérée, la prédiction ne devrait prendre que quelques secondes. Chaque ligne du résultat correspond à un seul atome et comprend l'identité de l'acide aminé, trois coordonnées spatiales et un score pLDDT représentant la confiance de prédiction à cet emplacement.
| PDB_GROUP | ID | ATOM_LABEL | RES_ID | CHAIN_ID | SEQ_ID | CARTN_X | CARTN_Y | CARTN_Z | OCCUPATION | PLDDT | ATOM_ID |
| ATOM | 1 | N | GLU | A | 1 | 14.578 | - 19.953 | 1.47 | 1 | 0.83 | N |
| ATOM | 2 | CA | GLU | A | 1 | 13.166 | - 19.595 | 1.577 | 1 | 0.84 | C |
| ATOM | 3 | CA | GLU | A | 1 | 12.737 | - 18.693 | 0.423 | 1 | 0.86 | C |
| ATOM | 4 | CB | GLU | A | 1 | 12.886 | - 18.906 | 2.915 | 1 | 0.8 | C |
| ATOM | 5 | O | GLU | A | 1 | 13.417 | - 17.715 | 0.106 | 1 | 0.83 | O |
| ATOM | 6 | cg | GLU | A | 1 | 11.407 | - 18.694 | 3.2 | 1 | 0.71 | C |
| ATOM | 7 | cd | GLU | A | 1 | 11.141 | - 18.042 | 4.548 | 1 | 0.68 | C |
| ATOM | 8 | OE1 | GLU | A | 1 | 12.108 | - 17.805 | 5.307 | 1 | 0.68 | O |
| ATOM | 9 | OE2 | GLU | A | 1 | 9.958 | - 17.767 | 4.847 | 1 | 0.61 | O |
| ATOM | 10 | N | VAL | A | 2 | 11.678 | - 19.063 | - 0.258 | 1 | 0.87 | N |
| ATOM | 11 | CA | VAL | A | 2 | 11.207 | - 18.309 | - 1.415 | 1 | 0.87 | C |
En utilisant la même méthode que précédemment, nous constatons que les prédictions du bloc-notes et du point de terminaison sont identiques.
| PDBchain1 | PDBchain2 | Score TM |
| data/endpoint_prediction.pdb:A | data/prediction.pdb:A | 1.0 |
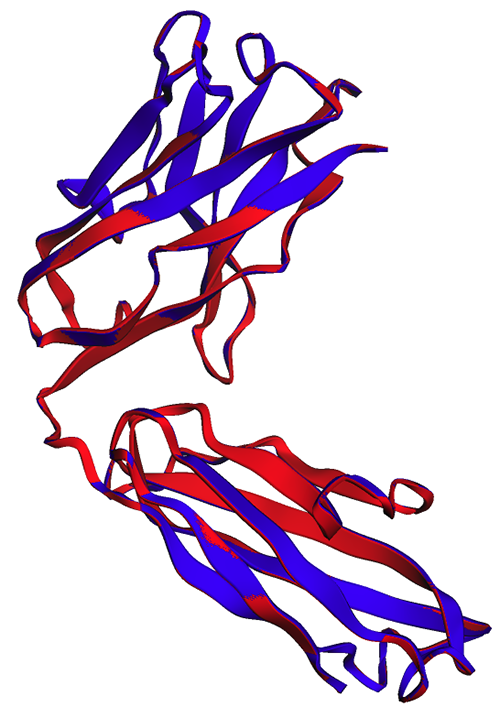
Comme observé dans la figure suivante, les prédictions ESMFold générées dans le bloc-notes (rouge) et par le point de terminaison (bleu) montrent un alignement parfait.
Nettoyer
Pour éviter des frais supplémentaires, nous supprimons notre point de terminaison d'inférence et nos données de test :
Résumé
La prédiction computationnelle de la structure des protéines est un outil essentiel pour comprendre la fonction des protéines. Outre la recherche fondamentale, des algorithmes comme AlphaFold et ESMFold ont de nombreuses applications en médecine et en biotechnologie. Les informations structurelles générées par ces modèles nous aident à mieux comprendre comment les biomolécules interagissent. Cela peut ensuite conduire à de meilleurs outils de diagnostic et à de meilleurs traitements pour les patients.
Dans cet article, nous montrons comment déployer le modèle de langage protéique ESMFold de Hugging Face Hub en tant que point de terminaison d'inférence évolutif à l'aide de SageMaker. Pour plus d'informations sur le déploiement des modèles Hugging Face sur SageMaker, reportez-vous à Utiliser l'étreinte du visage avec Amazon SageMaker. Vous pouvez également trouver plus d'exemples de science des protéines dans le Analyse de protéines impressionnante sur AWS Dépôt GitHub. S'il vous plaît laissez-nous un commentaire s'il y a d'autres exemples que vous aimeriez voir!
À propos des auteurs
 Brian Loyal est architecte principal de solutions IA/ML au sein de l'équipe mondiale des soins de santé et des sciences de la vie chez Amazon Web Services. Il a plus de 17 ans d'expérience en biotechnologie et en apprentissage automatique, et se passionne pour aider les clients à résoudre les défis génomiques et protéomiques. Dans ses temps libres, il aime cuisiner et manger avec ses amis et sa famille.
Brian Loyal est architecte principal de solutions IA/ML au sein de l'équipe mondiale des soins de santé et des sciences de la vie chez Amazon Web Services. Il a plus de 17 ans d'expérience en biotechnologie et en apprentissage automatique, et se passionne pour aider les clients à résoudre les défis génomiques et protéomiques. Dans ses temps libres, il aime cuisiner et manger avec ses amis et sa famille.
 Shamika Ariyawansa est architecte de solutions spécialisées en IA/ML au sein de l'équipe mondiale des soins de santé et des sciences de la vie chez Amazon Web Services. Il travaille avec passion avec les clients pour accélérer leur adoption de l'IA et du ML en leur fournissant des conseils techniques et en les aidant à innover et à créer des solutions cloud sécurisées sur AWS. En dehors du travail, il aime le ski et le tout-terrain.
Shamika Ariyawansa est architecte de solutions spécialisées en IA/ML au sein de l'équipe mondiale des soins de santé et des sciences de la vie chez Amazon Web Services. Il travaille avec passion avec les clients pour accélérer leur adoption de l'IA et du ML en leur fournissant des conseils techniques et en les aidant à innover et à créer des solutions cloud sécurisées sur AWS. En dehors du travail, il aime le ski et le tout-terrain.
 Yanjun Qi est Senior Applied Science Manager au AWS Machine Learning Solution Lab. Elle innove et applique l'apprentissage automatique pour aider les clients d'AWS à accélérer leur adoption de l'IA et du cloud.
Yanjun Qi est Senior Applied Science Manager au AWS Machine Learning Solution Lab. Elle innove et applique l'apprentissage automatique pour aider les clients d'AWS à accélérer leur adoption de l'IA et du cloud.
- Contenu propulsé par le référencement et distribution de relations publiques. Soyez amplifié aujourd'hui.
- PlatoAiStream. Intelligence des données Web3. Connaissance Amplifiée. Accéder ici.
- Frapper l'avenir avec Adryenn Ashley. Accéder ici.
- Achetez et vendez des actions de sociétés PRE-IPO avec PREIPO®. Accéder ici.
- La source: https://aws.amazon.com/blogs/machine-learning/accelerate-protein-structure-prediction-with-the-esmfold-language-model-on-amazon-sagemaker/
- :possède
- :est
- :ne pas
- $UP
- 1
- 10
- 100
- 11
- 12
- 13
- 3d
- 500
- 7
- 8
- 9
- a
- A Propos
- au dessus de
- AC
- accélérer
- accéléré
- précision
- Avec cette connaissance vient le pouvoir de prendre
- avec précision
- la réalisation de
- activité
- ajout
- Adoption
- AI
- AI / ML
- algorithme
- algorithmes
- aligné
- alignement
- aussi
- Bien que
- Amazon
- Amazon Sage Maker
- Amazon Web Services
- an
- selon une analyse de l’Université de Princeton
- ainsi que
- Une autre
- tous
- Application
- applications
- appliqué
- approprié
- SONT
- AS
- Évaluation
- At
- atome
- précaution
- Automatique
- disponibilité
- éviter
- AWS
- Apprentissage automatique AWS
- Colonne vertébrale
- Banque
- Essentiel
- BE
- before
- commencer
- Améliorée
- jusqu'à XNUMX fois
- propriétés de liant
- biotechnologie
- Bleu
- construire
- mais
- by
- CAN
- Cancer
- dessin animé
- maisons
- cas
- célèbre
- chaîne
- globaux
- des charges
- vérifier
- classe
- le cloud
- adoption du cloud
- code
- Couleur
- vient
- commentaire
- Commun
- comparer
- comparant
- complet
- confiance
- Contenant
- contraste
- correspond
- rentable
- Costs
- pourriez
- engendrent
- créée
- critique
- Clients
- données
- bases de données
- datetime
- profond
- l'apprentissage en profondeur
- DeepMind
- Degré
- demandes
- Selon
- déployer
- déployé
- déployer
- déploiement
- Déterminer
- déterminé
- développé
- développement
- Développement
- dispositif
- difficile
- Commande
- do
- Ne fait pas
- download
- motivation
- Médicaments
- pendant
- chacun
- Easy
- effet
- les effets
- permet
- fin
- Endpoint
- assurer
- Environment
- exemple
- exemples
- cher
- d'experience
- expériences
- externe
- extrêmement
- Visage
- famille
- plus rapide
- few
- moins
- champ
- Figure
- Déposez votre dernière attestation
- Fichiers
- Trouvez
- fin
- Prénom
- Abonnement
- Pour
- le format
- amis
- De
- fonction
- plus
- générer
- généré
- GitHub
- Global
- Vert
- l'orientation
- Vous avez
- he
- la médecine
- lourd
- vous aider
- aider
- Haute
- sa
- hôte
- Comment
- How To
- Cependant
- HTML
- HTTPS
- Moyeu
- Étreindre
- identique
- Identite
- if
- image
- importer
- important
- améliorer
- amélioré
- in
- inclut
- indiquer
- indique
- d'information
- innovons
- contribution
- perspicacité
- idées.
- instance
- plutôt ;
- intégrer
- interagir
- interactions
- Interactif
- interagit
- développement
- IT
- SES
- laboratoire
- langue
- gros
- conduire
- apprentissage
- Laisser
- bibliothèques
- Bibliothèque
- VIE
- Life Sciences
- lumière
- comme
- Probable
- charge
- emplacement
- rechercher
- click
- machine learning
- a prendre une
- FAIT DU
- manager
- de nombreuses
- Match
- Mai..
- médecine
- Découvrez
- méthode
- méthodes
- métrique
- Michigan
- des millions
- Minutes
- ML
- MLOps
- Mode
- modèle
- numériques jumeaux (digital twin models)
- Modifications
- moléculaire
- PLUS
- beaucoup
- prénom
- nécessaire
- Besoin
- NIH
- cahier
- roman
- nombre
- objets
- obsolète
- of
- on
- ONE
- uniquement
- exploite
- l'optimisation
- Option
- or
- Orange
- OS
- Autre
- nos
- sortie
- au contrôle
- paramètres
- passionné
- patientforward
- patients
- motifs
- parfaite
- performant
- pipeline
- Platon
- Intelligence des données Platon
- PlatonDonnées
- veuillez cliquer
- Post
- l'éventualité
- prévoir
- prédit
- prévoir
- prédiction
- Prédictions
- Préparer
- précédemment
- processus
- les process
- Protéines
- Protéines
- fournir
- fournit
- aportando
- Python
- pytorch
- Qi
- vite.
- rapidement
- domaine en évolution rapide
- en temps réel
- recommander
- Rouge
- région
- en relation
- compter
- représentent
- représentation
- représente
- exigent
- un article
- chercheurs
- résultat
- RANGÉE
- Courir
- pour le running
- sagemaker
- Inférence SageMaker
- même
- Épargnez
- évolutive
- mise à l'échelle
- Sciences
- STARFLEET SCIENCES
- But
- marquant
- scripts
- secondes
- sécurisé
- sur le lien
- supérieur
- Séquence
- Services
- set
- plusieurs
- Partager
- elle
- devrait
- montrer
- montré
- côté
- similaires
- simplifier
- unique
- sur mesure
- Solutions
- RÉSOUDRE
- quelques
- Spatial
- spécialiste
- Spectroscopie
- vitesse
- Commencer
- state-of-the-art
- étapes
- de construction
- structure
- studio
- Étude
- tel
- convient
- Support
- survie
- Prenez
- prend
- objectifs
- tâches
- équipe
- équipes
- Technique
- tester
- que
- qui
- La
- leur
- Les
- puis
- Là.
- Ces
- this
- trois
- tridimensionnel
- fiable
- long
- fois
- à
- tokenize
- tokenisé
- outil
- les outils
- torche
- qualifié
- Formation
- transformateurs
- transport
- traitement
- deux
- type
- types
- comprendre
- compréhension
- université
- contrairement à
- us
- utilisé
- d'utiliser
- en utilisant
- VALIDER
- très
- Voir
- visualisation
- W
- voulu
- Façon..
- we
- web
- services Web
- bien connu
- Quoi
- Wikipédia
- sera
- comprenant
- Outils de gestion
- activités principales
- vos contrats
- pourra
- radiographie
- Vous n'avez
- Votre
- zéphyrnet