Progettare una proteina è un po' come realizzare un armadietto. Il primo passo è costruire la spina dorsale che tiene insieme la proteina. Ma poi arriva la parte difficile: capire dove installare i cardini sull'impalcatura, ovvero trovare i migliori "punti caldi" per posizionare porte, scaffali e altri accessori che alla fine rendono l'armadio completamente funzionale.
In un certo senso, le proteine hanno anche punti caldi incorporati nelle loro strutture. Fedeli al loro nome, "siti funzionali", questi intriganti angoli e fessure formano intricati bacini per altre proteine o farmaci a cui aggrapparsi. I siti sono fondamentali per eseguire la maggior parte dei nostri processi biologici di base. Sono anche un'enorme miniera d'oro per la progettazione di nuovi trattamenti e farmaci.
Il problema? I siti funzionali sono difficili da mappare. Gli scienziati tradizionalmente dovevano mutare le aree sospette su una proteina una per una, passando da un aminoacido a un altro, per individuare punti di legame precisi. Come un detective che controlla centinaia di sospetti, di cui potrebbero essercene molti, è estremamente noioso.
A nuovo studio in Scienze ha rovesciato l'intero libro di gioco. Guidato dal Dr. David Baker dell'Università di Washington, un team ha sfruttato l'"immaginazione" di un'IA per inventare da zero una miriade di siti funzionali. È la migliore "creatività" di una mente macchina: un algoritmo di apprendimento profondo che prevede l'area generale del sito funzionale di una proteina, ma poi scolpisce ulteriormente la struttura.
Per verificare la realtà, il team ha utilizzato il nuovo software per generare farmaci che combattono il cancro e progettare vaccini contro virus comuni, anche se a volte mortali. In un caso, la mente digitale ha escogitato una soluzione che, se testata in cellule isolate, era perfetta per un anticorpo esistente contro un virus comune. In altre parole, l'algoritmo ha "immaginato" un hotspot da una proteina virale, rendendolo vulnerabile come obiettivo per progettare nuovi trattamenti.
L'algoritmo è la prima incursione del deep learning nella costruzione di proteine attorno alle loro funzioni, aprendo una porta a trattamenti prima inimmaginabili. Ma il software non si limita agli hotspot proteici naturali. "Le proteine che troviamo in natura sono molecole straordinarie, ma le proteine progettate possono fare molto di più", ha affermato Baker in un comunicato stampa. L'algoritmo sta "facendo cose di cui nessuno di noi pensava sarebbe stato capace".
L'hotspot proteico
Il team di Baker non è estraneo alla previsione delle proteine con menti artificiali. Alcuni anni fa, hanno scosso il campo della biologia strutturale rilasciando Rosetta, un software in grado di prevedere la struttura 3D di una proteina basata solo sulla sua sequenza di amminoacidi. Hanno ulteriormente mappato i complessi proteici e progettato "cacciaviti" proteici da zero per separare le interazioni proteiche indesiderabili. Alla fine dell'anno scorso, hanno rilasciato a rete di deep learning soprannominato trRosetta, un "architetto" di intelligenza artificiale che generalizza il modo in cui le stringhe di amminoacidi si dispongono in strutture complesse su scala nanometrica.
Torniamo indietro.
È facile immaginare le proteine come l'ala di pollo carnosa e nerboruta che sto addentando mentre scrivo questa frase. Ma a livello molecolare, sono molto più eleganti. Immagina più blocchi Lego, amminoacidi, tenuti insieme da una corda. Ora giralo, attorcigliando la catena finché alcuni blocchi non si agganciano l'uno all'altro. Questo forma una struttura delicata che spesso ricorda un'elica o lenzuola sgualcite. In alcune proteine, questi elementi costitutivi si assemblano ulteriormente in complessi, ad esempio creando un canale che attraversa la membrana protettiva di una cellula come un'autostrada interstatale sorvegliata.
Le proteine alimentano ogni singolo processo biologico, spesso attraverso una cascata di interazioni con altre proteine o farmaci, che, a seconda del partner, possono innescare conseguenze completamente diverse: una cellula dovrebbe vivere o morire? Attaccare un potenziale invasore o ritirarsi? In altre parole, le proteine sono i mattoni della vita e l'analisi della loro struttura è il modo in cui possiamo hackerare la vita.
Ecco il fatto: non tutte le parti di una proteina sono uguali. Se una proteina è un corpo umano, i siti funzionali sono le sue "mani", dove si aggrappa a un'altra proteina o farmaco, provoca reazioni enzimatiche o combatte gli agenti patogeni invasori. Incorporati direttamente nella struttura della proteina, questi siti sono difficili da definire e ancora più difficili da ricreare.
Il nuovo studio ha affrontato il problema con una versione di Rosetta: con alcune conoscenze precedenti, è possibile per un computer immaginare una catena di amminoacidi che si ripiegano naturalmente in un sito funzionale?
Il sognatore e il realista
Il problema può sembrare esotico, ma c'è un esempio precedente, in un campo diverso. Utilizzando una rete neurale, OpenAI ha creato un'ampia gamma di immagini da sole didascalie di testo. Uno spin-off del generatore di testo AI di Rockstar GPT-3, l'algoritmo DALL·E ha generato immagini fantastiche ma dall'aspetto realistico basate su semplici istruzioni di testo rilevando i modelli dal suo addestramento. "Prende i recessi più profondi e oscuri della tua immaginazione e li trasforma in qualcosa di stranamente pertinente", disse Dr. Hany Farid alla UC Berkeley dopo il rilascio iniziale dello strumento.
La costruzione di un sito funzionale proteico è simile. Qui, gli amminoacidi sono le lettere e il sito funzionale della proteina è l'immagine. "L'idea è la stessa: le reti neurali possono essere addestrate per vedere i modelli nei dati. Una volta addestrato, puoi dargli un suggerimento e vedere se può generare una soluzione elegante", ha affermato il dottor Joseph Watson, uno degli autori principali del nuovo lavoro. Tranne che invece di scrivere un romanzo, l'algoritmo potrebbe aiutare a riscrivere la vita.
Il team ha iniziato con una creazione precedente, trRosetta. È una rete neurale originariamente progettata per inventare nuove proteine basate su sequenze di amminoacidi pur essendo in grado di predire la loro struttura, alcune così estranee da quelle naturali che il team ha soprannominato "allucinazione" il funzionamento interno del deep learning. L'algoritmo sembrava perfetto: poteva prevedere sia la sequenza di amminoacidi di una proteina che la sua struttura.
Il singhiozzo? Non ha funzionato davvero. In contrasto, l'OG di previsione della struttura delle proteine, RoseTTAFold, eseguito come un campione. La potenza dell'algoritmo deriva dalla sua progettazione: modellare ogni amminoacido su scala nanometrica, fornendo coordinate a ciascun atomo. Come bloccare un sito geografico utilizzando Google Maps, questo fornisce un livello di verità di base per una struttura su cui un'IA può ulteriormente rifarsi: una sorta di "allucinazione vincolata".
Traduzione? RoseTTAFold è in grado di prevedere una struttura funzionale, specifica per il problema in questione, e di elaborare uno schizzo approssimativo come progetto finale.
Poi è arrivato un altro trucco intelligente, soprannominato "dipinto". Qui, il team ha nascosto parti della sequenza o struttura della proteina. Il software ha dovuto imparare a decifrare le informazioni da quella che è essenzialmente un'intercettazione radio rumorosa, in cui puoi ascoltare solo le prime parole ma cercare di capirne il significato riempiendo gli spazi vuoti. RoseTTAFold ha affrontato il "problema di recupero delle informazioni mancanti" con entusiasmo, completando automaticamente sia le sequenze di amminoacidi che le strutture per costruire una determinata regione funzionale con alta fedeltà.
RoseTTAFold è in grado di affrontare i problemi di costruzione di sequenze di amminoacidi e allo stesso tempo di generare una spina dorsale per il sito. È come mettere le parole su carta: chi scrive si assicura che ogni lettera sia al posto giusto, controllando nel contempo che la grammatica e il significato abbiano un senso.
Mettere in discussione la natura della realtà
Mettendo alla prova la loro nuova creazione, il team ha generato diversi progetti di farmaci e vaccini che potrebbero potenzialmente combattere virus e cancro o aiutare con problemi di salute a basso contenuto di ferro.
Per guidare l'autore Dr. Jue Wang, l'algoritmo è diventato inaspettatamente pertinente. Mentre lavorava al progetto, suo figlio di due anni è stato ricoverato in ospedale nel reparto di emergenza a causa di un'infezione polmonare da RSV (virus respiratorio sinciziale), un virus che normalmente mostra sintomi simili al raffreddore, ma può essere mortale nei giovani e nel anziano.
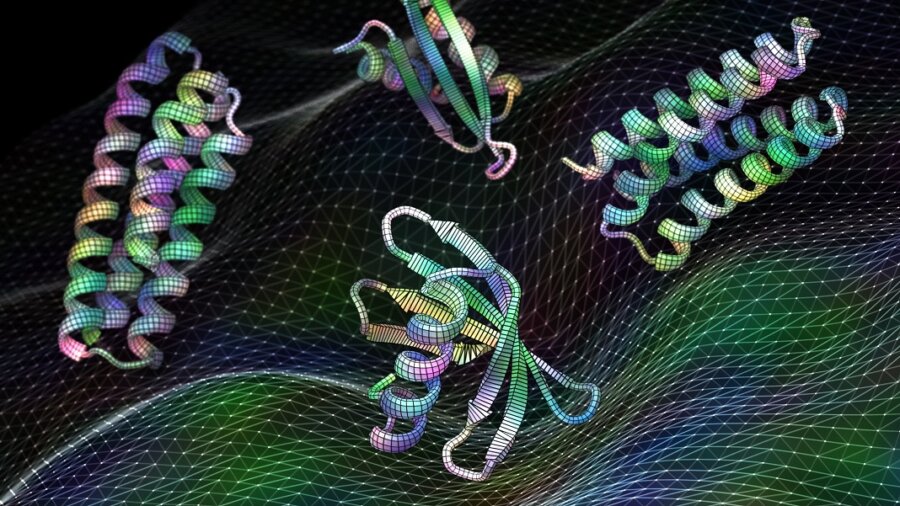
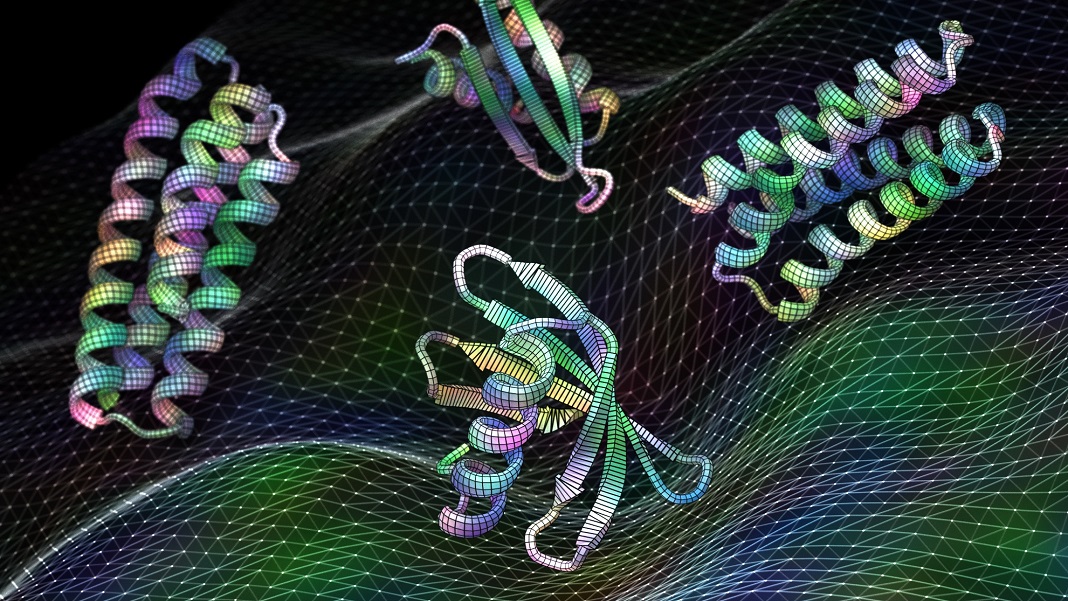
A quel tempo, Wang stava usando l'algoritmo per progettare nuovi trattamenti, che includevano potenziali siti su RSV per testare ulteriormente vaccini e farmaci contro. È una struttura relativamente ben mappata. Il software allucinava progetti che ricapitolavano due siti a cui potenzialmente legarsi i vaccini. I test che utilizzano proteine allucinate, ricostruite nei batteri, si sono rapidamente attaccate agli anticorpi esistenti, un segno che sono funzionali e che l'approccio del deep learning funziona.
L'incidente "mi ha fatto capire che anche i problemi di 'test' su cui stavamo lavorando erano in realtà abbastanza significativi", ha detto Wang.
In numerosi test aggiuntivi, il team ha progettato siti funzionali per un enzima, proteine che legano le proteine e proteine che si aggrappano agli ioni metallici, in pratica il modo in cui assorbi il ferro e altri metalli importanti.
Sebbene potente, c'è spazio per la crescita. Il metodo apre le porte alla demistificazione delle proteine naturali, ma anche alla potenziale progettazione di nuove per la biologia sintetica. "Si tratta di nuovi approcci molto potenti, ma c'è ancora molto margine di miglioramento", ha affermato Baker.
Nel complesso, è un'altra vittoria per il deep learning e un'avvincente vetrina di come l'IA e la biologia possono sinergizzare. "L'apprendimento profondo ha trasformato la previsione della struttura delle proteine negli ultimi due anni, ora siamo nel mezzo di una simile trasformazione del design delle proteine", ha affermato Baker.
Credito immagine: Ian C. Haydon/Istituto UW per la progettazione delle proteine. Il nuovo software di intelligenza artificiale addestrato sulle strutture proteiche può generare proteine funzionali, compresi questi vaccini candidati per il virus respiratorio RSV, in pochi secondi.