Techniki obrazowania medycznego, takie jak tomografia komputerowa (CT), rezonans magnetyczny (MRI), medyczne obrazowanie rentgenowskie, obrazowanie ultrasonograficzne i inne, są powszechnie stosowane przez lekarzy z różnych powodów. Niektóre przykłady obejmują wykrywanie zmian w wyglądzie narządów, tkanek i naczyń oraz wykrywanie nieprawidłowości, takich jak guzy i różne inne rodzaje patologii.
Zanim lekarze będą mogli wykorzystać dane z tych technik, dane muszą zostać przekształcone z ich natywnej surowej postaci do postaci, którą można wyświetlić jako obraz na ekranie komputera.
Ten proces jest znany jako rekonstrukcja obrazui odgrywa kluczową rolę w przepływie pracy z obrazowaniem medycznym — jest to etap tworzenia obrazów diagnostycznych, które mogą być następnie przeglądane przez lekarzy.
W tym poście omawiamy przypadek użycia rekonstrukcji MRI, ale koncepcje architektoniczne można zastosować do innych rodzajów rekonstrukcji obrazu.
Postępy w dziedzinie rekonstrukcji obrazu doprowadziły do pomyślnego zastosowania technik opartych na sztucznej inteligencji w obrazowaniu metodą rezonansu magnetycznego (MR). Techniki te mają na celu zwiększenie dokładności rekonstrukcji oraz w przypadku MR, a także skrócenie czasu wymaganego do pełnego skanu.
W ramach MR z powodzeniem zastosowano aplikacje wykorzystujące sztuczną inteligencję do pracy z niedopróbkowanymi akwizycjami, osiągnięcie prawie dziesięciokrotnego skrócenia czasu skanowania.
Czasy oczekiwania na badania takie jak rezonans magnetyczny i tomografia komputerowa gwałtownie wzrosły w ciągu ostatnich kilku lat, co prowadzi do: czas oczekiwania nawet 3 miesiące. Aby zapewnić dobrą opiekę nad pacjentem, rosnąca potrzeba szybkiej dostępności zrekonstruowanych obrazów wraz z koniecznością obniżenia kosztów operacyjnych spowodowała potrzebę rozwiązania zdolnego do skalowania zgodnie z potrzebami w zakresie przechowywania i obliczeń.
Oprócz potrzeb obliczeniowych w ostatnich latach obserwuje się stały wzrost ilości danych. Na przykład, patrząc na zbiory danych udostępnione przez Medyczne przetwarzanie obrazu i interwencja wspomagana komputerowo (MICCAI), można stwierdzić, że roczny wzrost wynosi 21% dla MRI, 24% dla CT i 31% dla funkcjonalnego MRI (fMRI). (Aby uzyskać więcej informacji, zobacz Wzrost zestawu danych w badaniach nad analizą obrazu medycznego.)
W tym poście przedstawiamy architekturę rozwiązania, która odpowiada na te wyzwania. To rozwiązanie może umożliwić ośrodkom badawczym, instytucjom medialnym i dostawcom modalności dostęp do nieograniczonych możliwości pamięci masowej, skalowalnej mocy GPU, szybkiego dostępu do danych na potrzeby szkolenia i rekonstrukcji uczenia maszynowego (ML), prostych i szybkich środowisk programistycznych ML oraz możliwości mieć lokalną pamięć podręczną, aby zapewnić szybką dostępność danych obrazu z małymi opóźnieniami.
Omówienie rozwiązania
To rozwiązanie wykorzystuje technikę rekonstrukcji MRI znaną jako Solidne sztuczne sieci neuronowe do interpolacji w przestrzeni k (RAKI). Takie podejście jest korzystne, ponieważ jest specyficzne dla skanowania i nie wymaga wcześniejszych danych do trenowania sieci neuronowej. Wadą tej techniki jest to, że wymaga dużej mocy obliczeniowej, aby była skuteczna.
Przedstawiona architektura AWS pokazuje, w jaki sposób podejście do rekonstrukcji oparte na chmurze może skutecznie wykonywać ciężkie zadania obliczeniowe, takie jak te wymagane przez sieć neuronową RAKI, skalując zgodnie z obciążeniem i przyspieszając proces rekonstrukcji. Otwiera to drzwi do technik, których nie można realistycznie wdrożyć lokalnie.
Warstwa danych
Warstwa danych została zaprojektowana zgodnie z następującymi zasadami:
- Bezproblemowa integracja z modalnościami, które przechowują dane generowane na podłączonym dysku pamięci masowej za pośrednictwem udziału sieciowego na urządzeniu NAS
- Nieograniczone i bezpieczne możliwości przechowywania danych w celu skalowania do ciągłego zapotrzebowania na przestrzeń dyskową
- Szybka dostępność pamięci masowej dla obciążeń ML, takich jak głębokie trenowanie neuronowe i rekonstrukcja obrazów neuronowych
- Możliwość archiwizacji danych historycznych przy użyciu taniego, skalowalnego podejścia
- Zezwalaj na dostępność najczęściej używanych danych zrekonstruowanych, jednocześnie zachowując mniej często używane dane zarchiwizowane przy niższych kosztach
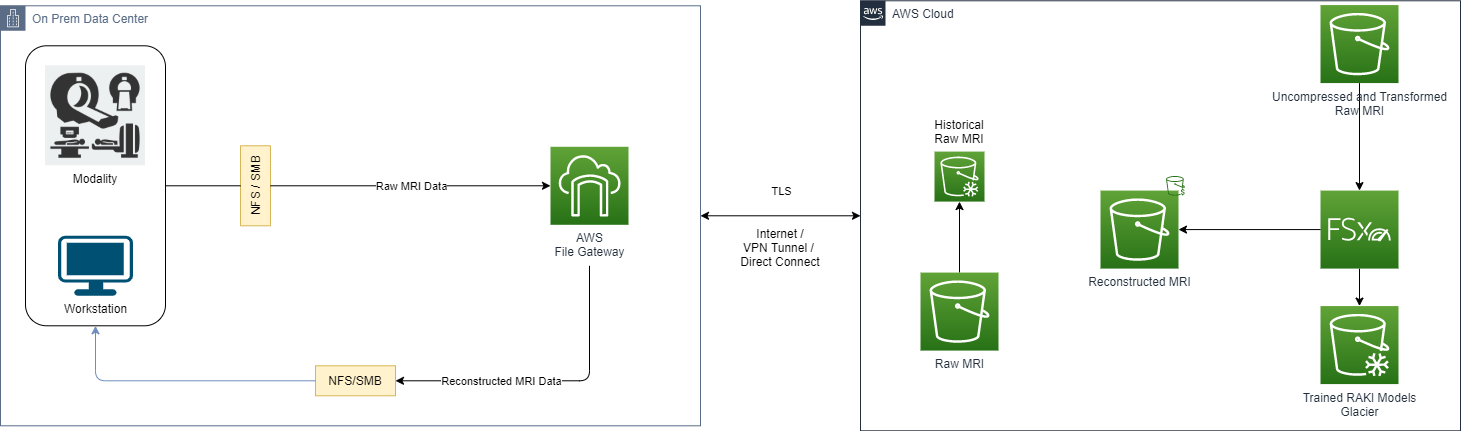
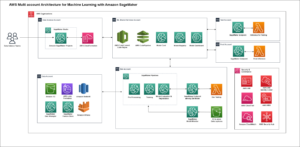
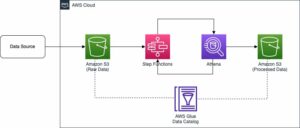
Poniższy diagram ilustruje tę architekturę.
Takie podejście wykorzystuje następujące usługi:
- Brama pamięci masowej AWS dla bezproblemowej integracji z lokalnym trybem wymiany informacji za pośrednictwem systemu udostępniania plików. Umożliwia to przejrzysty dostęp do następujących możliwości przechowywania danych w chmurze AWS przy zachowaniu sposobu wymiany danych w modalności:
- Szybkie przesyłanie do chmury wolumenów generowanych przez modalność MR.
- Dostęp z niskimi opóźnieniami do często używanych zrekonstruowanych badań MR za pośrednictwem lokalnego buforowania oferowanego przez Storage Gateway.
- Amazon Sage Maker do nieograniczonego i skalowalnego przechowywania w chmurze. Amazon S3 zapewnia również niedrogie, historyczne, surowe dane MRI do głębokiej archiwizacji dzięki Lodowiec Amazon S3, oraz inteligentna warstwa pamięci masowej dla zrekonstruowanego MRI z Inteligentne warstwowanie Amazon S3.
- Amazon FSx dla Luster do szybkiej i skalowalnej pamięci pośredniej używanej do zadań uczenia maszynowego i rekonstrukcji.
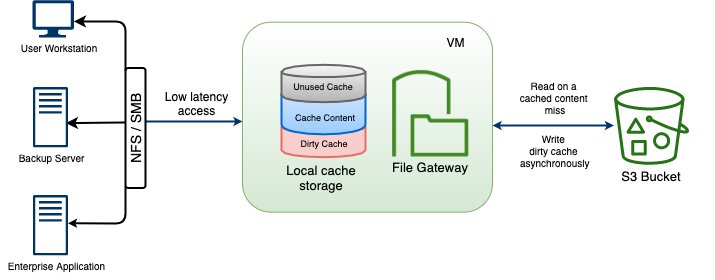
Poniższy rysunek przedstawia zwięzłą architekturę opisującą wymianę danych między środowiskami chmurowymi.
Korzystanie z usługi Storage Gateway z mechanizmem buforowania umożliwia aplikacjom lokalnym szybki dostęp do danych dostępnych w lokalnej pamięci podręcznej. Dzieje się tak, zapewniając jednocześnie dostęp do skalowalnej przestrzeni dyskowej w chmurze.
Dzięki takiemu podejściu modalności mogą generować nieprzetworzone dane z zadań akwizycji, a także zapisywać nieprzetworzone dane w udziale sieciowym obsługiwanym przez bramę pamięci masowej.
Jeśli modalność generuje wiele plików należących do tego samego skanowania, zaleca się utworzenie jednego archiwum (na przykład .tar) i wykonanie pojedynczego transferu do udziału sieciowego w celu przyspieszenia transferu danych.
Warstwa dekompresji i transformacji danych
Warstwa dekompresji danych odbiera nieprzetworzone dane, automatycznie przeprowadza dekompresję i stosuje potencjalne przekształcenia nieprzetworzonych danych przed przesłaniem wstępnie przetworzonych danych do warstwy rekonstrukcji.
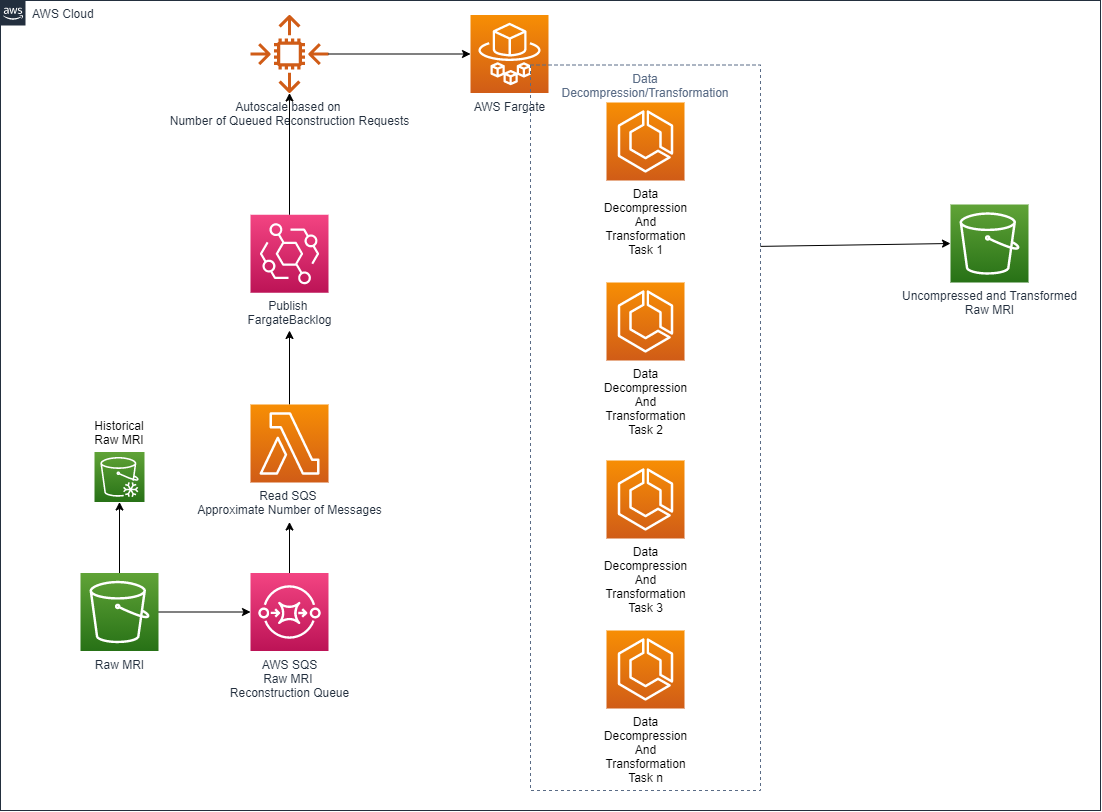
Przyjętą architekturę przedstawia poniższy rysunek.
W tej architekturze nieprzetworzone dane MRI trafiają do pojemnika nieprzetworzonego MRI S3, tym samym wywołując nowy wpis w Usługa Amazon Simple Queue (Amazon SQS).
An AWS Lambda funkcja pobiera głębokość kolejki nieprzetworzonego MRI Amazon SQS, która reprezentuje ilość akwizycji nieprzetworzonego MRI przesłanych do chmury AWS. Jest używany z AWS-Fargate aby automatycznie modulować rozmiar an Usługa Amazon Elastic Container Service (Amazon ECS).
Takie podejście do architektury umożliwia automatyczne skalowanie w górę i w dół zgodnie z liczbą nieprzetworzonych skanów umieszczonych w zasobniku wejściowym nieprzetworzonych danych.
Po zdekompresowaniu i wstępnym przetworzeniu nieprzetworzonych danych MRI są one zapisywane w innym pojemniku S3, aby można je było zrekonstruować.
Warstwa rozwoju modelu neuronowego
Warstwa rozwoju modelu neuronowego składa się z implementacji RAKI. W ten sposób powstaje model sieci neuronowej, który umożliwia szybką rekonstrukcję obrazu niedostatecznie próbkowanych surowych danych rezonansu magnetycznego.
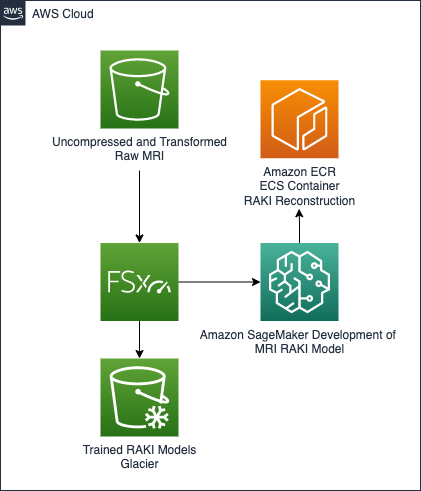
Poniższy rysunek przedstawia architekturę realizującą tworzenie modeli neuronowych i tworzenie kontenerów.
W tej architekturze Amazon Sage Maker służy do opracowania modelu neuronowego RAKI, a jednocześnie do stworzenia pojemnika, który później służy do wykonania rekonstrukcji MRI.
Następnie utworzony kontener trafia do w pełni zarządzanego Rejestr elastycznego pojemnika Amazon (Amazon ECR), dzięki czemu może następnie wydzielić zadania rekonstrukcji.
Szybkie przechowywanie danych gwarantuje przyjęcie Amazon FSx dla Luster. Zapewnia opóźnienia poniżej milisekundy, do setek GB/s przepustowości i do milionów IOPS. Takie podejście daje firmie SageMaker dostęp do ekonomicznego, wydajnego i skalowalnego rozwiązania pamięci masowej.
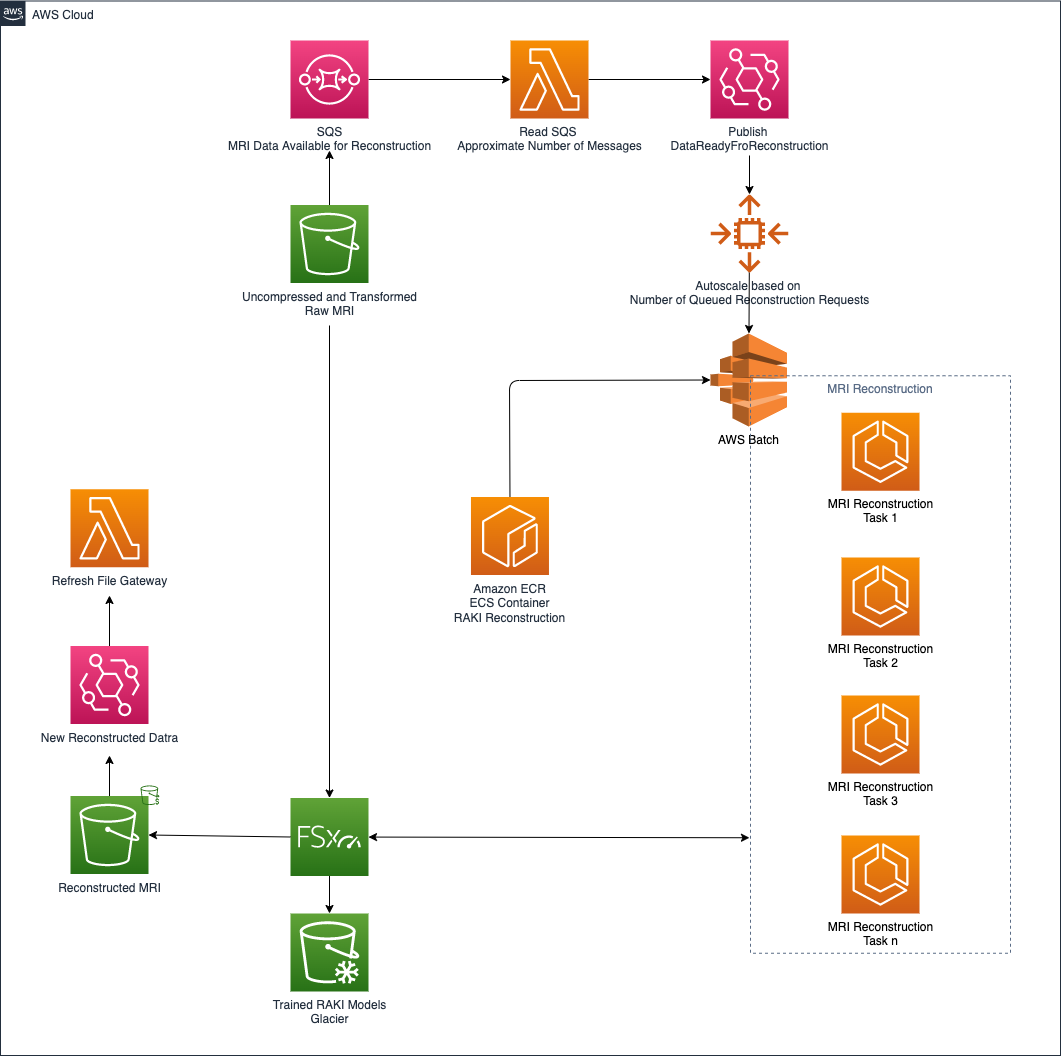
Warstwa rekonstrukcyjna MRI
Rekonstrukcja MRI oparta na sieci neuronowej RAKI jest obsługiwana przez architekturę pokazaną na poniższym schemacie.
Dzięki temu samemu wzorcowi architektonicznemu przyjętemu w warstwie dekompresji i przetwarzania wstępnego, warstwa rekonstrukcji automatycznie skaluje się w górę iw dół, analizując głębokość kolejki odpowiedzialnej za przechowywanie wszystkich żądań rekonstrukcji. W takim przypadku, aby włączyć obsługę GPU, Partia AWS służy do uruchamiania zadań rekonstrukcji MRI.
Amazon FSx for Luster służy do wymiany dużej ilości danych związanych z akwizycją MRI. Co więcej, gdy zadanie rekonstrukcji zostanie zakończone, a zrekonstruowane dane MRI są przechowywane w docelowym wiadrze S3, zastosowana architektura automatycznie żąda odświeżenia bramy pamięci masowej. Dzięki temu zrekonstruowane dane są dostępne dla placówki lokalnej.
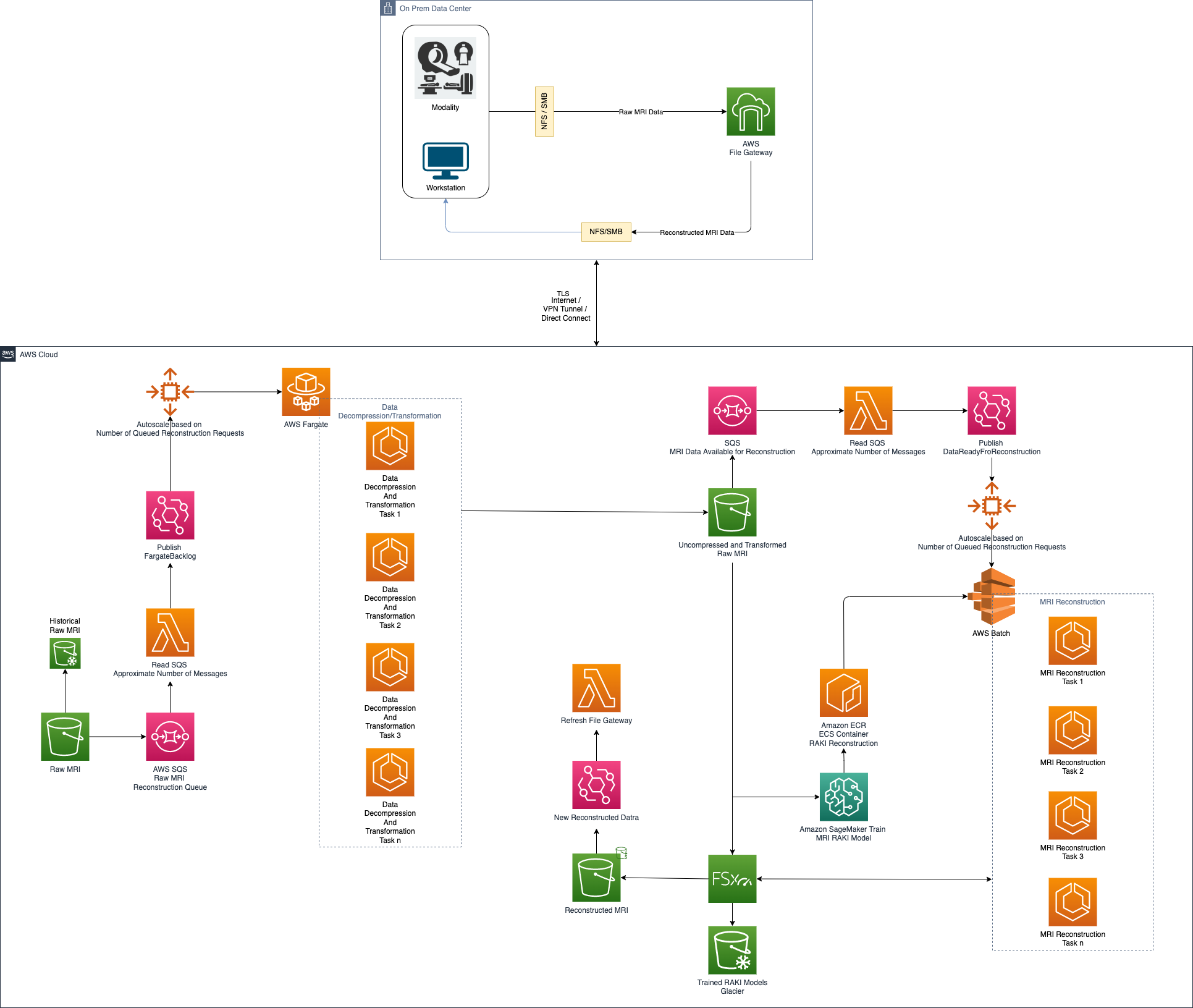
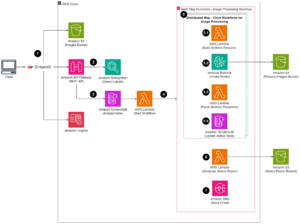
Ogólna architektura i wyniki
Ogólną architekturę przedstawiono na poniższym rysunku.
Zastosowaliśmy opisaną architekturę do zadań rekonstrukcji MRI z zbiory danych około 2.4 GB.
Szkolenie 210 zestawów danych zajęło około 221 sekund, co daje łącznie 514 GB surowych danych na jednym węźle wyposażonym w kartę Nvidia Tesla V100-SXM2-16GB.
Rekonstrukcja, po przeszkoleniu sieci RAKI, trwała średnio 40 sekund na pojedynczym węźle wyposażonym w Nvidia Tesla V100-SXM2-16GB.
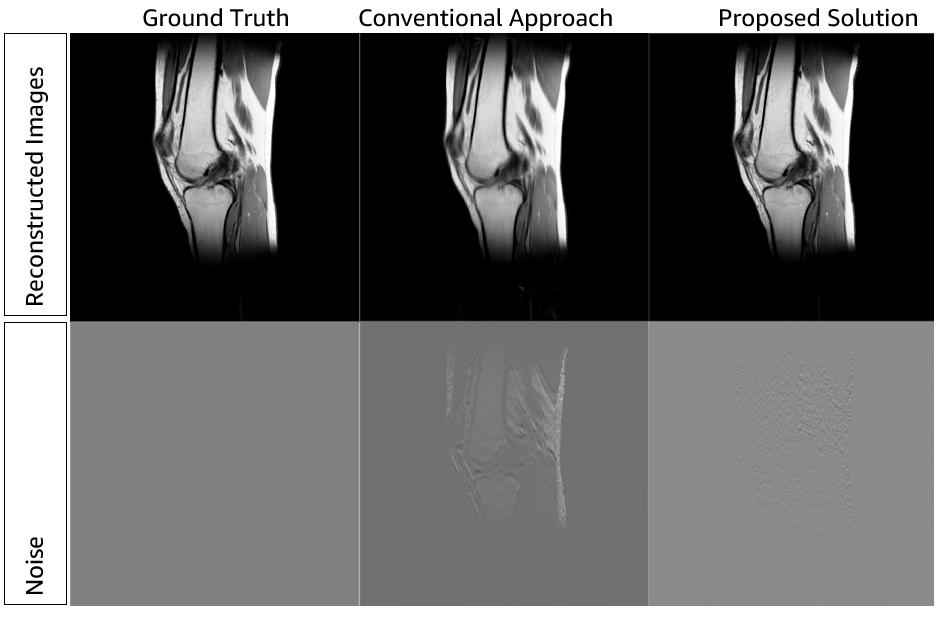
Zastosowanie poprzedniej architektury do zadania rekonstrukcji może dać wyniki przedstawione na poniższym rysunku.
Obraz pokazuje, że dobre wyniki można uzyskać za pomocą technik rekonstrukcji, takich jak RAKI. Co więcej, przyjęcie technologii chmury może sprawić, że podejścia wymagające dużej mocy obliczeniowej będą dostępne bez ograniczeń występujących w rozwiązaniach lokalnych, w których zasoby pamięci masowej i obliczeniowe są zawsze ograniczone.
wnioski
Dzięki takim narzędziom, jak Amazon SageMaker, Amazon FSx for Lustre, AWS Batch, Fargate i Lambda, możemy stworzyć zarządzane środowisko, które jest skalowalne, bezpieczne, opłacalne i zdolne do wykonywania złożonych zadań, takich jak rekonstrukcja obrazu na dużą skalę.
W tym poście zbadaliśmy możliwe rozwiązanie rekonstrukcji obrazu na podstawie surowych danych modalności przy użyciu intensywnej obliczeniowo techniki znanej jako RAKI: technika głębokiego uczenia bez bazy danych do szybkiej rekonstrukcji obrazu.
Aby dowiedzieć się więcej o tym, jak AWS przyspiesza innowacje w opiece zdrowotnej, odwiedź AWS dla zdrowia.
Referencje
O autorze
 Benedetta Carollo jest starszym architektem rozwiązań w zakresie obrazowania medycznego i opieki zdrowotnej w Amazon Web Services w Europie, na Bliskim Wschodzie i w Afryce. Jego praca koncentruje się na pomocy klientom z zakresu obrazowania medycznego i opieki zdrowotnej w rozwiązywaniu problemów biznesowych poprzez wykorzystanie technologii. Benedetto ma ponad 15-letnie doświadczenie w technologii i obrazowaniu medycznym i pracował dla firm takich jak Canon Medical Research i Vital Images. Benedetto otrzymał tytuł magistra inżyniera oprogramowania z wyróżnieniem na Uniwersytecie w Palermo – Włochy.
Benedetta Carollo jest starszym architektem rozwiązań w zakresie obrazowania medycznego i opieki zdrowotnej w Amazon Web Services w Europie, na Bliskim Wschodzie i w Afryce. Jego praca koncentruje się na pomocy klientom z zakresu obrazowania medycznego i opieki zdrowotnej w rozwiązywaniu problemów biznesowych poprzez wykorzystanie technologii. Benedetto ma ponad 15-letnie doświadczenie w technologii i obrazowaniu medycznym i pracował dla firm takich jak Canon Medical Research i Vital Images. Benedetto otrzymał tytuł magistra inżyniera oprogramowania z wyróżnieniem na Uniwersytecie w Palermo – Włochy.
- AI
- ai sztuka
- generator sztuki ai
- masz robota
- Amazona FSx
- Usługa Amazon Simple Storage (S3)
- sztuczna inteligencja
- certyfikacja sztucznej inteligencji
- sztuczna inteligencja w bankowości
- robot sztucznej inteligencji
- roboty sztucznej inteligencji
- oprogramowanie sztucznej inteligencji
- Uczenie maszynowe AWS
- Brama pamięci masowej AWS
- blockchain
- konferencja blockchain ai
- pomysłowość
- sztuczna inteligencja konwersacyjna
- konferencja kryptograficzna
- Dall's
- głęboka nauka
- Ekspert (400)
- google to
- uczenie maszynowe
- plato
- Platon Ai
- Analiza danych Platona
- Gra Platona
- PlatoDane
- platogaming
- skala ai
- składnia
- zefirnet